一種基于菌群優化算法的蛋白質結構從頭預測方法與流程
本發明涉及一種生物學信息學、智能優化、計算機應用領域,尤其涉及的是,一種基于菌群優化算法的蛋白質結構從頭預測方法。
背景技術:
蛋白質是所有生物中最重要的大分子之一,一個細胞的干重的一半以上是蛋白質。蛋白質是由單一的肽鍵連接在一起的氨基酸的順序鏈,這些連接的鏈折疊成能體現它們功能的三維結構,并調節生物體中的細胞活動,以保持生命。蛋白質分子在分子水平上具體體現了蛋白質結構和功能之間的顯著關系。蛋白質在生物學中有許多不同的功能。蛋白質的三維天然結構決定它的生物學機制,而反過來,蛋白質氨基酸單體的一維鏈又對它的天然結構進行編碼。蛋白質的三維結構在藥物設計、蛋白質工程和生物技術中具有決定性的重要,因此,蛋白質結構預測是一個重要的研究問題。
蛋白質結構實驗測定方法包括x射線晶體學、核磁共振光譜、和電子顯微鏡等,這些方法被廣泛用于蛋白質結構測定。x射線晶體學被認為是這些方法中相對可行且準確的測定方法之一。然而,x射線晶體學需要進行一個復雜的結晶過程,而對于一些不易結晶的蛋白(如膜蛋白),此方法無法用于結構測定。此外,這些實驗測定方法極其耗時,代價昂貴,而且容易發生錯誤。
anfinsen熱力學假說表明:蛋白質的空間結構由氨基酸序列唯一確定;蛋白質的空間結構是穩定的;蛋白質的天然構象處于自由能最低點。根據anfinsen原則,以計算機為工具,運用適當的算法,從氨基酸序列出發直接預測蛋白質的三維結構,成為當前生物信息學中一種主要的研究課題。在上世紀文獻中,通常將預測方法劃分為以下三類:針對高相似序列的同源建模方法;針對較低相似性序列的折疊識別方法;以及不依賴模板的從頭預測方法。從頭預測方法直接基于anfinsen假說建立蛋白質物理或知識能量模型,然后設計適當優化算法求解最小能量構象。一方面,從生物學意義上來講,有助于揭示蛋白質折疊機理,進而能夠最終闡明生物學中心法則中的第二遺傳密碼理論部分;另一方面,從現實意義來講,該方法具有普遍性,對于序列相似度<20%或寡肽(<10個殘基的小蛋白)來說,從頭預測方法是唯一的選擇。
目前主要的技術瓶頸在于兩個方面,第一方面在于采樣方法,現有技術對構象空間采樣能力不強,另一方面在于構象更新方法,現有技術對構象的更新精度仍然不足。因此,現有的構象空間搜索方法存在不足,需要改進。
技術實現要素:
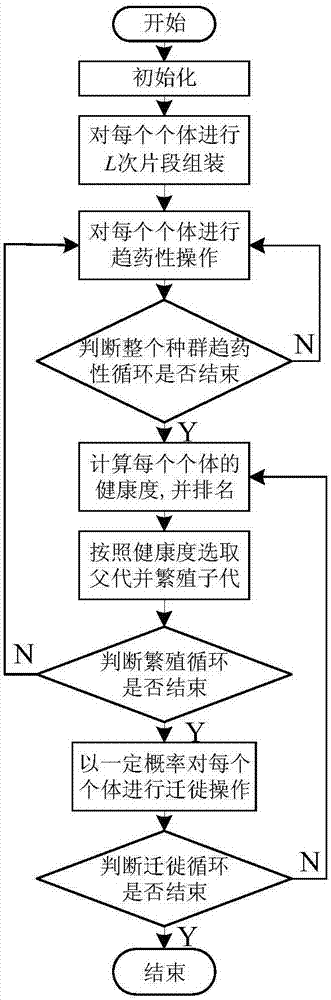
為了克服現有的蛋白質結構預測方法在構象空間搜索方面的不足,本發明提出一種基于菌群優化算法的蛋白質結構從頭預測方法。根據菌群具有趨藥性和聚集的特性,以及菌群優勝劣汰的自然規律,構象二面角在隨機方向上前進并接受能量低的構象;待構象聚集后,淘汰一半能量高的構象,能量低的構象繁殖生成子代。此外,為了盡量避免個體陷入局部極小和過早收斂,采用遷移機制對個體進行片段組裝,保證群體的多樣性,有效提高構象空間采樣能力,減少計算代價。
本發明解決其技術問題所采用的技術方案是:
一種基于菌群優化算法的蛋白質結構從頭預測方法,所述方法包括以下步驟:
1)選取rosettascore3作為蛋白質力場模型,即能量函數j(c),c表示蛋白質構象;
2)給定輸入序列信息,通過免費網絡服務器robettaserver獲取目標蛋白片段庫;
3)參數初始化:設置細菌的種群規模np,np為偶數,第i個細菌進行趨藥性行為的步長stepi,i∈{1,...,np},細菌趨藥性行為循環次數nc,趨藥性循環中最大前進次數ns,細菌進行繁殖行為次數nre,細菌進行遷徙行為的次數ned,遷徙概率ped;
4)用二面角對
5)構象初始化:根據給定輸入序列,生成np個伸展鏈構象,作為初始細菌群體p={c1,c2,...,cnp},依次對細菌群體中的構象ci進行l次隨機片段組裝,片段從目標蛋白的片段庫中隨機選擇;
6)趨藥性行為,過程如下:
6.1)計算個體ci的能量值,并記錄歷史最優能量值jlast=j(ci);
6.2)隨機產生維數為2l的方向向量βi,決定個體的前進方向;
6.3)根據
6.4)聚集行為,過程如下:
6.4.1)根據
6.4.2)根據
6.4.3)整個細菌種群對第i個個體的聚集作用即吸引力和排斥力之和
6.4.4)修正j(c),
6.5)令新構象
6.6)對下一個細菌進行趨藥性操作6.1)~6.5),直至整個種群完成一次趨藥性過程;
6.7)判斷趨藥性迭代過程是否結束,如果j<nc,返回步驟6),繼續對細菌種群進行趨藥性迭代過程,j表示當前趨藥性迭代次數;否則執行下一步;
7)繁殖循環,過程如下:
71)根據
7.2)取前np/2個個體作為父代,健康度不高的后np/2個個體死亡;生成np/2個與父代完全相同的子代;
7.3)父代和子代重新編號,若k<nre,對整個種群進行趨藥性操作,返回步驟6),否則,執行下一步;
8)為避免陷入局部最小和早熟,執行遷徙過程:對每個細菌個體,隨機產生[0,1]的隨機數rand,若rand<ped,則對該構象進行2l次隨機片段組裝,保證種群的多樣性;
9)若l<ned,對種群執行繁殖操作,返回步驟7),否則,輸出結果并退出。
本發明的技術構思為:根據菌群具有趨藥性和聚集的特性,以及菌群優勝劣汰的自然規律,對構象二面角在隨機方向上前進,并接受能量低的構象;待構象聚集后,淘汰一半能量高的構象,利用能量低的構象繁殖生成子代;此外,為了盡量避免個體陷入局部極小和過早收斂,采用遷移機制對個體進行片段組裝,保證群體的多樣性,從而有效提高構象空間采樣能力。
本發明的有益效果為:本發明在蛋白質結構預測中應用,可以有效提高構象空間的采樣能力,減少計算代價。
附圖說明
圖1是基于菌群優化算法的蛋白質結構從頭預測方法對目標蛋白質1acf預測結構的三維示意圖。
圖2是蛋白質結構的示意圖。
具體實施方式
下面結合附圖對本發明作進一步描述。
參照圖1,一種基于菌群優化算法的蛋白質結構從頭預測方法,包括以下步驟:
1)選取rosettascore3作為蛋白質力場模型,即能量函數j(c),c表示蛋白質構象;
2)給定輸入序列信息,通過免費網絡服務器robettaserver獲取目標蛋白片段庫;
3)參數初始化:細菌的種群規模np,np為偶數,第i個細菌進行趨藥性行為的步長stepi,i∈{1,...,np},細菌趨藥性行為循環次數nc,趨藥性循環中最大前進次數ns,細菌進行繁殖行為次數nre,細菌進行遷徙行為的次數ned,遷徙概率ped;
4)用二面角對
5)構象初始化:根據給定輸入序列,生成np個伸展鏈構象,作為初始細菌群體p={c1,c2,...,cnp},依次對細菌群體中的構象ci進行l次隨機片段組裝,片段從目標蛋白的片段庫中隨機選擇;
6)趨藥性行為,過程如下:
6.1)計算個體ci的能量值,并記錄歷史最優能量值jlast=j(ci);
6.2)隨機產生維數為2l的方向向量βi,決定個體的前進方向;
6.3)根據
6.4)聚集行為,過程如下:
6.4.1)根據
6.4.2)根據
6.4.3)整個細菌種群對第i個個體的聚集作用即吸引力和排斥力之和,
6.4.4)修正j(c),
6.5)令新構象
6.6)對下一個細菌進行趨藥性操作6.1)~6.5),直至整個種群完成一次趨藥性過程;
6.7)判斷趨藥性迭代過程是否結束,如果j<nc,返回步驟6),繼續對細菌種群進行趨藥性迭代過程,j表示當前趨藥性迭代次數;否則執行下一步;
7)繁殖循環,過程如下:
7.1)根據
7.2)取前np/2個個體作為父代,健康度不高的后np/2個個體死亡;生成np/2個與父代完全相同的子代;
7.3)父代和子代重新編號,若k<nre,對整個種群進行趨藥性操作,返回步驟6);否則,執行下一步;
8)為盡量避免陷入局部最小和早熟,執行遷徙過程:對每個細菌個體,隨機產生[0,1]的隨機數rand,若rand<ped,則對該構象進行2l次隨機片段組裝,保證種群的多樣性;
9)若l<ned,對種群執行繁殖操作,返回步驟7);否則,輸出結果并退出。
本實施例以目標蛋白質1acf為實施例,一種基于菌群優化算法的蛋白質結構從頭預測方法,所述方法包括以下步驟:
1)選取rosettascore3作為蛋白質力場模型,即能量函數j(c),c表示蛋白質構象;
2)給定輸入序列信息,通過免費網絡服務器robettaserver獲取目標蛋白片段庫;
3)參數初始化:細菌的種群規模np=50,第i個細菌進行趨藥性行為的步長stepi=30,i∈{1,...,np},細菌趨藥性行為循環次數nc=50,趨藥性循環中最大前進次數ns=50,細菌進行繁殖行為次數nre=5,細菌進行遷徙行為的次數ned=2,遷徙概率ped=0.2;
4)用二面角對
5)構象初始化:根據給定輸入序列,生成np個伸展鏈構象,作為初始細菌群體p={c1,c2,...,cnp},依次對細菌群體中的構象ci進行l次隨機片段組裝;
6)趨藥性行為,過程如下:
6.1)計算個體ci的能量值,并記錄歷史最優能量值jlast=j(ci);
6.2)隨機產生維數為2l的方向向量βi,決定個體的前進方向;
6.3)根據
6.4)聚集行為,過程如下:
6.4.1)根據
6.4.2)根據
6.4.3)整個細菌種群對第i個個體的聚集作用即吸引力和排斥力之和,
6.4.4)修正j(c),
6.5)令新構象
6.6)對下一個細菌進行趨藥性操作6.1)~6.5),直至整個種群完成一次趨藥性過程;
6.7)判斷趨藥性迭代過程是否結束,如果j<nc,返回步驟6),繼續對細菌種群進行趨藥性迭代過程,j表示當前趨藥性迭代次數;否則執行下一步;
7)繁殖循環,過程如下:
7.1)根據
7.2)取前np/2個個體作為父代,健康度不高的后np/2個個體死亡;生成np/2個與父代完全相同的子代;
7.3)父代和子代重新編號,若k<nre,對整個種群進行趨藥性操作,返回步驟6);否則,執行下一步;
8)為盡量避免陷入局部最小和早熟,執行遷徙過程:對每個細菌個體,隨機產生[0,1]的隨機數rand,若rand<ped,則對該構象進行2l次隨機片段組裝,保證種群的多樣性;
9)若l<ned,對種群執行繁殖操作,返回步驟7);否則,輸出結果并退出。
以上闡述的是本發明給出的一個實施例表現出來的優良效果,顯然本發明不僅適合上述實施例,在不偏離本發明基本精神及不超出本發明實質內容所涉及內容的前提下可對其做種種變化加以實施。
- 還沒有人留言評論。精彩留言會獲得點贊!