一種基因變異致病等級確定方法及裝置與流程
本發明涉及數據分析及生物信息技術領域,具體而言,涉及一種基因變異致病等級確定方法及裝置。
背景技術:
單基因遺傳病是指受一對等位基因控制的遺傳病,約有6600多種,并且每年在以10-50種的速度遞增,較為常見的有紅綠色盲、血友病、白化病等,目前,單基因遺傳病對人類的健康已經構成了很大的威脅,因此,在某些情況下需要對單基因遺傳病進行檢測,而在檢測單基因遺傳病時,需要使用單基因遺傳病數據庫。
現有的單基因遺傳病數據庫有人類孟德爾遺傳(0nlinemendelianinheritanceinman,omim)、人類基因突變數據庫(thehumangenemutationdatabase,hgmd)等,在單基因遺傳病數據庫中存儲有對單基因遺傳病進行研究的證據,以及基因變異信息對單基因遺傳病的致病性等信息,為了便于使用數據庫進行單基因遺傳病的檢測,一般會對數據庫中存儲的基因變異信息對單基因遺傳病的致病性進行分級。
現有技術中,在對基因變異信息對單基因遺傳病的致病性進行分級時,大都是按照美國醫學遺傳學與基因組學學會(americancollegemedicalgeneticsgenomics,acmg)推薦的分級標準采用人工方式對基因變異信息的致病性進行分級,工作量很大,需要耗費大量的人力和時間,并且采用人工方式進行分級準確性較低。
技術實現要素:
有鑒于此,本發明實施例的目的在于提供一種基因變異致病等級確定方法及裝置,以解決現有技術中采用人工方式確定基因變異的致病等級需要耗費大量的人力物力,且準確性很低的問題。
第一方面,本發明實施例提供了一種基因變異致病等級確定方法,包括:
獲取結構化單基因遺傳病研究數據;
建立所述結構化單基因遺傳病研究數據和所述結構化單基因遺傳病研究數據的強度等級之間的第一對應關系;
確定與待分析基因變異對應的結構化單基因遺傳病研究數據的強度等級;
根據所述強度等級與預先建立的基因變異致病等級決策樹模型,確定所述待分析基因變異的致病等級。
結合第一方面,本發明實施例提供了上述第一方面的第一種可能的實現方式,其中,所述獲取結構化單基因遺傳病研究數據,包括:
收集非結構化單基因遺傳病研究數據;
將所述非結構化單基因遺傳病研究數據轉換為所述結構化單基因遺傳病研究數據。
結合第一方面的第一種可能的實現方式,本發明實施例提供了上述第一方面的第二種可能的實現方式,其中,所述將所述非結構化單基因遺傳病研究數據轉換為所述結構化單基因遺傳病研究數據,包括:
提取所述非結構化單基因遺傳病研究數據中的關鍵詞;
建立所述關鍵詞和所述非結構化單基因遺傳病研究數據的第二對應關系,得到所述結構化單基因遺傳病研究數據。
結合第一方面,本發明實施例提供了上述第一方面的第三種可能的實現方式,其中,所述建立所述結構化單基因遺傳病研究數據和所述結構化單基因遺傳病研究數據的強度等級之間的第一對應關系,包括:
獲取所述結構化單基因遺傳病研究數據中的關鍵詞;
將所述關鍵詞與預先建立的強度等級劃分標準進行匹配;
根據匹配結果,確定所述結構化單基因遺傳病研究數據對應的強度等級;
根據所述結構化單基因遺傳病研究數據對應的強度等級建立所述第一對應關系。
結合第一方面,本發明實施例提供了上述第一方面的第四種可能的實現方式,其中,所述確定與待分析基因變異對應的結構化單基因遺傳病研究數據的強度等級,包括:
獲取待分析基因變異數據;
確定所述待分析基因變異數據對應的結構化單基因遺傳病研究數據;
根據所述第一對應關系,確定所述結構化單基因遺傳病研究數據對應的強度等級。
結合第一方面,本發明實施例提供了上述第一方面的第五種可能的實現方式,所述根據所述強度等級和預先建立的基因變異致病等級決策樹模型,確定所述待分析基因變異的致病等級之前,還包括:
建立基因變異致病等級決策樹模型。
第二方面,本發明實施例提供了一種基因變異致病等級確定裝置,其中,所述裝置包括:
獲取模塊,用于獲取結構化單基因遺傳病研究數據;
建立模塊,用于建立所述結構化單基因遺傳病研究數據和所述結構化單基因遺傳病研究數據的強度等級之間的第一對應關系;
第一確定模塊,用于確定與待分析基因變異對應的結構化單基因遺傳病研究數據的強度等級;
第二確定模塊,用于根據所述強度等級和預先建立的基因變異致病等級決策樹模型,確定所述待分析基因變異的致病等級。
結合第二方面,本發明實施例提供了上述第二方面的第一種可能的實現方式,其中,所述獲取模塊包括:
收集單元,用于收集非結構化單基因遺傳病研究數據;
轉換單元,用于將所述非結構化單基因遺傳病研究數據轉換為所述結構化單基因遺傳病研究數據。
結合第二方面,本發明實施例提供了上述第二方面的第二種可能的實現方式,其中,所述建立模塊包括:
第一獲取單元,用于獲取所述結構化單基因遺傳病研究數據中的關鍵詞;
匹配單元,用于將所述關鍵詞與預先建立的強度等級劃分標準進行匹配;
第一確定單元,用于根據匹配結果,確定所述結構化單基因遺傳病研究數據對應的強度等級;
建立單元,用于根據所述結構化單基因遺傳病研究數據對應的強度等級建立所述第一對應關系。
結合第二方面,本發明實施例提供了上述第二方面的第三種可能的實現方式,其中,所述第一確定模塊包括:
第二獲取單元,用于獲取待分析基因變異數據;
第二確定單元,用于據確定所述待分析基因變異數據對應的結構化單基因遺傳病研究數據;
第三確定單元,用于根據所述第一對應關系,確定所述結構化單基因遺傳病研究數據對應的強度等級。
在本發明實施例提供的基因變異致病等級確定方法及裝置中,實現了基因變異對單基因遺傳病的致病等級的自動確定,節省大量的時間和人力,并且劃分準確。
為使本發明的上述目的、特征和優點能更明顯易懂,下文特舉較佳實施例,并配合所附附圖,作詳細說明如下。
附圖說明
為了更清楚地說明本發明實施例的技術方案,下面將對實施例中所需要使用的附圖作簡單地介紹,應當理解,以下附圖僅示出了本發明的某些實施例,因此不應被看作是對范圍的限定,對于本領域普通技術人員來講,在不付出創造性勞動的前提下,還可以根據這些附圖獲得其他相關的附圖。
圖1示出了本發明實施例所提供的基因變異致病等級確定方法的流程圖;
圖2示出了本發明實施例所提供的基因變異致病等級確定方法中,確定單基因遺傳病研究數據和強度等級之間的對應關系的流程圖;
圖3示出了本發明實施例所提供的基因變異致病等級確定方法中,致病和可能致病的決策樹模型示意圖;
圖4示出了本發明實施例所提供的基因變異致病等級確定方法中,良性和可能良性的決策樹模型示意圖;
圖5示出了本發明實施例所提供的基因變異致病等級確定裝置的結構示意圖;
圖6示出了本發明實施例所提供的基因變異致病等級確定裝置的第二種結構示意圖。
具體實施方式
為使本發明實施例的目的、技術方案和優點更加清楚,下面將結合本發明實施例中附圖,對本發明實施例中的技術方案進行清楚、完整地描述,顯然,所描述的實施例僅僅是本發明一部分實施例,而不是全部的實施例。通常在此處附圖中描述和示出的本發明實施例的組件可以以各種不同的配置來布置和設計。因此,以下對在附圖中提供的本發明的實施例的詳細描述并非旨在限制要求保護的本發明的范圍,而是僅僅表示本發明的選定實施例。基于本發明的實施例,本領域技術人員在沒有做出創造性勞動的前提下所獲得的所有其他實施例,都屬于本發明保護的范圍。
考慮到現有技術中,在對基因變異信息對單基因遺傳病的致病性進行分級時,大都是按照acmg推薦的分級標準采用人工方式確定基因變異信息對單基因遺傳病的致病等級,工作量很大,需要耗費大量的人力和時間,并且采用人工方式確定的致病等級準確性很低。基于此,本發明實施例提供了一種基因變異致病等級確定方法及裝置,下面通過實施例進行描述。
參考圖1所示,本發明實施例提供了一種基因變異致病等級確定方法,包括步驟s110-s140,具體如下。
s110,獲取結構化單基因遺傳病研究數據。
上述獲取結構化單基因遺傳病研究數據,包括:收集非結構化單基因遺傳病研究數據;將非結構化單基因遺傳病研究數據轉換為結構化單基因遺傳病研究數據。
具體的,在收集非結構化單基因遺傳病研究數據時,主要從如下幾個方面進行收集:已經發表的與變異信息相關的研究文獻、生物信息軟件預測的結果、人類基因變異數據庫中的變異頻率、權威數據庫對變異的分級及家系研究的基因測序數據等。
其中,上述收集到的非結構化單基因遺傳病研究數據的格式可以是圖片、文本等非結構化的數據,當收集到非結構化單基因遺傳病研究數據后,對上述非結構化單基因遺傳病研究數據進行結構化轉換,得到結構化單基因遺傳病研究數據,結構化數據為計算機可以識別的語言,這樣當將結構化單基因遺傳病研究數據存儲到單基因遺傳病數據庫中后,可以實現對結構化單基因遺傳病研究數據的自動檢索、讀取及匹配等。
上述將非結構單基因遺傳病研究數據轉換為結構化單基因遺傳病研究數據,包括如下過程:提取非結構化單基因遺傳病研究數據中的關鍵詞;建立上述關鍵詞和非結構化單基因遺傳病研究數據的第二對應關系,得到結構化單基因遺傳病研究數據。
在本發明實施例中,首先按照acmg標準對上述單基因遺傳病研究數據進行分類,確定出上述單基因遺傳病研究數據所屬的種類,具體的,單基因遺傳病研究數據的種類包括人群數據、預測數據、功能數據、分離數據、新發變異數據、等位基因數據、其它數據庫數據及其它數據八種。
在本發明實施例中,上述人群數據指的是基因變異在人群中的變異頻率的研究或記錄;上述預測數據指的是生物信息分析軟件對基因變異的影響的預測結果;上述功能數據指的是對基因變異在活體內或者活體外的分子功能研究;上述分離數據指的是對患有單基因遺傳病的家系進行疾病和基因變異是否共分離的研究;上述新發變異數據指的是對未患有基因遺傳病的家系中出現單基因遺傳病患者、患者攜帶發現新發變異、并且患者父母不攜帶該變異的研究;上述等位基因數據指的是對于單基因遺傳病在變異位點的順式或反式基因上發現致病性變異的研究;上述其它數據庫數據指的是權威的研究或者數據庫(比如說,hgmd,clinvar等)對基因變異的致病性分類結果;上述其它數據指的是其它方面的研究。
在本發明實施例中,可以采用基于語義的關鍵詞提取(semantic-basedkeywordextraction,ske)算法提取非結構化單基因遺傳病研究數據中的關鍵詞。
具體的,上述單基因遺傳研究數據包括基因變異信息、每個基因變異對應的單基因遺傳病以及該單基因遺傳病的發病情況。
s120,建立上述結構化單基因遺傳病研究數據和結構化單基因遺傳病研究數據的強度等級之間的第一對應關系。
不同的單基因遺傳病研究數據對應著不同的強度等級,上述強度等級指的是單基因遺傳病研究數據對確定基因變異致病性的影響力的大小。
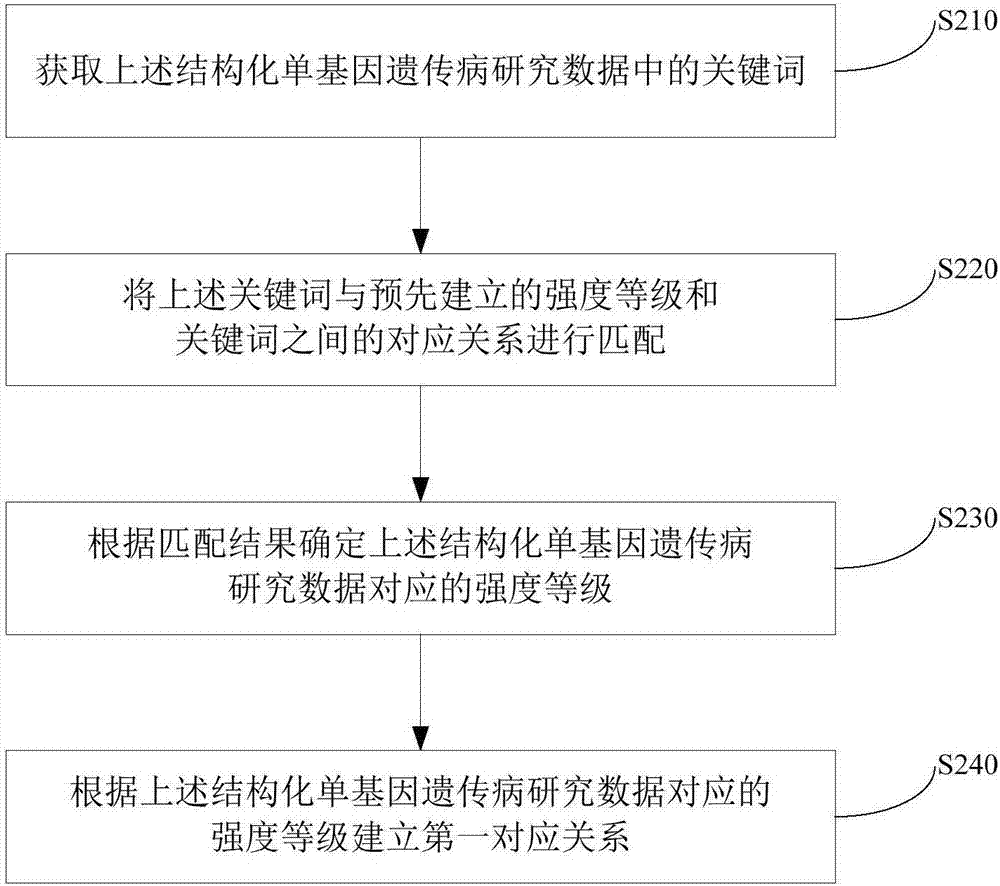
參考圖2所示,上述建立結構化單基因遺傳病研究數據和結構化單基因遺傳病研究數據的強度等級之間的對應關系,包括步驟s210-s230,具體如下:
s210,獲取上述結構化單基因遺傳病研究數據中的關鍵詞;
s220,將上述關鍵詞與預先建立的強度等級劃分標準進行匹配;
s230,根據匹配結果確定上述結構化單基因遺傳病研究數據對應的強度等級;
s240,根據上述結構化單基因遺傳病研究數據對應的強度等級建立第一對應關系。
具體的,上述單基因遺傳病研究數據可以分為致病性的單基因遺傳病研究數據和良性變異的單基因遺傳病研究數據,而致病性的單基因遺傳病研究數據分為如下四個等級:致病性非常強(pathogenicverystrong,pvs)、致病性強(pathogenicstrong,ps)、致病性中等(pathogenicnoderate,pm)和致病性支持(pathogenicsupporting,pp);而良性的單基因遺傳病研究數據又分為如下三個等級:良性變異獨立(benignstand-alone,ba)、良性變異強(benignstrong,bs)和良性變異支持(benignsupporting,bp)。
上述pvs指的變異為無效變異,并且變異所在基因的功能缺失是相關的單基因遺傳病的致病機制。具體的,上述無效變異包括無義突變、移碼變異、位于剪接位點+1/2堿基位置或-1/2堿基位置的變異、初始密碼子變異、單個或多個外顯子缺失,這些變異影響基因轉錄和翻譯過程,不能產生功能正常的基因產物,所以稱為無效變異。
其中,上述ps包括ps1、ps2、ps3和ps4,ps1指的是堿基變異引發的氨基酸變化和之前定義的某一致病變異一致,ps2指的是在無家族史的病人中檢測到的新發變異,ps3指的是體內或體外功能研究證實變異對基因和基因產物的損害效應,ps4指的是變異在受影響的個體中的發生率顯著高于對照人群中的發生率。
上述pm包括pm1、pm2、pm3、pm4、pm5和pm6,pm1指的是基因變異位于突變熱點、關鍵區域或經過驗證的功能區,且在該區域沒有良性變異,pm2指的是外顯子組測序項目(exomesequencingproject,esp),千人基因組計劃(1000genomesproject)和外顯子組集合聯合(exomeaggregationconsortium,exac)等數據庫顯示對照人群中沒有變異或者變異的頻率極低,pm3指的是對于隱性疾病,在反式基因上發現致病性突變,pm4指的是引起蛋白長度改變的變異,具體包括發生于非重復區的框內缺失和插入,終止丟失變異,pm5指的是新發現的錯義突變,且該氨基酸殘基處發生的另一錯義突變已被認為是致病的,pm6指的是假定是新發變異,但是沒有進行父母的驗證。
上述pp包括pp1、pp2、pp3、pp4和pp5,pp1指的是在受影響的家族成員中該變異與疾病呈現共分離,該變異所在基因被認為是可以致病的,pp2指的是錯義突變,良性錯義變異在該基因中發生的頻率較低,并且錯義突變被認為是導致疾病的一個常見機制,pp3指的是多項計算數據均顯示該變異會對基因或基因產物產生有害影響,pp4指的是患者的表型或家族病史對一個單一遺傳因素的疾病具有高度特異性,pp5指的是較權威的研究和數據庫支持該變異是致病的,但缺乏實驗室獨立評估的證據。
上述ba指的是在外顯子組測序項目、千人基因組計劃或外顯子組集合聯合等數據庫中該等位基因頻率大于5%。
上述bs包括bs1、bs2、bs3和bs4,bs1指的是等位基因頻率大于疾病的預期發病率,bs2指的是當疾病為隱性、顯性、x-連鎖遺傳,且在年幼階段表現出完全外顯時,仍可在健康的成體中檢測到純合、雜合和半合子突變,bs3指的是體內或體外功能測試已表明對蛋白功能或剪接無有害影響,bs4指的是在一個家族的發病成員中缺乏分離。
上述bp包括bp1、bp2、bp3、bp4、bp5、bp6和bp7,bp1指的是錯義突變,已知該基因中原發的截短突變可導致發病,bp2指的是對于一個完全外顯的顯性基因或疾病,在反式基因上發現致病性變異,bp3指的是功能未知的重復區中發生的框內缺失或插入,bp4指的是多項計算分析證據表明對基因或基因產物無影響,bp5指的是一個病例中發現的變異,該疾病中存在可替代的分子機制,bp6指的是較權威研究和數據庫評定變異為良性,但該證據無法用試驗進行獨立評估,bp7指的是一個同義(或沉默)突變,剪接算法預測其對剪接結果無影響,不會產生新的剪接位點并且該核苷酸并非高度保守。
上述強度等級劃分標準描述了上述每個級別的單基因遺傳病研究數據的描述。
上述致病性強度等級中的很強對應pvs、強對應ps1-ps4,中等水平對應pm1-pm6,支持對應pp1-pp5;上述良性的強度等級中的獨立證據對應ba,強對應bs1-bs4,支持對應bp1-bp7。
因此,將上述關鍵詞與預先建立的強度等級劃分標準進行匹配時,首先根據上述關鍵詞匹配出該結構化單基因遺傳病研究數據對應的級別,再根據該級別所對應的強度等級確定出該結構化單基因遺傳病研究數據對應的強度等級。
其中,上述劃分標準是預先建立好的,即采用本發明實施例提供的方法確定基因變異的致病性之前,就已經建立了上述劃分標準,具體的,上述強度等級和關鍵詞之間的對應關系可以根據acmg推薦的標準建立。
s130,確定與待分析基因變異對應的結構化單基因遺傳病研究數據的強度等級。
在本發明實施例中,將需要分析的基因變異記為待分析基因變異,首先確定出待分析基因變異對應的結構化單基因遺傳病研究數據,再根據結構化單基因遺傳病研究數據與強度等級之間的對應關系,確定出待分析基因變異對應的結構化單基因遺傳病研究數據對應的強度等級,具體包括:
獲取待分析基因變異數據;確定待分析基因變異對應的結構化單基因遺傳病研究數據;根據第一對應關系,確定上述結構化單基因遺傳病研究數據對應的強度等級。
在本發明實施例中,上述待分析基因變異數據包括待分析變異所在的染色體、待分析變異在染色體的起始物理位置和終止物理位置、待分析變異在變異前的堿基序列和待分析變異在變異后的堿基序列等。
將上述待分析基因變異數據與上述第一對應關系中每個結構化單基因遺傳病研究數據進行匹配,確定出上述待分析基因變異數據對應的結構化單基因遺傳病研究數據,這樣再根據上述結構化單基因遺傳病研究數據與強度等級的第一對應關系,確定出上述待分析基因變異數據對應的結構化單基因遺傳病研究數據的強度等級。
s140,根據上述強度等級和預先建立的基因變異致病等級決策樹模型,確定待分析基因變異的致病等級。
上述致病等級包括致病、可能致病、良性、可能良性和不確定意義。
上述致病指的是某個基因變異會引起相應的單基因遺傳病;上述良性指的是某個變異不會引起相應的單基因遺傳病;上述可能致病指的是某個變異引起相應的單基因遺傳病的可能性在90%以上;上述可能良性指的是某個變異不會引起相應的單基因遺傳病的可能性在90%以上;上述不確定意義指的是某個基因變異與相應的單基因遺傳病發病關系不確定。
其中,上述基因變異致病等級決策樹模型是預先建立好的,即在采用本發明實施例提供的基因變異致病性確定方法或者在確定待分析基因變異的致病等級之前,需要建立基因變異致病等級決策樹模型,具體的,上述基因變異致病等級決策樹模型是根據acmg推薦的基因變異致病性分級標準建立的,過程包括:
根據acmg推薦的基因變異致病性分級標準確定出基因變異致病等級決策樹模型的分裂節點,根據確定出的分裂節點,建立基因變異致病等級決策樹模型。
在本發明實施例中,單基因遺傳病研究數據的強度等級為決策樹模型的非葉節點,每個強度等級對應的單基因遺傳病研究數據的數量為決策樹模型的分支,致病等級結果為葉節點,根據強度等級的大小依次確定決策樹模型的非葉節點,對于致病和可能致病決策樹模型,如圖3所示,強度等級非常強(verystrong)作為決策樹模型的根節點,強(strong)作為決策樹模型上非常強的子節點,中等(moderate)作為決策樹模型上強的子節點,而支持(supporting)作為決策樹模型上中等的子節點,最后得出的致病或者可能致病的結果作為決策樹型的葉節點,在決策樹模型的分支上的數字為該強度等級對應的單基因遺傳病研究數據的數量。
對于良性和可能良性決策樹模型,如圖4所示,強度等級獨立(stand-alone)是該決策樹模型的根節點,強(strong)是該決策樹模型上獨立的子節點,支持(supporting)是該決策樹模型上強的子節點,最后得出的良性或者可能良性作為決策樹模型的葉節點。
具體的,acmg推薦的基因變異致病性分級標準如下所示:
致病對應的判斷標準為:1個pvs等級的單基因遺傳病研究數據和至少一個ps1-ps4等級的單基因遺傳病研究數據;或者1個pvs等級的單基因遺傳病研究數據和至少兩個pm1-pm6等級的單基因遺傳病研究數據;或者1個pvs等級的單基因遺傳病研究數據、1個pm1-pm6等級的單基因遺傳病研究數據和1個pp1-pp5等級的單基因遺傳病研究數據;或者1個pvs等級的單基因遺傳病研究數據和至少兩個pp1-pp5等級的單基因遺傳病研究數據;或者至少2個ps1-ps4等級的單基因遺傳病研究數據;或者1個ps1-ps4等級的單基因遺傳病研究數據和至少3個pm1-pm6等級的單基因遺傳病研究數據;或者1個ps1-ps4等級的單基因遺傳病研究數據、2個pm1-pm6等級的單基因遺傳病研究數據和至少兩個pp1-pp5等級的單基因遺傳病研究數據;或者1個ps1-ps4等級的單基因遺傳病研究數據、1個pm1-pm6等級的單基因遺傳病研究數據和至少4個pp1-pp5等級的單基因遺傳病研究數據。
可能致病對應的判斷標準為:1個pvs等級的單基因遺傳病研究數據和1個pm1-pm6等級的單基因遺傳病研究數據;或者1個ps1-ps4等級的單基因遺傳病研究數據和1個或2個pm1-pm6等級的單基因遺傳病研究數據;或者1個ps1-ps4等級的單基因遺傳病研究數據和至少2個pp1-pp5等級的單基因遺傳病研究數據;或者至少3個pm1-pm6等級的單基因遺傳病研究數據;或者2個pm1-pm6等級的單基因遺傳病研究數據和2個pp1-pp5等級的單基因遺傳病研究數據;或者1個pm1-pm6等級的單基因遺傳病研究數據和至少4個pp1-pp5等級的單基因遺傳病研究數據。
良性對應的判斷標準為:1個ba等級的單基因遺傳病研究數據或者至少2個bs1-bs4等級的單基因遺傳病研究數據。
可能良性對應的判斷標準為:1個bs1-bs4等級的單基因遺傳病研究數據和1個bp1-bp7等級的單基因遺傳病研究數據;或者至少2個bp1-bp7等級的單基因遺傳病研究數據。
致病性不明確的對應的判斷標準為:未滿足上述判斷標準;或者良性致病對應的單基因遺傳病研究數據與致病的對應的單基因遺傳病研究數據相互矛盾。
本發明實施例提供的基因變異致病等級確定方法,實現了基因變異對單基因遺傳病的致病等級的自動確定,節省大量的時間和人力,并且劃分準確。
參考圖5所示,本發明實施例還提供了一種基因變異致病等級確定裝置,該裝置用于執行本發明實施例提供的基因變異致病性確定方法,該裝置包括獲取模塊310、建立模塊320、第一確定模塊330和第二確定模塊340;
上述獲取模塊310,用于獲取結構化單基因遺傳病研究數據;
上述建立模塊320,用于建立上述結構化單基因遺傳病研究數據和結構化單基因遺傳病研究數據的強度等級之間的第一對應關系;
上述第一確定模塊330,用于確定與待分析基因變異對應的結構化單基因遺傳病研究數據的強度等級;
上述第二確定模塊340,用于根據上述強度等級與預先建立的基因變異致病等級決策樹模型,確定待分析基因變異的致病等級。
具體的,本發明實施例中的獲取模塊310獲取結構化單基因遺傳病研究數據是通過收集單元和轉換單元實現的,具體包括:
上述收集單元,用于收集非結構化單基因遺傳病研究數據;上述轉換單元,用于將非結構化單基因遺傳病研究數據轉換為結構化單基因遺傳病研究數據。
其中,參考圖6所示,上述建立模塊320建立上述結構化單基因遺傳病研究數據和強度等級之間的對應關系,是通過第一獲取單元321、匹配單元322和第一確定單元323和建立單元324實現的,具體包括:
上述第一獲取單元321,用于獲取上述結構化單基因遺傳病研究數據中的關鍵詞;上述匹配單元322,用于將上述關鍵詞與預先建立的強度等級劃分標準進行匹配;上述第一確定單元323,用于根據匹配結果,確定結構化單基因遺傳病研究數據對應的強度等級;上述建立單元324,用于根據上述結構化單基因遺傳病研究數據對應的強度等級建立上述第一對應關系。
上述第一確定模塊330確定待分析基因變異對應的結構化單基因遺傳病研究數據的強度等級是通過第二獲取單元、第二確定單元和第三確定單元實現的,具體包括:
上述第二獲取單元,用于獲取待分析基因變異數據;上述第二確定單元,用于確定待分析基因變異數據對應的結構化單基因遺傳病研究數據;上述第三確定單元,用于根據上述第一對應關系,確定結構化單基因遺傳病研究數據對應的強度等級。
本發明實施例提供的基因變異致病等級確定裝置,實現了基因變異對單基因遺傳病的致病等級的自動確定,節省大量的時間和人力,并且劃分準確。
本發明實施例所提供的基因變異致病等級確定裝置可以為設備上的特定硬件或者安裝于設備上的軟件或固件等。本發明實施例所提供的裝置,其實現原理及產生的技術效果和前述方法實施例相同,為簡要描述,裝置實施例部分未提及之處,可參考前述方法實施例中相應內容。所屬領域的技術人員可以清楚地了解到,為描述的方便和簡潔,前述描述的系統、裝置和單元的具體工作過程,均可以參考上述方法實施例中的對應過程,在此不再贅述。
在本發明所提供的實施例中,應該理解到,所揭露裝置和方法,可以通過其它的方式實現。以上所描述的裝置實施例僅僅是示意性的,例如,所述單元的劃分,僅僅為一種邏輯功能劃分,實際實現時可以有另外的劃分方式,又例如,多個單元或組件可以結合或者可以集成到另一個系統,或一些特征可以忽略,或不執行。另一點,所顯示或討論的相互之間的耦合或直接耦合或通信連接可以是通過一些通信接口,裝置或單元的間接耦合或通信連接,可以是電性,機械或其它的形式。
所述作為分離部件說明的單元可以是或者也可以不是物理上分開的,作為單元顯示的部件可以是或者也可以不是物理單元,即可以位于一個地方,或者也可以分布到多個網絡單元上。可以根據實際的需要選擇其中的部分或者全部單元來實現本實施例方案的目的。
另外,在本發明提供的實施例中的各功能單元可以集成在一個處理單元中,也可以是各個單元單獨物理存在,也可以兩個或兩個以上單元集成在一個單元中。
所述功能如果以軟件功能單元的形式實現并作為獨立的產品銷售或使用時,可以存儲在一個計算機可讀取存儲介質中。基于這樣的理解,本發明的技術方案本質上或者說對現有技術做出貢獻的部分或者該技術方案的部分可以以軟件產品的形式體現出來,該計算機軟件產品存儲在一個存儲介質中,包括若干指令用以使得一臺計算機設備(可以是個人計算機,服務器,或者網絡設備等)執行本發明各個實施例所述方法的全部或部分步驟。而前述的存儲介質包括:u盤、移動硬盤、只讀存儲器(rom,read-onlymemory)、隨機存取存儲器(ram,randomaccessmemory)、磁碟或者光盤等各種可以存儲程序代碼的介質。
應注意到:相似的標號和字母在下面的附圖中表示類似項,因此,一旦某一項在一個附圖中被定義,則在隨后的附圖中不需要對其進行進一步定義和解釋,此外,術語“第一”、“第二”、“第三”等僅用于區分描述,而不能理解為指示或暗示相對重要性。
最后應說明的是:以上所述實施例,僅為本發明的具體實施方式,用以說明本發明的技術方案,而非對其限制,本發明的保護范圍并不局限于此,盡管參照前述實施例對本發明進行了詳細的說明,本領域的普通技術人員應當理解:任何熟悉本技術領域的技術人員在本發明揭露的技術范圍內,其依然可以對前述實施例所記載的技術方案進行修改或可輕易想到變化,或者對其中部分技術特征進行等同替換;而這些修改、變化或者替換,并不使相應技術方案的本質脫離本發明實施例技術方案的精神和范圍。都應涵蓋在本發明的保護范圍之內。因此,本發明的保護范圍應所述以權利要求的保護范圍為準。
- 還沒有人留言評論。精彩留言會獲得點贊!