DNA中數字信息的高容量存儲的制作方法
本申請是分案申請,其原申請的國際申請號為pct/ep2013/061300,國際申請日是2013年05月31日,中國國家申請號為201380028511.8,進入中國的日期為2014年11月28日,發明名稱為“dna中數字信息的高容量存儲”。
本公開涉及存儲dna中數字信息的方法和設備。
背景技術:
dna具備保持巨額數量信息的能力,易于以緊湊結構長期保存1,2。利用dna作為數字信息的存儲器的想法自1995年3已經存在。dna存儲的物理實現迄今為止僅存儲了微量的信息——典型的是少數英文文本的單詞4-8。發明人沒有意識到編碼在物理dna中的大規模存儲和對任意規格數字信息的還原,而非磁性基底或光學基底上的數據存儲。
目前,dna合成是集中應用于生物醫藥的專業技術。dna合成的成本在過去的幾十年已經穩步縮減。相比于磁帶上的數據存儲每過3至5年會少見但定期地轉移到新介質,推斷在什么時間段如本文所公開的dna分子上的數據存儲將會比其當前長期存檔過程更具成本效益是有趣的。當前dna合成的“現成”技術相當于一美元約100字節的價格。市場上可從agilent科技(圣克拉拉,加利福尼亞州)購買的更時新的技術可能會大幅縮減該成本。然而,仍然需要建立賬戶,用于磁帶介質之間的定期數據轉移。問題是,該數據傳遞的成本,以及該成本是固定不變的還是隨時間削減的。如果假定大量成本是固定不變的,則存在一個時間范圍,其間利用dna分子進行數據存儲比常規磁帶介質上的數據存儲更具成本效益。在400年以后(至少80次介質轉移),利用dna分子的數據存儲可能已經具有成本效益。
本公開中描述了一種比先前使用的程序存儲更多信息的實用編碼解碼程序。發明人已經對五個計算機文件進行編碼——總共757051字節的(739kb)硬盤存儲并具有約為5.2×106比特的香農信息9——變為dna碼。發明人隨后合成了該dna,將該合成dna從美國經英國運輸到德國,對dna進行測序并以100%的精確度重構了五個計算機文件。
該五個計算機文件包括一個英文語言文本(莎士比亞十四行詩的全部154句),一個經典科技論文的pdf文件10,一個jpeg色彩的照片和一個mp3格式的音頻文件,包含26秒的演講(選自馬丁路德金的演講“我有一個夢想”)。該數據存儲代表了近800倍于已知的先前基于dna的存儲的信息量,并涵蓋了更豐富的數字格式。結果證明,dna存儲正逐漸成為現實,并可以在未來提供有成本效益的數字信息存檔方法,且用于慢速存取、多年代存檔任務可能已經具有成本效益。
現有技術
在易于獲取的情況下1,2,自1995年以來3,穩定存儲信息的dna高容量已經使得dna成為極具吸引力的信息存儲目標。除信息密度之外,dna分子作為信息載體具有已證實的業績記錄,dna分子的壽命已知,且事實上作為地球生命的基礎,只要有基于dna的智能生命體存在1,2,操作、存儲和讀取dna分子的方法將依然是持續技術創新的主題。基于活性載體dna5-8(在活體(invivo)dna分子中)和合成dna4,1(在離體(invitro)dna中)的數據存儲系統已經被提出。在活體內的數據存儲系統具有若干缺陷。這些缺陷包括對數量、染色體成分和在活性載體有機體中可操作而不影響dna分子生存力的位置的限制。此種活性載體有機體的示例包括但不限于細菌。生存力的下降包括容量的縮減和信息編碼方案的復雜度的增加。此外,胚系突變和體細胞突變將引發所保存信息的保真度和解碼信息隨時間下降,并可能引發對活dna存儲條件細致監管的需求。
相比之下,“分離的dna”(即,離體dna)更易于“寫入”,且對幾萬年前11-14的樣本的非活dna實例進行的例行還原顯示,在易于獲取且少量維護的環境下(即,寒冷,干燥和黑暗的環境)15-17,良好制備的非活dna樣本應具有格外長的生命期限。
在dna中進行信息(也稱數據)存儲的先前工作典型地集中于在dna中以編碼形式“寫入”人類可讀的消息,而后通過確定dna的序列和解碼該序列來“讀取”該編碼的人類可讀的消息。在dna計算領域的工作產生了原則上容許大型聯合associative(相聯)存儲器的方案3,18-20,但尚無將這項工作推進到實用dna存儲方案的嘗試。圖1示出了14個在先前研究中(在y軸標注對數刻度)被成功編碼和還原的信息數量。為14個先前實驗(空心圓)和本公開(實心圓)示出了點。以這種方式存儲的最大數量的人類可讀消息為1280個英文語言文本字符8,約相當于6500比特的香農信息9。
印度科學與工業研究委員會提交了一份序列號為us2005/0053968(巴拉得瓦杰等)的美國專利申請,教導了一種在dna中存儲信息的方法。美國968號申請的方法包括使用編碼方法,其利用4個dna堿基表示拓展ascii字符集中的每一個字符。于是產生了合成的dna分子,其包括數字信息、加密密鑰,并在每個側面與引物序列相接。最后,合成的dna被并入存儲dna。在dna數量過大的情況下,信息可以被分裂成為若干片段。美國968號專利公開的方法可以通過匹配某片段的頭端引物和下一片段的尾端引物來重構分裂的dna片段。
已知其它描述了在dna中存儲信息的技術的專利文獻。例如,美國專利6,312,911教導了一種密寫方法,用于隱藏dna中編碼的消息。該方法包括運用基因組dna樣本隱藏dna編碼消息,隨后進一步將dna樣本隱藏為微點。該美國911號專利的申請特別地用于機密信息的隱藏。這樣的信息通常具有有限長度,因此文件不會討論如何存儲更長長度的信息項。相同的發明人提交了國際專利申請,并作為第wo03/025123號國際公布公開。
技術實現要素:
公開了一種用于存儲信息項的方法。該方法包括編碼在信息項中的字節。使用一方案(schema)由dna核苷酸來表示該編碼字節,產生計算機模擬(in-silico)的dna序列。下一步,dna序列被拆分成若干個重疊的dna片段,且標引信息被添加到若干個dna片段上。最后,若干個dna片段被合成并存儲。
對dna片段添加標引信息意味著片段在表示信息項的dna序列中的位置能夠被單一識別。沒有必要依賴頭端引物與尾端引物的匹配。這使得,即使片段之一不能正確重現,也可以還原幾乎整個信息項。如果不存在標引信息,則存在風險,也即,如果因“孤兒”(orphan)片段而導致片段不能夠相互匹配,可能不能夠正確重現整個信息項,所述孤兒片段是其在dna序列中的位置不能清楚識別的片段。
利用重疊的dna片段意味著在信息項的存儲中寫入了一定程度的冗余。如果某一dna片段不能夠被解碼,則該編碼字節仍然能夠從相鄰的dna片段還原。因此冗余成為了系統的組成部分。
可以利用已知dna合成技術制作dna片段的若干個副本。這提供了額外程度的冗余,以使得信息項能夠被解碼,即使dna片段中的某些副本損壞并不能被解碼。
在發明的一個方面,用于解碼的表示方案(representationschema)被設計為相鄰的dna核苷酸各不相同。這樣增加了合成、重現和測序(讀取)dna片段的可靠性。
在本發明的進一步方面,向標引信息添加了奇偶校驗(parity-check)。該奇偶校驗使得錯誤合成、重現或測序被識別出來。奇偶校驗能夠被拓展(expanded)并包括糾錯信息。
合成dna片段的交替片段是反向互補的。它們在dna中提供了額外的冗余度,并意味著如果任一dna片段損壞,會有更多可得信息。
附圖說明
圖1為存儲在dna中并成功還原的信息數量的時間函數圖;
圖2示出了本公開方法的示例;
圖3示出了存儲隨時間的成本效益的圖示;
圖4示出了自反向互補模式的基序;
圖5示出了編碼效率;
圖6示出了誤碼率;
圖7示出了方法的編碼的流程圖;
圖8示出了方法的解碼的流程圖;
具體實施方式
迄今,實現實用dna存儲的主要挑戰之一是根據指定(specified)設計創造長序列dna的困難。dna長序列要求存儲大量數據文件,諸如長文本項和視頻文件。更優選的是,利用每個設計的dna的若干個副本的編碼。該冗余防止了編碼和解碼錯誤,下文將做解釋。使用基于獨立的長dna鏈的系統來編碼每條(尤其是大量的)消息8不具有成本效益。發明人已開發了一種方法,運用與每個dna片段相關的“標引”信息來指示dna片段在假設編碼整個消息的更長的dna分子中的位置。
發明人運用編碼理論的方法來加強dna片段上的編碼消息的可還原性,包括禁止dna均聚物(即,存在不只一個相同堿基),已知其在現存的高通量技術中具有更高的誤碼率。發明人進一步并入了簡單的錯誤檢測組件,類似于碼中標引信息的奇偶校驗位9。更復雜的方案,包括但不限于錯誤校正碼9,以及當前切實用于信息學的、實質上任何形式的數字數據安全(例如,基于raid的方案21),可以在dna存儲方案3的未來發展中實現。
發明人選擇了五個計算機文件進行編碼,作為本公開dna存儲的概念驗證。選擇了使用一系列常見格式的文件,而非限制文件為人類可讀信息。這演示了本公開的存儲任意類型數字信息的教導。該文件包含莎士比亞十四行詩的全部154句(txt格式)、參考文獻10的全部文本和圖像(pdf格式)、中等分辨率色彩的、歐洲分子生物學實驗室-歐洲生物信息研究所的照片(jpeg2000格式)、從馬丁路德金的“我有一個夢想”的演講中提取了26秒時長(mp3格式),和一個定義霍夫曼代碼的文件,本研究使用霍夫曼代碼將字節轉換為三進制數字(作為人類可讀的文本文件)。
該選擇用于dna存儲的五個文件如下:
wssnt10.txt—107738字節—ascii文本格式。莎士比亞十四行詩的全部154句(來自古登堡計劃,http://www.gutenberg.org/ebooks/1041)。
watsoncrick.pdf—280864字節—pdf格式文件。沃森和克里克(1953)描述dna結構的文獻10(經修正以實現更高的壓縮率從而得到較小的文件,來自“自然”網站,http://www.nature.com/nature/dna50/archive.html)。
ebi.jp2—184264字節—jpeg2000格式圖像文件。歐洲分子生物學實驗室-歐洲生物信息研究所(自行拍攝)的彩色照片(16.7m色彩,640x480像素分辨率)。
mlk_excerpt_vbr_45-85.mp3—168539字節—mp3格式聲音文件。從馬丁路德金的“我有一個夢想”的演講中提取了26秒時長(來自http://www.americanrhetoric.com/speeches/mlkihaveadream.htm,經修正以實現更高的壓縮率:可變比特率,典型為48-56kps;采樣頻率為44.1khz)。
view_huff3.cd.new—15646字節—ascii文件。定義本研究用來將字節轉換為三進制數字(三進制數位)的霍夫曼代碼的人類可讀文件。
五個計算機文件一共包含757051字節,約等于5.2×106位的香農信息,或800倍于作為先前最大已知存儲量的、編碼和還原的人類設計信息(見圖1)。
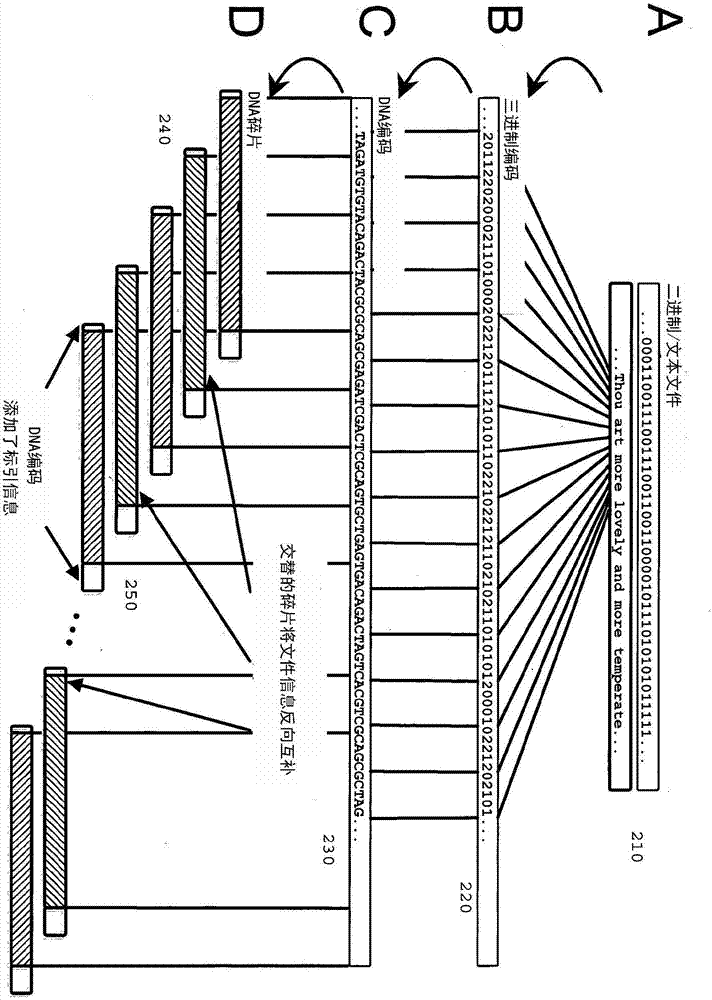
對每個計算機文件進行dna編碼是用軟件計算的,且該方法在圖7中示出。在本文描述的本發明700的一個方面,包含每個計算機文件210的字節在步驟720處通過編碼方案表示為不含均聚物的dna序列230,用以產生編碼文件220,以形成dna序列230的五或六個堿基(見下)替換每個字節。構造用在編碼方案中的碼,以便容許接近有限運行長度通道(即無重復核苷酸)的最佳信息容量的直接編碼。而將理解,也可以使用其他編碼方案。
所得的計算機模擬的dna序列230過長,而不易于標準低聚核苷酸合成的產生。每個dna序列230因此在步驟730被拆分為重疊的片段240,其長度為100個堿基,具有重疊的75個堿基。為降低被引入堿基的任一特定排列中的系統合成錯誤風險,于是這些片段的交替片段在步驟740處被轉變為它們的反向互補序列,意為每個堿基被“寫入”四次,每個方向兩次。每個片段在750處采用標引信息250進行擴充,加之簡單的錯誤檢測信息,標引信息250可確定片段240來源于哪個計算機文件以及在該文件210上的位置。標引信息250也在步驟760處作為無重復的dna核苷酸進行編碼,并在步驟770處被添加到dna片段240的100個信息存儲堿基。將理解,將dna片段240分為具有重疊的75個堿基的100個堿基的長度純粹是任意的。也可以使用其它長度和重疊部分,這并非是對本發明的限制。
總之,所有的五個計算機文件被153335串dna所表示。每一串dna包含117個核苷酸(編碼原始數字信息和標引信息)。所使用的編碼方案具有合成dna的多個特征(例如統一的片段長度,無均聚物),使該合成dna明顯不具有自然(生物)來源。因此,很明顯合成dna具有思慮周密的設計以及編碼信息2。
如上所述,也可以使用其他對dna片段240的編碼方案,例如提供增強的錯誤校正性能。增加標引信息的數量將使其更明確,以便使得更多或更大的文件能夠被編碼。已經提出,嵌套引物分子存儲器(npmm)方案19達到了16.8m單一地址的實用最大容量20,且似乎沒有原因顯示,本公開的方法不能超越它以使得幾乎任意大量的信息能夠被編碼。
為了避開在dna片段240中的系統模式,對編碼方案的拓展可以是增加變動信息。嘗試了兩種途徑。第一種途徑包含對dna片段240中的信息進行“洗牌”,如果知道“洗牌”的模式,可以恢復該信息。在本公開的一個方面,不同模式的洗牌被用于不同的dna片段240。
另一個途徑是在每個dna片段240的信息中增加隨機度。對此可運用一系列隨機數字,利用該系列隨機數字的模加,且這些數字包含編碼到dna片段240中的信息。如果知道所用的隨機數字序列,則可以在解碼中輕易地通過模減恢復該信息。在本發明的一個方面,不同系列的隨機數字被用于不同的dna片段240。
在步驟720處的數字信息編碼執行如下:用軟件編碼存儲在硬盤驅動器中的數字信息的五個計算機文件210(表示在圖2a中)。五個計算機文件210中每個文件的每個字節在步驟720處被編碼,并經由三進制數字(“三進制數位”0,1,2)表示為一系列dna堿基,運用表1(下文)列出的為特定目的設計的霍夫曼代碼來產生編碼文件220。該示例性編碼方案概括地在圖2b中展示。256個可能字節中的每一個被5至6個三進制數位表示。隨后,每個三進制數位被編碼為從不同于上一個核苷酸(圖2c)的三個核苷酸中選出的一個dna核苷酸230。換言之,在為本公開的此方面所選的編碼方案中,三個核苷酸中的每一個均與上一個不同,以保證沒有均聚物。所得的dna序列230在步驟730處被拆分為dna片段240,具有100個堿基的長度,如圖2d所示。每個dna片段與上一個dna片段重疊75個堿基,以便給予dna片段一個易于合成并提供冗余的長度。交替的dna片段是反向互補的。
標引信息250包括兩個三進制數位用于文件識別(在該實現中,可區別32=9個文件),12個三進制數位用于文件內部位置信息(每個文件312=531441個位置),以及一個“奇偶校驗”三進制數位。標引信息250在步驟760處編碼為無重復的dna核苷酸,并在步驟770處被添加到100個信息存儲堿基上。每個標引的dna片段240具有另一個堿基,在步驟780處添加到每一端,符合“無均聚物”的原則,其能夠在實驗的“讀取”階段表明整個dna片段240是否為反向互補的。
總之,五個計算機文件210由153335串dna表示,每串包含117個(1+100+2+12+1+1個)核苷酸(編碼原始數字信息和標引信息)。
本文描述的本發明的方面中,每串數據編碼組件可以包含香農信息,每dna堿基5.07比特,其接近理論最佳的每dna堿基5.05比特,用于運行長度限制為1的4進制通道。標引實現250容許314=4782969個單一數據位置。將用于指定文件和文件內部位置的標引三進制數位的數目從2到16增加,給出316=43046721個單一位置,超過了npmm方案19,20的16.8m的實用最大值。
步驟790處的dna合成過程也用于向每個低聚核苷酸(低聚糖)的每一端并入33bp的銜接頭,來促進在illumina測序平臺上的測序:
5’銜接頭:acactctttccctacacgacgctcttccgatct
3’銜接頭:agatcggaagagcggttcagcaggaatgccgag
該153335個dna片段設計240在步驟790處合成為三個不同的運行段(dna片段240被隨機地分入運行段),利用上述22,23新版的agilent科技的ols(低聚糖庫合成)過程,為每個dna片段設計生成約1.2x107個副本。可以見到錯誤僅出現在每500個堿基中的一處,且對于dna片段240的不同副本是獨立出現的。agilent科技調整了先前24開發的酰胺三酯合成法,并在微陣列合成平臺上就地使用了agilentsureprint的噴墨打印和流動池反應器技術。具有無水室的噴墨打印容許向二維平面表面的有限耦合區域傳遞非常小量的亞磷酰胺,得到成百上千增加的并行堿基。隨后的氧化和脫三苯甲基在流動池反應器中實現。一旦dna合成完成,低聚核苷酸將被從表面分離并去保護25。
添加到dna片段的銜接頭使得dna片段的若干個副本能夠易于制作。沒有銜接頭的dna片段將需要額外的化學過程來“啟動”通過向dna片段的端添加額外的組來合成若干個副本的化學過程。
利用超過千倍的亞磷酰胺和催化劑方案,可獲得高達~99.8%的耦合效率。類似的,超過數百萬倍的脫三苯甲基藥劑驅使5’-羥基保護基團被移除,使之接近完成。在流動池反應器中的控制過程顯著地降低了脫嘌呤作用,其為最普遍的副反應22。可以并行合成高達244000個單一序列,并作為~1–10皮摩爾池的低聚糖傳遞。
凍干低聚糖的三個樣本整晚在氨丁三醇緩沖劑中以4℃進行培養,通過移液管和渦流周期性混合,并最終在50℃下進行培養1小時,達到5ng/ml的濃度。因為有不溶性的物質殘留,樣本以4℃被另外靜置5天,每天有2-4次混合。隨后樣本以50℃培養1小時、以68℃培養10分鐘,并且從ampurexp順磁珠(貝克曼庫爾特)上的剩余合成副產品中凈化,并可以在步驟795處存儲。測序和解碼如圖8所示。
該結合的低聚糖樣本在步驟810處(利用設計為給予a/t與g/c相等處理26的熱循環儀環境的22個pcr周期)利用雙末端illuminapcr引物和高保真度accuprime試劑(試劑盒)被放大,taq聚合酶和火球菌聚合酶具有耐熱輔助蛋白。該放大的產品經磁珠凈化且經agilent2100生物分析儀定量,并在illuminahiseq2000上以雙末端模式利用ayb軟件測序,以產生104個堿基的讀數。
數字信息解碼執行如下:每個低聚糖的中央的91個堿基在步驟820處從兩端進行測序,由此很明顯要對全長(117個堿基)的低聚糖進行快速計算,并移除與設計不相符的序列讀數。該序列讀數在步驟830處利用精確的反向進行編碼過程的計算機軟件進行解碼。出于進一步考慮,經奇偶校驗的三進制數位表明有錯、在任何階段不能夠準確解碼、或被分配到重構的計算機文件的序列讀數在步驟840處被舍棄。
在多個不同的經測序的dna低聚糖上檢測到每個解碼文件的絕大多數位置,在步驟850處利用了簡單多數表決來解決因dna合成或測序錯誤引起的差異。程序860完成時,四或五個原始計算機文件210被精確重構。第五個文件需要人工干預來更正各有25個堿基的兩個區域,它們沒有從任何序列讀數中還原。
在步驟850的解碼過程中,注意到經計算機模擬重建的dna層次(在解碼之前,經由三進制至字節)的一個文件(最后確定是watsoncrick.pdf)包括兩個各有25個堿基的區域,它們沒有從任何一個序列讀數中還原。給定解碼的重疊片段結構,每個區域表明四個用于合成或測序的連貫片段均是無效的,因為四個連貫重疊片段中的任意一個均應含有與位置對應的堿基。對兩個區域的檢查表明,未檢測到的堿基屬于下一20堿基的基序的長距離重復。
5’gagcatctgcagatgctcat3’
注意到該基序的重復具有一個自反向互補的模式。其在圖4中示出。
長距離的、自反向互補的dna片段可能不易于利用illumina雙末端過程進行測序,因為dna片段可能形成內部非線性的莖環結構,將抑制本文件中約定使用的合成測序反應。結果,經計算機模擬的dna序列被修正以糾正該重復基序模式,而后經過隨后的解碼步驟。沒有碰到進一步的問題,且最終的解碼文件與文件watsoncrick.pdf精確匹配。將來可能用到確保在任一設計的dna片段中沒有長距離自互補區域存在的代碼。
霍夫曼代碼方案實例
表1示出了典型的霍夫曼代碼方案實例,用于將字節值(0-255)轉換為三進制。對于高度壓縮的信息,每字節值應以相等頻率出現,且每字節的三進制數位平均數將為(239*5+17*6)/256=5.07。每字節的三進制數位理論最大數值為log(256)/log(3)=5.05。
表1
文件的編碼
該任意計算機文件210表示為字節的串
現寫下len()作為計算串s1長度(以字符)的函數,并定義n=len(s1)。將n以三進制表示,并預設0s來產生三進制數位串s2,由此len(s2)=20。形成串的級聯s4=s1.s3.s2,,其中s3為最多選擇24個零的串,由此len(s4)為25的整數倍。
利用下表示出的方案,s4轉換為沒有重復核苷酸(nt)的dna字符{a,c,g,t}的串s5。s4的第一個三進制數位利用表的“a”行進行編碼。對每下一個三進制數位,從前一個字符轉換所定義的行中取字符。
表:確保無重復核苷酸的三進制至dna的編碼。
定義n=len(s5),并使2進制串的id識別原始文件,且它在給定的實驗中是單一的(在一次實驗中容許來自不同文件
每個dna片段fi進一步處理如下:
如果i為奇數,反向互補該dna片段fi。
使i3作為i的三進制表示,添加足夠的前導零,由此len(i3)=12。計算id中的奇數位的三進制數位和i3的和p,即id1+i31+i33+i35+i37+i39+i311。(p作為“奇偶性三進制數位”–類似于奇偶校驗位–來校驗有關id和i的編碼信息中的錯誤)。
形成標引信息250的串ix=id.i2.p(包含2+12+1=15個三進制數位)。利用上表示出的相同策略,將ix的dna-編碼的(步驟760)版本添加到fi,從由上一個字符fi定義的代碼表的行開始,給出標引片段f′i。
通過預設a或t并將c或g添加到f′i形成f″i–可以隨機選擇a或t以及c或g,但總沒有重復的核苷酸。這確保將已經在dna測序過程中反向逆轉(步驟240)的dna片段240從沒有逆轉的片段中區別出來。前者以g|c開始,以t|a結束;后者以t|a開始,以g|c結束。
片段f″i在步驟790處以實際dna低聚核苷酸合成,并在步驟790處存儲,且可提供給步驟820處的測序。
解碼
在步驟720處的解碼是簡單的反向編碼,從已測序的117個核苷酸長的dna片段240f″i開始。在dna測序程序中的(例如,在pcr反應中)反向互補可以通過觀察碎片是否以t|a開始、以g|c結束而被識別出來并用于隨后的反轉。隨著這兩個“方向”的核苷酸被移除,每個dna片段240剩下的115個核苷酸可以被拆分成在先的100個“消息”核苷酸和剩下的15個“標引信息250”核苷酸。標引信息核苷酸250可以解碼以確定文件標識符id、位置標引i3和i,且通過測試奇偶性三進制數位p可以檢測到錯誤。位置標引信息250容許構件dna編碼文件230,其可以利用以上編碼表的反向轉換為三進制,隨后利用給定的霍夫曼代碼轉換為原始字節。
就數據存儲的討論
dna存儲相比傳統的基于磁帶的或基于磁盤的存儲具有不同性能。在本實例中,~750kb的信息被合成了10pmol的dna,給出了大約1太字節/克的信息存儲密度。dna存儲不要求電源,且保守估計,可能保持(潛在地)活性幾千年。
dna存檔也可以通過將pcr應用到成對引物,隨后是等分(拆分)所得的dna的方案,從而以大規模并行的方式復制dna。在測序過程的該技術的實際證明中多次履行了該程序,但其也可以明確用于復制大規模信息,并在物理上將該信息發送到兩個或多個位置。在多個位置存儲該信息將為任何存檔方案提供提供進一步的穩定性,其本身可被用于設施之間的巨大規模的數據復制操作。
該實例中的解碼帶寬為3.4比特/秒,相比于磁盤(約太比特/秒)或磁帶(140兆比特/秒),且時延也較高(本實例中為~20天)。人們希望將來的測序技術能夠提高這幾項因數。
對使用本公開的dna存儲或磁帶存儲進行存檔的全部成本建模,表明主要的參數是在磁帶存儲技術和介質之間的轉移頻率和固定成本。圖3示出了dna存儲為有成本效益的時間量程。上方的粗體曲線表示了平衡時間(x軸),超過該點本公開中教導的dna存儲比磁帶更便宜。假設磁帶存檔每3年(f=1/3)需要讀取并重寫,并取決于dna存儲合成的相關成本和磁帶轉移的固定成本(y軸)。下方的粗體曲線對應每5年進行磁帶轉移。下方粗體曲線下面的區域表示當轉移比5年發生得更加頻繁時dna存儲是有成本效益的;在兩條粗體曲線之間,當轉移發生在3至5年時dna存儲是有成本效益的;且在上方粗體曲線以上,當轉移比3年發生得更不頻繁時磁帶更廉價。水平點劃線表明dna合成轉移到磁帶的相關成本的范圍是125-500(當前值)和12.5-50(若dna合成成本以數量級降低而得到)。垂直點劃線表明相應的平衡時間。注意所有軸線采用對數刻度。
長期數字存檔的一個主題是基于dna的存儲如何擴展到更大型的應用。需要編碼信息的合成dna的堿基數量正隨著需要存儲的信息量呈線性增長。必須考慮由短dna片段240重構全長文件時所需的標引信息。標引信息250僅以需要標引的dna片段240的數量的對數而增長。合成dna的總量需要以次線性增長。每個dna片段240需要標引的部分越來越多,然而,雖然期望未來可能出現長串合成是合理的,但該方案的行為建立在可用于數據和標引信息250的恒定的114個核苷酸的保守約束條件下。
隨著信息總量增加,編碼效率僅緩慢地下降(圖5)。在實驗中(兆字節規模),編碼方案具有88%的效率。圖5示出了當數據存儲達到拍字節(pb,1015字節)時效率仍然大于70%,且達到艾字節(eb,1018字節)規模時仍然大于65%,并且基于dna的存儲在比當前全球數據量大若干數量級時仍然可行。圖5也示出了當數據量以數量級增加時,成本(每單位存儲信息)僅緩慢增長。如果考慮到利用最新技術合成dna片段240的可用長度,該效率和成本規模更為有利。隨著存儲信息量的增加,解碼要求測序更多低聚糖。每字節的編碼信息具有固定的解碼支出,可能意味著每個堿基將被以較少次數讀取,因此更可能出現解碼錯誤。延展尺度分析來模擬降低測序覆蓋范圍對每堿基解碼錯誤率的影響,其顯示當編碼信息量增長達到并超過全球數據規模時,錯誤率增長非常緩慢。這也表明1308次的平均測序覆蓋范圍遠遠超過了對可靠解碼的需求。這被從79.6x3106個成對讀數中二次抽樣來模擬低覆蓋范圍的實驗所證實。
圖5表明,以因數10(或更多)減少覆蓋范圍會導致不變的解碼特征,其進一步說明了dna存儲方法的穩定性。基于dna存儲的應用已經在經濟上可行,用于長期存檔,具有低預期的廣泛接入性,諸如政府或歷史記錄。在科學界的一個實例是cern的castor系統,其存儲了總共80pb的大型強子對撞機數據并以15pbyr-1的速度增長。只有10%是在磁盤上維護的,且castor定期地在磁帶格式之間移動。為在潛在的未來中查實事件,需要對較舊數據進行存檔,但在收集的2-3年后訪問速率顯著降低。進一步的實例可以在天文、醫藥和行星際空間探測中找到。
圖5示出了當存儲信息量增加時的編碼效率和成本變化。x-軸(對數刻度)表示需要編碼的總信息量。示出了通常的數據規模,包括估計的三澤字節(3zb,3x1021字節)的全球數據。左側的y-軸刻度表示編碼效率,以可用與數據編碼的合成堿基的比例測定。右側的y-軸可讀表示對編碼成本的相應影響,在當前合成成本水平(實線)且在減少了兩個數量級的情況下(虛線)。
圖6示出了每還原堿基的錯誤率(y軸),作為測序覆蓋范圍的函數,表示為原始的79.6x106個成對讀數的采樣百分比(x軸;對數刻度)。一條曲線表示了無需人工干預還原的四個文件:當使用了≥2%的原始讀數時錯誤為零。通過蒙特卡洛模擬理論錯誤率模型,得到另外一條曲線。最終曲線表示了需要人工修正的文件(watsoncrick.pdf):可能的最小錯誤率為0.0036%。圍合的區域在插頁中放大示出。
除數據存儲之外,本公開的教導也可用于隱寫術。
參考文獻
1.bancroft,c.,bowler,t.,bloom,b.&clelland,c.t.long-termstorageofinformationindna.science293,1763–1765(2001)
2.cox,j.p.l.long-termdatastorageindna.trendsbiotech.19,247–250(2001)
3.baum,e.b.buildinganassociativememoryvastlylargerthanthebrain.science268,583–585(1995)
4.clelland,c.t.,risca,v.&bancroft,c.hidingmessagesindnamicrodots.nature399,533–534(1999)
5.kac,e.genesis(1999)http://www.ekac.org/geninfo.htmlaccessedonline,2april2012
6.wong,p.c.,wong,k.-k.&foote,h.organicdatamemory.usingthednaapproach.comm.acm46,95–98(2003)
7.ailenberg,m.&rotstein,o.d.animprovedhuffmancodingmethodforarchivingtext,images,andmusiccharactersindna.biotechniques47,747–754(2009)
8.gibson,d.g.etal.creationofabacterialcellcontrolledbyachemicallysynthesizedgenome.science329,52–56(2010)
9.mackay,d.j.c.informationtheory,inference,andlearningalgorithms.(cambridgeuniversitypress,2003)
10.watson,j.d.&crick,f.h.c.molecularstructureofnucleicacids.nature171,737–738(1953)
11.shapiro,b.etal.riseandfalloftheberingiansteppebison.science306,1561–1565(2004)
12.poinar,h.k.etal.metagenomicstopaleogenomics:large-scalesequencingofmammothdna.science311,392–394(2005)
13.willerslev,e.etal.ancientbiomoleculesfromdeepicecoresrevealaforestedsoutherngreenland.science317,111–114(2007)
14.green,r.e.etal.adraftsequenceoftheneanderthalgenome.science328,710–722(2010)
15.anchordoquy,t.j.&molina,m.c.preservationofdna.cellpreservationtech.5,180–188(2007)
16.bonnet,j.etal.chainandconformationstabilityofsolid-statedna:implicationsforroomtemperaturestorage.nucl.acidsres.38,1531–1546(2010)
17.lee,s.b.,crouse,c.a.&kline,m.c.optimizingstorageandhandlingofdnaextracts.forensicsci.rev.22,131–144(2010)
18.tsaftaris,s.a.&katsaggelos,a.k.ondesigningdnadatabasesforthestorageandretrievalofdigitalsignals.lecturenotescomp.sci.3611,1192–1201(2005)
19.yamamoto,m.,kashiwamura,s.,ohuchi,a.&furukawa,m.large-scalednamemorybasedonthenestedpcr.naturalcomputing7,335–346(2008)
20.kari,l.&mahalingam,k.dnacomputing:aresearchsnapshot.inatallah,m.j.&blanton,m.(eds.)algorithmsandtheoryofcomputationhandbook,vol.2.2nded.pp.31-1–31-24(chapman&hall,2009)
21.chen,p.m.,lee,e.k.,gibson,g.a.,katz,r.h.&patterson,d.a.raid:high-performance,reliablesecondarystorage.acmcomputingsurveys26,145–185(1994)
22.leproust,e.m.etal.synthesisofhigh-qualitylibrariesoflong(150mer)oligonucleotidesbyanoveldepurinationcontrolledprocess.nucl.acidsres.38,2522–2540(2010)
23.kosuri,s.etal.ascalablegenesynthesisplatformusinghigh-fidelitydnamicrochips.naturebiotech.28,1295–1299(2010)
24.beaucage,s.l.&caruthers,m.h.deoxynucleosidephosphoramidites—anewclassofkeyintermediatesfordeoxypolynucleotidesynthesis.tetrahedronlett.22,1859–1862(1981)
25.cleary,m.a.etal.productionofcomplexnucleicacidlibrariesusinghighlyparallelinsituoligonucleotidesynthesis.naturemethods1,241–248(2004)
26.aird,d.etal.analysingandminimizingpcramplificationbiasinilluminasequencinglibraries.genomebiol.12,r18(2011)
- 還沒有人留言評論。精彩留言會獲得點贊!