修飾植物基因組的方法和手段與流程
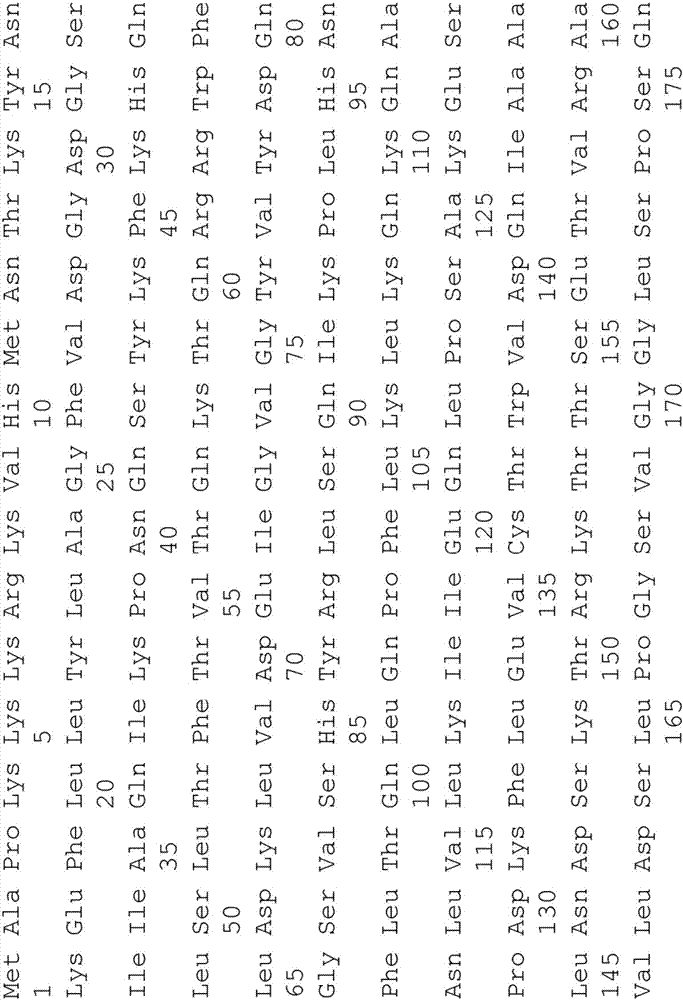
本申請是申請日為2012年8月14日、發明名稱為“修飾植物基因組的方法和手段”的中國專利申請201280049574.7(國際申請號pct/ep2012/065867)的分案申請。發明領域本發明涉及農學領域。更特別地,本發明提供使用定制設計的雙鏈dna斷裂誘導酶在植物基因組中精確定位的核苷酸序列上引入靶向修飾,包括插入、缺失或取代的方法和手段。本發明還涉及包含嵌合基因的棉花植物細胞、植物部分、植物或種子,所述嵌合基因包含編碼具有hppd活性的蛋白質的核酸序列,其中所述蛋白質在對應于seqidno:1的位置336的位置上具有色氨酸,其中所述蛋白質向所述植物提供對至少1x田間劑量的至少一種hppd抑制劑的耐受性;并且提供制備此類植物的方法。
背景技術:
:在植物基因組中引入靶向修飾的需求,例如以向植物提供農學上有用的性狀,如除草劑耐受性(包括控制在植物中外源dna整合的定位)已變得日益重要。為了滿足這一需求,已經開發了若干方法(綜述參見kumar(庫馬爾)和fladung(弗拉東),2001,trendsinplantscience(《植物科學趨勢》),6,pp155-159)。這些方法大部分依賴于經由雙鏈斷裂誘導(dsbi)酶的表達,在靶向位置初始引入的雙鏈dna斷裂。經由罕見切割內切核酸酶(例如i-scei),通過誘導雙鏈dna斷裂(dsb),激活靶基因座和/或修復或供體dna,已經示出出增加若干數量級的同源重組的頻率(puchta(普赫塔)等人,1996,proc.natl.acad.sci.u.s.a.(美國國家科學院院刊)),93,pp5055-5060;chilton(奇爾頓)和que(秋),plantphysiol.(《植物生理學》),2003;d’halluin(阿呂安)等人,2008plantbiotechnol.j.(《植物生物技術雜志》)6,93-102)。wo96/14408描述了一種編碼酶i-scei的分離dna。這種dna序列可以并入克隆和表達載體、轉化細胞系和轉基因動物中。在基因作圖和基因的定點插入中這些載體可以是有用的。wo00/46386描述了通過i-scei誘導的雙鏈斷裂,在細胞中修飾、修復、減弱和滅活一個基因或其他染色體dna的方法。還披露了在需要其的個體中治療或預防遺傳性疾病的方法。進一步披露了嵌合的限制性內切酶。wo2005/049842描述了使用罕見分裂“雙鏈斷裂”誘導(dsbi)酶連同改進的i-scei編碼核苷酸序列改進植物中靶向dna插入的方法和手段。wo2006/105946描述了用于通過同源重組在植物細胞和植物中將靶dna序列精確交換為感興趣的dna序列的方法,由此可以隨后去除在用于基因取代事件的臨時選擇的同源重組階段期間使用的可選擇的或可篩選的標記,而在去除步驟期間不遺留痕跡并且不依靠體外培養,采用其中描述的方法用于通過小孢子特異性表達的dsbi罕見分裂內切核酸酶去除選擇的dna。wo2008/037436描述了wo2006/105946方法和手段的變體,其中通過雙鏈斷裂誘導罕見分裂內切核酸酶誘導的選擇的dna片段的去除步驟處于種系特異啟動子的控制下。該方法的其他實施例依賴在修復dna的一端的非同源末端-連接和在另一端的同源重組。wo08/148559描述了wo2008/037436方法的變體,即在真核細胞中(例如植物細胞)中通過同源重組用于將靶dna序列精確交換為感興趣的dna序列的方法,由此可以隨后去除在用于基因取代事件的臨時選擇的同源重組階段期間使用的可選擇的或可篩選的標記,而不遺留痕跡,采用用于去除在直接重復中側翼是兩個核苷酸序列的選擇的dna的方法。wo2003/004659披露了重組系統以及用于從真核生物染色體dna去除核酸序列的方法。本發明還涉及轉基因生物(優選地植物),包含所述系統或由所述方法產生。wo2006/032426披露了改進的重組系統以及用于從植物基因組中消除標記序列的方法。具體地,本發明基于一種表達盒的用途,該表達盒包括歐芹泛素啟動子,以及可操作地連接到其上的編碼特異dna-核酸內切酶序列的核酸序列。美國臨時申請61/493579和ep11004570.5描述了使用雙鏈dna斷裂誘導酶和胚胎發生性愈傷組織以靶定方式修飾棉花植物的基因組的方法和手段。此外,已經描述了這樣的方法,其允許設計稀有切割內切核酸酶,以改變酶的底物或序列特異性,因此允許在目的基因座上誘導雙鏈斷裂,但不依賴于任何天然稀有切割內切核酸酶的識別位點的存在。簡言之,可以使用設計用于識別特異性核苷酸序列的鋅指結構域與來自天然限制性內切核酸酶,如foki的非特異性dna切割結構域之間的雜合體來制備嵌合的限制酶。此類方法已經描述于例如wo03/080809、wo94/18313或wo95/09233和isalan等,2001,naturebiotechnology19,656-660;liu等1997,proc.natl.acad.sci.usa94,5525-5530)中。通過從變體文庫中進行選擇來產生定制的大范圍核酸酶的另一方式描述于wo2004/067736中。還可以通過如wo2007/047859中描述的合理設計獲得具有改變的序列特異性和dna結合親和力的定制大范圍核酸酶或重新設計的大范圍核酸酶。wo2007/049095描述了“ladglidadg”歸巢內切核酸酶變體,其在兩個分開的亞結構域中具有突變,每一個亞結構域結合修飾的dna靶半位點的不同部分,從而內切核酸酶變體能夠切割包含每一個亞結構域結合的核苷酸的嵌合dna靶序列。wo2007/049156和wo2007/093836描述了具有新的切割特異性的i-crei歸巢內切核酸酶變體及其用途。wo2007/047859描述了合理設計的具有改變的序列特異性和dna結合親和力的大范圍內切核酸酶。wo11/064736描述了優化的內切核酸酶,以及使用優化的內切核酸酶靶向整合、靶向缺失或靶向突變多核苷酸的方法。wo11/064750描述了包含內切核酸酶和異源dna結合域(其包含一個或多個zn2c6鋅指)的嵌合內切核酸酶,以及使用嵌合內切核酸酶靶向整合、靶向缺失或靶向突變多核苷酸的方法,并且wo11/064751描述了包含內切核酸酶和異源dna結合域的嵌合內切核酸酶,以及使用嵌合內切核酸酶靶向整合、靶向缺失或靶向突變多核苷酸的方法。pct/ep11/002894和pct/ep11/002895描述了以靶定方式修飾包含嵌合基因的轉基因植物的植物基因組的方法和手段,其中所述嵌合基因具有通常用于植物分子生物學中的dna元件,以及重新設計的大范圍內切核酸酶,以切割通常用于植物分子生物學中的該元件。wo2009/006297描述了用于改變單子葉植物細胞和單子葉植物的基因組的方法和組合物,其涉及使用雙鏈斷裂誘導劑,以改變包含雙鏈斷裂誘導劑的識別序列的單子葉植物或植物細胞基因組序列。gao等2010,theplantjournal61,第176–187頁描述了使用重新設計的內切核酸酶在玉米中進行可遺傳定向誘變。然而,為了有效產生農藝學有用性狀的組合而不必借助于精細的育種方案或不必測試大量單一事件,因此仍然需要在功能上重新設計的大范圍核酸酶(其可以識別緊靠已存在的原種事件的識別位點),及其用途,以在單個遺傳基因座上產生賦予農藝學上有用的性質的大量基因。此類農藝學上有用的性狀之一是對除草劑,如hppd-抑制劑型除草劑的耐受性。hppd(羥苯丙酮酸二加氧酶)蛋白質是催化這樣的反應的酶,在所述反應中對羥基苯丙酮酸(本文中縮寫為hpp)(其為酪氨酸降解產物)被轉化成尿黑酸(本文中縮寫為hg),尿黑酸為植物中生育酚和質體醌的前體(crouchn.p.等(1997)tetrahedron,53,20,6993-7010,fritze等,(2004),plantphysiology134:1388-1400)。生育酚作為膜結合的抗氧化劑起作用。質體醌首先作為光系統ii(psii)與細胞色素b6/f復合體之間的電子載體起作用,其次是八氫番茄紅素去飽和酶(其參與類胡蘿卜素的生物合成)的氧化還原輔助因子。迄今為止,ncbi數據庫中存在的多種生物的多于700條核酸序列被注解為編碼具有hppd結構域的推測蛋白質。已經在現有技術中描述了若干hppd蛋白質及其一級序列,特別是細菌,如假單胞菌屬(pseudomonas)(rüetschi等,eur.j.biochem.,205,459-466,1992,wo96/38567)、植物,如擬南芥(wo96/38567,genebankaf047834)、胡蘿卜(wo96/38567,genebank87257)、燕麥(avenasativa)(wo02/046387)、小麥(wo02/046387)、寬葉臂形草(brachiariaplatyphylla)(wo02/046387)、蒺藜草(cenchrusechinatus)(wo02/046387)、硬直黑麥草(loliumrigidum)(wo02/046387)、葦葉羊茅(festucaarundinacea)(wo02/046387)、狗尾草(setariafaberi)(wo02/046387)、牛筋草(eleusineindica)(wo02/046387)、高粱(wo02/046387)、球孢子菌屬(coccicoides)(genebankcoitrp)、日本黃連(coptisjaponica)(wo06/132270)、萊茵衣藻(chlamydomonasreinhardtii)(es2275365)或哺乳動物,如小鼠或豬的hppd。對hppd的抑制導致光合作用的解偶聯,缺乏輔助捕光色素(accessorylight-harvestingpigments),以及最重要地,由于缺少正常情況下由類胡蘿卜素提供的光保護導致的uv照射和活性氧類別對葉綠體的破壞(褪色)(norris等(1995),plantcell7:2139-2149)。光合活躍的組織的褪色導致生長抑制和植物死亡。已經證明抑制hppd并且特異性結合酶以抑制hpp轉化成尿黑酸的一些分子是非常有效的選擇性除草劑。目前,大多數市售hppd抑制劑型除草劑屬于這三個化學家族之一:1)三酮類,例如磺草酮(sulcotrione)、甲基磺草酮(mesotrione);環磺酮(tembotrione);tefuryltrione;bicyclopyrone;雙環磺草酮(benzobicyclon)2)異唑類,例如異氟草(isoxaflutole),或對應的二酮腈類化合物。在植物中,異唑類,如異氟草被快速轉化成二酮腈類化合物,其顯示hppd抑制劑性質;和3)pyrazolinones,例如,苯吡唑草酮(topramezone)、磺酰草吡唑(pyrasulfotole)和芐草唑(pyrazoxyfen)。這些抑制hppd的除草劑可在展現出代謝耐受性的作物植物,如玉米(zeamays)(它們在其中被迅速降解)中使用以抵抗草(grass)和/或闊葉雜草(schulz等,1993;mitchell等,2001;garcia等,2000;pallett等,2001)。為拓展這些抑制hppd的除草劑的范圍,已經進行了若干努力,以賦予植物,特別是沒有代謝耐受性或代謝耐受性較差的植物在農藝學田間條件下可接受的耐受性水平。在該上下文中,已經首次證明轉化的敏感植物中僅天然hppd酶的過表達就為轉化的植物提供對hppd抑制劑的有效耐受性(wo96/38567)。另一種策略是突變hppd,以獲得下述靶酶,其保留其催化hpp轉化為尿黑酸的性質,同時對hppd抑制劑不如突變前的天然hppd敏感。該策略已經通過利用編碼在其c末端部分的一個或多個位置上突變的hppd酶的基因轉化植物而成功應用于產生對hppd抑制劑耐受的植物(wo99/24585)。在可以賦予對hppd抑制劑耐受性的hppd酶的c末端部分的有用突變中,顯示某些突變提供對某些二酮腈類除草劑的增加的耐受性,例如突變pro215leu、gly336glu、gly336ile和gly336trp(突變氨基酸的位置參照假單胞菌屬hppd而被指出)。更近來,在專利申請wo2009/144079中已經顯示,hppd的位置336處的某些特定氨基酸取代在體外提供對某些hppd抑制劑型除草劑的耐受性。us2010/0197503還指出了來自燕麥的hppd活性位點內或附近的不同位置上的許多突變,并檢查了某些hppd抑制劑,如磺草酮對一些這些突變的hppd酶的抑制。盡管開發針對上文所述的一些hppd抑制劑型除草劑顯示出耐受性的植物獲得了這些成功,但仍期望開發和/或改良特定植物,如棉花對較多的、較新的或對若干不同的hppd抑制劑的耐受性,特別是對屬于三酮類(例如磺草酮、甲基磺草酮、環磺酮、莊無忌、bicyclopyrone和雙環磺草酮)、pyrazolinones(例如,苯吡唑草酮、磺酰草吡唑和pyrazoxifen)和異唑類(例如異氟草)或對應的二酮腈類化合物的類別的hppd抑制劑的耐受性。如本文在不同實施方案、實施例和權利要求中描述后解決了該問題。如下文所述在本發明不同的詳細實施方案以及權利要求中解決了這些及其他問題。發明概述在一個實施方案中,本發明涉及在預定位點修飾植物細胞的基因組的方法,其包括以下步驟:在所述預定位點附近或在所述預定位點上誘導雙鏈dna斷裂,所述雙鏈斷裂由向所述細胞中引入識別所述預定位點附近或所述預定位點上的識別序列的雙鏈dna斷裂誘導(dsbi)酶來誘導;選擇植物細胞,其中已經修復了所述雙鏈dna斷裂,導致在所述預選位點處對基因組的修飾,其中所述修飾選自i.至少一個核苷酸的替換;ii.至少一個核苷酸的缺失;iii.至少一個核苷酸的插入;或iv.i.–iii.的任何組合;其特征在于所述預定位點和/或識別位點緊靠原種事件。在特定實施方案中,事件是ghb119。在另一個實施方案中,識別序列包含在seqidno:3或seqidno:4中。所述識別序列可以包含seqidno:1或seqidno:2的核苷酸序列。本發明還提供在預定位點修飾植物細胞的基因組的方法,其包括以下步驟:在所述預定位點附近或在所述預定位點上誘導雙鏈dna斷裂,所述雙鏈斷裂由向所述細胞中引入識別所述預定位點附近或所述預定位點上的識別序列的雙鏈dna斷裂誘導(dsbi)酶來誘導;選擇植物細胞,其中已經修復了所述雙鏈dna斷裂,導致在所述預選位點處對基因組的修飾,其中所述修飾選自至少一個核苷酸的替換;至少一個核苷酸的缺失;至少一個核苷酸的插入;或i.–iii.的任何組合;其特征在于所述識別位點包含seqidno.1或seqidno.2的核苷酸序列。可以通過向所述細胞中遞送包含編碼(一起)所述內切核酸酶的一個或多個嵌合基因的核酸分子來向所述細胞中引入dsbi酶。所述dsbi酶可以是單鏈大范圍核酸酶或一對大范圍核酸酶,其識別或一致識別所述預定位點并誘導所述雙鏈斷裂。在特定實施方案中,所述大范圍核酸酶或大范圍核酸酶對來自i-crei,并且其中以下氨基酸存在于大范圍核酸酶單元1中:a.位置32處的s;b.位置33處的y;c.位置38處的q;d.位置80處的q;e.位置40處的s;f.位置42處的t;g.位置77處的r;h.位置68處的y;i.位置70處的q;j.位置75處的h;k.位置44處的t;l.位置24處的i;m.位置26處的q;n.位置28處的k;o.位置30處的n。并且其中以下氨基酸存在于大范圍核酸酶單元2中:p.位置70處的s;q.位置44處的q;r.位置24處的k;s.位置26處的a;t.位置28處的k;u.位置30處的n;v.位置32處的s;w.位置33處的y;x.位置38處的q;y.位置80處的q;z.位置40處的s;aa.位置42處的t;bb.位置77處的q;cc.位置68處的y。所述大范圍核酸酶或大范圍核酸酶對可以包含seqidno.6從氨基酸位置11-165和位置204-360的氨基酸序列。所述大范圍核酸酶或大范圍核酸酶對還可以由包含或一起包含seqidno5的從核苷酸位置3120-3584和位置3698-4169的核苷酸序列的一條或多條核苷酸序列編碼。在一個實施方案中,在步驟b之前,向所述細胞中遞送修復dna分子,所述修復dna分子用作修復所述雙鏈dna斷裂的模板。所述修復dna可以包含至少一個側翼區,其包含與所述預定位點上游或下游dna區具有充分同源性的核苷酸序列,以允許與所述上游或下游dna區重組。或者,所述修復dna可以包含位于所述修復dna相反端的兩個側翼區,所述側翼區之一包含與所述預定位點的上游dna區具有充分同源性的核苷酸序列,另一個側翼區包含與所述預定位點的下游序列具有充分同源性的核苷酸序列,以允許所述側翼核苷酸序列與所述上游和下游dna區進行重組。在其他實施方案中,所述修復dna包含可選擇標志物基因和/或植物可表達的目的基因。所述植物可表達的目的基因可以選自除草劑耐受基因、昆蟲抗性基因、疾病抗性基因、非生物脅迫抗性基因、參與油類生物合成、碳水化合物生物合成的酶、參與纖維素強度或纖維素長度的酶、參與次生代謝物生物合成的酶。在仍另一實施方案中,將在預定位置上基因組被修飾的植物細胞進一步再生成植物,其在所述預定位置上也含有所述修飾。然后所述植物可以與另一植物進行雜交,產生也含有基因組修飾的后代。本發明還涉及通過上文所述的方法獲得的在基因組的預定位點上包含修飾的植物細胞。本發明還包括:植物、植物部分、種子或其繁殖材料,在基因組的預定位點上包含修飾,通過本發明的方法獲得或基本上由本發明的植物細胞組成。還提供培養本發明的植物,即在基因組的預定位點上包含修飾的植物的方法,其包括向所述植物或其中生長所述植物的基質應用化學物質的步驟,以及包括用于產生在基因組的預定位點上包含修飾的植物的方法,其包括使基本上由本發明的植物細胞組成的植物或本發明的植物(包含預期基因組修飾的植物細胞和植物)與另一植物或其本身雜交并任選地收獲種子的方法。在一個實施方案中,本發明還提供包含嵌合基因的棉花植物細胞、植物部分、植物或種子,所述嵌合基因包含(a)編碼具有hppd活性的蛋白質的核酸序列,其中所述蛋白質在對應于seqidno:19的位置336的位置上具有色氨酸,其中所述蛋白質向所述植物提供對至少1x田間劑量的至少一種hppd抑制劑的耐受性,所述核酸序列有效連接(b)植物可表達的啟動子和任選地(c)翻譯終止和多腺苷酸化區。具有hppd活性的蛋白質可以與seqidno:21的氨基酸序列具有至少95%的序列同一性。可以優化編碼具有hppd活性的蛋白質的核酸序列用于在棉花中表達。所述蛋白質可以包含seqidno:21的氨基酸序列或可以由包含seqidno:21的從nt949-nt2025的核苷酸序列的核酸編碼。所述棉花植物細胞、植物部分、植物或種子具有對至少1.5x田間劑量的所述至少一種hppd抑制劑,或至少2x田間劑量的所述至少一種hppd抑制劑,或至少4x田間劑量的所述至少一種hppd抑制劑的耐受性。所述至少一種hppd抑制劑可以選自甲基磺草酮、異氟草、苯吡唑草酮、pyrasulfutole和環磺酮。本發明的棉花植物細胞、植物部分、植物或種子可以對至少兩種hppd抑制劑,優選至少三種hppd抑制劑,更優選至少四種hppd抑制劑,如至少5種或至少6種hppd抑制劑耐受。本發明的棉花植物細胞、植物部分、植物或種子的嵌合基因可以包含seqidno:20從位置88-位置2714的核酸序列。本發明的棉花植物細胞、植物部分、植物或種子還可以包含至少一個其他嵌合基因,其包含編碼這樣的酶的核酸序列,所述酶向植物提供對非hppd抑制劑的除草劑的耐受性或提供對至少一種昆蟲或真菌種的耐受性。在另一實施方案中,本發明提供用于獲得對至少1x田間劑量的至少一種hppd抑制劑耐受的棉花植物或植物細胞的方法,其包括-引入嵌合基因,其包含:(a)編碼具有hppd活性的蛋白質的核酸序列,其中所述蛋白質在對應于seqidno:19的位置336的位置上具有色氨酸,其中所述蛋白質向所述植物提供對至少1x田間劑量的至少一種hppd抑制劑的耐受性,所述核酸序列有效連接(b)植物可表達的啟動子和任選地(c)翻譯終止和多腺苷酸化區。還提供用于控制棉花植物附近或植物田地中雜草的方法,其包括-向棉花植物附近或棉花植物田地應用至少1x田間劑量的至少一種hppd抑制劑。在本發明的方法中,所述至少一種hppd抑制劑可以選自甲基磺草酮、異氟草、苯吡唑草酮、pyrasulfutole和環磺酮。以至少1.5x、至少2x或至少4x的田間劑量應用所述至少一種hppd抑制劑。在本發明方法的特定實施方案中,所述至少一種hppd抑制劑是異氟草和甲基磺草酮,其中所述異氟草在出苗前應用,而所述甲基磺草酮在出苗后應用。附圖簡述圖1:識別位點以及與不同的大范圍核酸酶單體單元cot5和cot6的氨基酸的相互作用的圖示。圖2:單鏈cot-5/6大范圍核酸酶的氨基酸序列,其包含sv40核定位信號(氨基酸1-10)、cot-5亞基(氨基酸11-165)、接頭序列(氨基酸166-203)和cot-6亞基(氨基酸204-360)。圖3:通過至少一側同源重組靶向插入的圖表概述。剪刀表示dsbi酶(cot-5/6)的識別位點,方箭頭(blockarrow)代表啟動子,小的矩形代表終止子,而大的矩形代表基因,小箭頭代表pcr引物,雙頭箭代表dna印跡片段,垂直方箭頭代表限制酶切位點,虛線代表側翼dna序列,其中同源區由連譜號表示。修復dna載體pcv211包含2mepsps基因和hppd-336w,其側翼為與ghb119原種事件3’側翼序列中的cot-5/6識別位點周圍區域具有同源性的側翼區。cot-5/6誘導的雙鏈dna斷裂后,pcv211載體的1561bp和2072bp側翼區與ghb119的3’側翼序列的對應區域之間同源重組,pcv211載體的2mepsps基因和hppd-336w基因被插入到靶位點上。可以使用引物對ib617xib572和ib616xib572通過pcr并利用針對epsps、hppd或cry2ae的探針在draiii/stui消化的基因組dna上通過dna印跡鑒定11kb基因組帶來鑒定正確疊加的事件。本發明不同實施方案的描述本發明基于這樣的觀察,即可以獲得在功能上重新設計的大范圍核酸酶,其特異性識別并切割核苷酸序列(seqidno.1和seqidno.2–圖1),所述核苷酸序列緊靠現有事件存在,即棉花事件ghb119(描述于2008/151780,保藏號atccpta-8398),其包含cry2ae基因和bar基因(分別賦予鱗翅目抗性和草銨膦(glufosinate)耐受性)。使用該特異的大范圍核酸酶,可以將包含epsps基因和hppd基因(分別賦予對草甘膦(glyphosate)和hppd抑制型除草劑的耐受性)的額外dna片段插入到現有ghb119事件的少數kb內,由此產生應該作為單個遺傳單位遺傳的四倍基因疊加。因此產生了棉花植物,其包含編碼具有hppd活性的蛋白質的嵌合dna分子,其中對應于熒光假單胞菌(pseudomonasfluorescens)hppd蛋白質位置336的位置上的甘氨酸(gly或g)的保守氨基酸已經被色氨酸(trp或w)所替換。此外驚奇地發現,植物,特別是棉花植物顯示對至少1x田間劑量的若干hppd抑制劑型除草劑,如甲基磺草酮、異氟草、環磺酮、pyrasulfutole和苯吡唑草酮或如本文列出的任何其他可應用hppd抑制劑的耐受性,所述植物包含導致這樣的蛋白質表達的嵌合基因,所述蛋白質具有hppd活性,在對應于熒光假單胞菌的蛋白質氨基酸序列的位置336的位置上的保守天然氨基酸殘基gly突變為trp,無論是四倍疊加(即靶定的)或是通過隨機轉化產生的。令人驚奇的是,本發明人發現,表達具有hppd活性,在對應于熒光假單胞菌的蛋白質氨基酸序列的位置336的位置上的保守天然氨基酸殘基gly突變為trp的蛋白質的植物,特別是棉花植物顯示對至少1x田間劑量的若干hppd抑制劑的耐受性。能夠緊靠現有原種事件引入基因組修飾具有若干優勢。首先,修飾將與較早事件共分離,由此避免組合單一事件賦予的某些性狀通常需要的復雜育種方案。其次,修飾將在有利的基因組環境中發生,用于表達起因于修飾的想要的性狀,因為也假定現有的原種事件由于其特定基因組定位而顯示正確的、適當的和穩定的空間和時間表型表達,如下文詳細說明。因此,在一個實施方案中,本發明涉及在預定位點修飾植物細胞的基因組的方法,其包括以下步驟:在所述預定位點附近或在所述預定位點上誘導雙鏈dna斷裂,所述雙鏈斷裂由向所述細胞中引入識別所述預定位點附近或所述預定位點上的識別序列的雙鏈dna斷裂誘導(dsbi)酶來誘導;選擇植物細胞,其中已經修復了所述雙鏈dna斷裂,導致在所述預選位點處對基因組的修飾,其中所述修飾選自i.至少一個核苷酸的替換;ii.至少一個核苷酸的缺失;iii.至少一個核苷酸的插入;或iv.i.–iii.的任何組合;其特征在于所述預定位點和/或所述識別序列緊靠現有的原種事件。如本文所用,“雙鏈dna斷裂誘導的稀有切割內切核酸酶”是能夠在特定核苷酸序列(其稱為“識別位點”)上誘導雙鏈dna斷裂的酶。稀有切割內切核酸酶在如下意義上是稀有切割的,即由于其長的識別序列(通常約14-40nt),它們甚至在更大的植物基因組中具有非常低的切割頻率,例如,每個基因組它們僅切割10次、僅5次、僅4次、僅3次、僅2次、或僅1次。歸巢內切核酸酶由此類稀有切割內切核酸酶的家族組成,有時候也稱為大范圍核酸酶。它們由內含子編碼,不依賴于基因或間插序列,并且呈現明顯的結構和功能性質,將其區別于一般來自細菌限制性修飾的類型ii系統的更經典的限制酶。它們的識別位點具有一般的不對稱性,其與大多數限制酶識別位點的特征性二重對稱性不同。已經顯示由內含子編碼的若干歸巢內切核酸酶或內含肽促進其各自遺傳元件歸巢到等位的無內含子或無內含肽位點中。通過在無內含子或無內含肽等位基因中產生位點特異性的雙鏈斷裂,這些核酸酶產生重組基因端,其參與基因轉換過程,該過程復制編碼序列并導致dna水平上插入內含子或間插序列。一種良好表征的標記的尋靶內切核酸酶是i-scei。i-scei是一種位點特異性內切核酸酶,是造成釀酒酵母中位于線粒體中內含子移動的原因。該酶是由21srrna基因可任選的內含子sclsu.1編碼的并且在產生具有3’oh突出端的4bp交錯切口的內含子插入位點引發雙鏈dna斷裂。i-scei內切核酸酶的識別位點擴大到18bp的非對稱序列(colleaux(科萊阿克斯)等人1988proc.natl.acad.sci.usa(《美國國家科學院院刊》85:6022-6026)。i-scei的氨基酸序列以及線粒體i-scei基因的通用密碼等效物已經由例如wo96/14408提供。wo96/14408進一步披露了多個i-scei蛋白突變體,這些突變體仍然是功能性的。pct申請pct/ep04/013122(并入本文作為參考)提供i-scei的合成的核苷酸序列變體,其已經被優化用于在植物中表達。其他稀有切割dsb誘導酶及其各自識別位點的列表提供在wo03/004659的表i中(第17-20頁)(并入本文作為參考)。這些包括i-scei、i-chui、i-dmoi、i-crei、i-csmi、pi-flii、pt-mtui、i-ceui、i-sceii、i-sceiii、ho、pi-civi、pi-ctri、pi-aaei、pi-bsui、pi-dhai、pi-drai、pi-mavi、pi-mchi、pi-mfui、pi-mfli、pi-mgai、pi-mgoi、pi-mini、pi-mkai、pi-mlei、pi-mmai、pi-mshi、pi-msmi、pi-mthi、pi-mtui、pi-mxei、pi-npui、pi-pfui、pi-rmai、pi-spbi、pi-sspi、pi-faci、pi-mjai、pi-phoi、pi-tagi、pi-thyi、pi-tkoi或pi-tspi。此外,用于設計識別基本上任何所選靶核苷酸序列的定制稀有切割內切核酸酶的方法是可用的。簡言之,可以使用設計用于識別特異性核苷酸序列的鋅指結構域和來自天然限制酶,如foki的非特異性dna切割結構域之間的雜合體來制備嵌合的限制酶。這些酶一般被稱為鋅指內切核酸酶(zfes)。此類方法已經描述于例如wo03/080809、wo94/18313或wo95/09233和isalan等,2001,naturebiotechnology19,656-660;liu等1997,proc.natl.acad.sci.usa94,5525-5530)中。通過從變體文庫中進行選擇來產生定制的大范圍核酸酶的另一方式描述于wo2004/067736中。還可以通過如wo2007/047859中描述的合理設計獲得具有改變的序列特異性和dna結合親和力的定制大范圍核酸酶。定制設計的稀有切割內切核酸酶的另一實例包括所謂的tale核酸酶,其基于來自細菌屬黃單胞菌屬(xanthomonas)的轉錄激活因子樣效應子(tales),其與例如foki的催化結構域融合。這些tales的dna結合特異性定義為串聯排列的34/35氨基酸重復單元的重復可變雙殘基(rvds),其可以被修飾,以識別特異的靶序列(christian等,2010,genetics186:757–761、wo11/072246、wo10/079430和wo11/146121。此類定制設計的內切核酸酶還被稱為非天然存在的內切核酸酶。因為重新設計的大范圍核酸酶來自天然存在的內切核酸酶,因此可用的潛在識別位點不是完全隨機的,而是看起來與作為重新設計的大范圍核酸酶基礎的天然存在的內切核酸酶最初識別的核苷酸序列具有一定程度的相似性。gao等(2010,theplantjournal,第1-11頁)還說明了,修飾i-crei的dna結合特征的基于結構的蛋白質設計方法是基于對i-crei-dna共晶體結構的視覺觀察,其導致對大量氨基酸取代的預測,所述氨基酸取代改變其識別位點的特定位置上的i-crei堿基偏好性。通過實驗評價單個氨基酸取代,并且向可以“混合和匹配”的突變數據庫中添加賦予堿基偏好想要的改變的那些氨基酸取代,以產生識別高度不同的dna位點的i-crei的衍生物。理論上,使用目前突變數據庫可以獲得的組合多樣性足以在隨機dna序列中大約每1000bp靶向改造的內切核酸酶。如本文所用,“事件”定義為(人造)遺傳基因座,由于遺傳改造,其在特定基因組位置上攜帶包含至少一個拷貝的目的基因或多個目的基因的外源dna或轉基因。事件的典型等位狀態是存在或缺失外源dna。事件的表型特征在于轉基因的表達。在遺傳水平上,事件是植物遺傳組成的一部分。在分子水平上,事件的特征在于轉基因的限制性圖譜(例如通過southern印跡確定)、轉基因的上游和/或下游側翼序列(反映了基因組位置)、分子標志物的位置和/或轉基因的分子構型。一般地,利用包含至少一個目的基因的轉化dna轉化植物導致包含多個單獨事件(其每一個是獨特的)的轉化體群體。事件的特征在于外源dna和至少一個側翼序列。如本文所用,原種事件為基于轉基因的表達和穩定性及其賦予的性狀,以及其與包含它的植物的最佳農藝學特性的相容性從一組事件選擇的事件,其通過用同一轉化dna轉化而獲得。因此原種事件選擇的標準為一種或多種,優選兩種或多種,有利地為所有下列的標準:a)外源dna的存在不損害植物的其他所期望的特性,如與農藝學性能或商業價值相關的那些特性(例如不引起對疾病的增加的易感性,不引起產量下降,或不引起增加的倒伏等);b)事件的特征在于充分定義的穩定遺傳的分子構型,且為其可開發用于鑒定對照的適當工具;c)在事件的雜合(或半合子)和純合狀態下,目的基因在一系列的環境條件下以商業可接受水平顯示正確、適當且穩定的空間和時間表型表達,在所述環境條件中攜帶所述事件的植物可能處于正常的農藝學用途。還優選外源dna與植物基因組的位置有關,其中所述位置允許易于漸滲想要的商業遺傳背景。事件作為原種事件的狀態通過將原種事件漸滲不同的相關遺傳背景并且觀察對例如上述a)、b)和c)中的一個、兩個或所有標準的符合而證實。因此“原種事件”指包含外源dna的遺傳基因座,其滿足上述標準。植物、植物材料或后代如種子可在其基因組中包含一個或多個原種事件。一旦已對外源dna的一個或兩個側翼序列進行了測序,可用分子生物學技術開發特異性識別樣品核酸(dna或rna)中這種(這些)序列的引物和探針。例如可開發pcr方法來鑒定生物樣品(如植物、植物材料或包含植物材料的產品的樣品)中的原種事件。此類pcr基于至少兩條特異“引物”,一條識別原種事件5’或3’側翼序列中的序列,而另一條識別位于外源dna中的序列。所述引物優選具有15-35個核苷酸的序列,其在優化的pcr條件下分別“特異性識別”原種事件5’或3’側翼序列中的序列和原種事件的外源dna中的序列,以便從包含原種事件的核酸樣品中擴增出特異片段(“整合片段”或區別性擴增子)。這意味著在優化的pcr條件下植物基因組或外源dna中只有靶向整合片段而沒有其他序列被擴增。可根據本發明方法使用的含有原種轉化事件或轉化事件組合的轉基因植物包括例如在多個國家或地區調控管理機構的數據庫中列出的那些,并包括事件1143-14a(棉花,昆蟲控制,未保藏,描述于wo2006/128569);事件1143-51b(棉花,昆蟲控制,未保藏,描述于wo2006/128570);事件1445(棉花,除草劑耐受性,未保藏,描述于us2002120964或wo2002/034946);事件17053(稻,除草劑耐受性,保藏為pta-9843,描述于wo2010/117737);事件17314(稻,除草劑耐受性,保藏為pta-9844,描述于wo2010/117735);事件281-24-236(棉花,昆蟲控制-除草劑耐受性,保藏為pta-6233,描述于wo2005/103266或us2005216969);事件3006-210-23(棉花,昆蟲控制-除草劑耐受性,保藏為pta-6233,描述于us2007143876或wo2005/103266);事件3272(玉米,質量性狀,保藏為pta-9972,描述于wo2006098952或us2006230473);事件40416(玉米,昆蟲控制-除草劑耐受性,保藏為atccpta-11508,描述于wo2011/075593);事件43a47(玉米,昆蟲控制-除草劑耐受性,保藏為atccpta-11509,描述于wo2011/075595);事件5307(玉米,昆蟲控制,保藏為atccpta-9561,描述于wo2010/077816);事件asr-368(糠穗草,除草劑耐受性,保藏為atccpta-4816,描述于us2006162007或wo2004053062);事件b16(玉米,除草劑耐受性,未保藏,描述于us2003126634);事件bps-cv127-9(大豆,除草劑耐受性,保藏為ncimbno.41603,描述于wo2010/080829);事件ce43-67b(棉花,昆蟲控制,保藏為dsmacc2724,描述于us2009217423或wo2006/128573);事件ce44-69d(棉花,昆蟲控制,未保藏,描述于us20100024077);事件ce44-69d(棉花,昆蟲控制,未保藏,描述于wo2006/128571);事件ce46-02a(棉花,昆蟲控制,未保藏,描述于wo2006/128572);事件cot102(棉花,昆蟲控制,未保藏,描述于us2006130175或wo2004039986);事件cot202(棉花,昆蟲控制,未保藏,描述于us2007067868或wo2005054479);事件cot203(棉花,昆蟲控制,未保藏,描述于wo2005/054480);事件das40278(玉米,除草劑耐受性,保藏為atccpta-10244,描述于wo2011/022469);事件das-59122-7(玉米,昆蟲控制-除草劑耐受性,保藏為atccpta11384,描述于us2006070139);事件das-59132(玉米,昆蟲控制-除草劑耐受性,未保藏,描述于wo2009/100188);事件das68416(大豆,除草劑耐受性,保藏為atccpta-10442,描述于wo2011/066384或wo2011/066360);事件dp-098140-6(玉米,除草劑耐受性,保藏為atccpta-8296,描述于us2009137395或wo2008/112019);事件dp-305423-1(大豆,質量性狀,未保藏,描述于us2008312082或wo2008/054747);事件dp-32138-1(玉米,雜交系統,保藏為atccpta-9158,描述于us20090210970或wo2009/103049);事件dp-356043-5(大豆,除草劑耐受性,保藏為atccpta-8287,描述于us20100184079或wo2008/002872);事件ee-1(茄子,昆蟲控制,未保藏,描述于wo2007/091277);事件fi117(玉米,除草劑耐受性,保藏為atcc209031,描述于us2006059581或wo1998/044140);事件ga21(玉米,除草劑耐受性,保藏為atcc209033,描述于us2005086719或wo1998/044140);事件gg25(玉米,除草劑耐受性,保藏為atcc209032,描述于us2005188434或wo1998/044140);事件ghb119(棉花,昆蟲控制-除草劑耐受性,保藏為atccpta-8398,描述于wo2008/151780);事件ghb614(棉花,除草劑耐受性,保藏為atccpta-6878,描述于us2010050282或wo2007/017186);事件gj11(玉米,除草劑耐受性,保藏為atcc209030,描述于us2005188434或wo1998/044140);事件gmrz13(甜菜,病毒抗性,保藏為ncimb-41601,描述于wo2010/076212);事件h7-1(甜菜,除草劑耐受性,保藏為ncimb41158或ncimb41159,描述于us2004172669或wo2004/074492);事件joplin1(小麥,疾病耐受性,未保藏,描述于us2008064032);事件ll27(大豆,除草劑耐受性,保藏為ncimb41658,描述于wo2006/108674或us2008320616);事件ll55(大豆,除草劑耐受性,保藏為ncimb41660,描述于wo2006/108675或us2008196127);事件llcotton25(棉花,除草劑耐受性,保藏為atccpta-3343,描述于wo2003013224或us2003097687);事件llrice06(稻,除草劑耐受性,保藏為atcc-23352,描述于us6468747或wo2000/026345);事件llrice601(稻,除草劑耐受性,保藏為atccpta-2600,描述于us20082289060或wo2000/026356);事件ly038(玉米,質量性狀,保藏為atccpta-5623,描述于us2007028322或wo2005061720);事件mir162(玉米,昆蟲控制,保藏為pta-8166,描述于us2009300784或wo2007/142840);事件mir604(玉米,昆蟲控制,未保藏,描述于us2008167456或wo2005103301);事件mon15985(棉花,昆蟲控制,保藏為atccpta-2516,描述于us2004-250317或wo2002/100163);事件mon810(玉米,昆蟲控制,未保藏,描述于us2002102582);事件mon863(玉米,昆蟲控制,保藏為atccpta-2605,描述于wo2004/011601或us2006095986);事件mon87427(玉米,授粉控制,保藏為atccpta-7899,描述于wo2011/062904);事件mon87460(玉米,脅迫耐受性,保藏為atccpta-8910,描述于wo2009/111263或us20110138504);事件mon87701(大豆,昆蟲控制,保藏為atccpta-8194,描述于us2009130071或wo2009/064652);事件mon87705(大豆,質量性狀-除草劑耐受性,保藏為atccpta-9241,描述于us20100080887或wo2010/037016);事件mon87708(大豆,除草劑耐受性,保藏為atccpta9670,描述于wo2011/034704);事件mon87754(大豆,質量性狀,保藏為atccpta-9385,描述于wo2010/024976);事件mon87769(大豆,質量性狀,保藏為atccpta-8911,描述于us20110067141或wo2009/102873);事件mon88017(玉米,昆蟲控制-除草劑耐受性,保藏為atccpta-5582,描述于us2008028482或wo2005/059103);事件mon88913(棉花,除草劑耐受性,保藏為atccpta-4854,描述于wo2004/072235或us2006059590);事件mon89034(玉米,昆蟲控制,保藏為atccpta-7455,描述于wo2007/140256或us2008260932);事件mon89788(大豆,除草劑耐受性,保藏為atccpta-6708,描述于us2006282915或wo2006/130436);事件ms11(油菜,授粉控制-除草劑耐受性,保藏為atccpta-850或pta-2485,描述于wo2001/031042);事件ms8(油菜,授粉控制-除草劑耐受性,保藏為atccpta-730,描述于wo2001/041558或us2003188347);事件nk603(玉米,除草劑耐受性,保藏為atccpta-2478,描述于us2007-292854);事件pe-7(稻,昆蟲控制,未保藏,描述于wo2008/114282);事件rf3(油菜,授粉控制-除草劑耐受性,保藏為atccpta-730,描述于wo2001/041558或us2003188347);事件rt73(油菜,除草劑耐受性,未保藏,描述于wo2002/036831或us2008070260);事件t227-1(甜菜,除草劑耐受性,未保藏,描述于wo2002/44407或us2009265817);事件t25(玉米,除草劑耐受性,未保藏,描述于us2001029014或wo2001/051654);事件t304-40(棉花,昆蟲控制-除草劑耐受性,保藏為atccpta-8171,描述于us2010077501或wo2008/122406);事件t342-142(棉花,昆蟲控制,未保藏,描述于wo2006/128568);事件tc1507(玉米,昆蟲控制-除草劑耐受性,未保藏,描述于us2005039226或wo2004/099447);事件vip1034(玉米,昆蟲控制-除草劑耐受性,保藏為atccpta-3925.,描述于wo2003/052073),事件32316(玉米,昆蟲控制-除草劑耐受性,保藏為pta-11507,描述于wo2011/084632)和事件4114(玉米,昆蟲控制-除草劑耐受性,保藏為pta-11506,描述于wo2011/084621)。如本文所用,“緊靠”指預定位點定位在距現有轉基因事件這樣的距離,以便在預定位點附近或預定位點上引入的修飾將與現有事件遺傳連鎖,即它們在至少99%的情況中作為單個遺傳單位進行遺傳。遺傳連鎖一般以厘摩(縮寫為cm)表示。厘摩是用于測定遺傳連鎖的重組頻率的單位,定義為100個減數分裂產物中一個是重組體的基因之間的距離,或者換言之,厘摩等于染色體上一個遺傳基因座上的標志物由于在一代中交換將與第二個基因座上的標志物分離的1%可能性。其經常用于推斷沿染色體的距離。cm對應的堿基對的數量在基因組(染色體的不同區域對交換具有不同的偏好性)和物種(即基因組的總大小)之間廣泛變化。例如,已經估計四倍體棉花基因組包括分布在26條染色體上的約2200-3000mb的dna,其總的重組長度為每厘摩約400kb(smith和cothren,“cotton:origin,history,technologyandproduction”,第421頁)。在擬南芥中,1cm對應于大概217kb,而在例如玉米中,其為約1460kb(mézardc.meioticrecombinationhotspotsinplants.biochemsoctrans.2006aug34:531-4;civardi等,therelationshipbetweengeneticandphysicaldistancesinthecloneda1-sh2intervalofthezeamaysl.genome.procnatlacadsciusa.199491(17):8268-72,第8271頁;來自http://bionumbers.hms.harvard.edu/default.aspx)。如本文所用,“緊靠”因此指修飾和原種事件將作為單個遺傳單位遺傳至少一代的至少99%的可能性,并因而表示在原種事件的1cm、0.5cm、0.1cm、0.05cm、0.01cm、0.005cm或0.001cm內。對于堿基對,“緊靠”可以指距離現有原種事件5000kb、1000kb、500kb、100kb、50kb、10kb、5kb、4kb、3kb、2kb、1kb、750bp或500bp內(取決于物種和基因組中的定位),例如在現有原種事件1kb-10kb之間或1kb-5kb之間。將會清楚的是,預定位點以及識別位點應該如此定位,以至于不以負面方式干擾現有原種事件。反之亦然,現有原種事件也應該不負面影響新引入的修飾的功能,例如新插入的轉基因的功能表達。目前證明,緊靠現有原種事件,例如在事件的側翼序列之一產生修飾是可能的,其不負面影響現有事件(在該情況下是草銨膦耐受性或鱗翅目抗性)并導致新修飾的良好功能性表達(例如草甘膦耐受性或hppd抑制劑型除草劑耐受性)。在一個實施方案中,“緊靠”因而指在原種事件的側翼序列之一內。在本發明的上下文中,優選事件是棉花ghb119,也稱為ee-gh6(描述于2008/151780,其中也稱為gbh119,保藏號atccpta-8398)。seqidno.4代表ghb119事件5’側翼序列的核苷酸序列,而seqidno.3代表ghb119事件3’側翼序列的核苷酸序列,其中后者含有本文所述cot-5/6大范圍核酸酶(seqidno.1及其反向互補序列seqidno.2)的識別位點。seqidno.1的識別位點對應于seqidno.3從核苷酸2114-2135的核苷酸序列。本文所述的大范圍核酸酶因此能夠識別并切割緊靠現有轉基因事件,如ghb119的核苷酸序列。因此,在一個實施方案中,預定位點和/或識別位點定位在原種事件的側翼序列之一中。本發明包含的原種事件的側翼序列并非詳盡地列于表1中。原種事件ghb119的側翼序列的核苷酸序列由seqidno:3(3’)和seqidno:4(5’)代表。在另一實施方案中,所述識別序列包含seqidno:1或2的核苷酸序列。本文所述的重新設計的大范圍核酸酶基于用作支架的天然存在的大范圍核酸酶i-crei。i-crei是發現于chlamydomonasrheinhardti的葉綠體中的歸巢內切核酸酶(thompson等1992,gene119,247-251)。該內切核酸酶是識別23srrna基因中的假-回文22bpdna位點并產生用作引入內含子的雙鏈dna斷裂的同二聚體。i-crei是攜帶單個laglidadg基序的內切核酸酶組的成員。laglidadg酶含有共有基序的一個或兩個拷貝。單一基序酶,如i-crei作為同二聚體起作用,而雙重基序酶是具有兩個分離結構域的單體。因此,當來自i-crei支架的重新設計的大范圍核酸酶用于識別22bp的目的核苷酸序列時,設計兩個單體單元,每一個識別22bp識別位點的一部分,其為在22bp識別位點誘導雙鏈斷裂一致所需(wo2007/047859)。可通過將兩個單體單元連接成一個單鏈大范圍核酸酶來完成協調作用,或者也可以通過促進異二聚體的形成來完成,如例如在wo2007/047859中所述。此類特定設計的大范圍核酸酶的實例描述于例如ep10005926.0和ep10005941.9中(未公布)。天然存在的i-crei單體的氨基酸序列提供為seqidno.16。為了重新設計i-crei單體單元,以便其異二聚體識別seqidno.1和/或2的核苷酸序列,以下氨基酸存在于大范圍核酸酶單元1中所提及的位置上:a.位置32處的s;b.位置33處的y;c.位置38處的q;d.位置80處的q;e.位置40處的s;f.位置42處的t;g.位置77處的r;h.位置68處的y;i.位置70處的q;j.位置75處的h;k.位置44處的t;l.位置24處的i;m.位置26處的q;n.位置28處的k;o.位置30處的n;并且其中以下氨基酸存在于大范圍核酸酶單元2中:p.位置70處的s;q.位置44處的q;r.位置24處的k;s.位置26處的a;t.位置28處的k;u.位置30處的n;v.位置32處的s;w.位置33處的y;x.位置38處的q;y.位置80處的q;z.位置40處的s;aa.位置42處的t;bb.位置77處的q;cc.位置68處的y。在圖1中提供了其圖示。任選地,對于i-crei氨基酸序列,谷氨酰胺(gln/q)存在于位置47上的兩個亞基中。重新設計的雙鏈斷裂誘導酶可以包含,但不需要包含核定位信號(nls),如sv40大t抗原的nls[raikhel,plantphysiol.100:1627-1632(1992)及其中的參考文獻[kalderon等cell39:499-509(1984)]。所述核定位信號可以定位在蛋白質中的任何位置,但便利地定位在蛋白質的n末端。該核定位信號可以替換雙鏈斷裂誘導酶的一個或多個氨基酸。應該指出,如果已經為重新設計的大范圍核酸酶在蛋白質n末端提供了nls,如sv40的10或12個氨基酸nls,應該相應地易位(增加)氨基酸位置。同樣,在事件中,兩個單體單元連接成單鏈大范圍核酸酶,也應該易位第二個單元的位置。還可以通過確定如下所述的最佳比對鑒定i-crei氨基酸序列(seqidno.16)的對應氨基酸位置。將清楚的是,在單鏈重新設計的大范圍核酸酶中,單元的順序是不相關的,即無論上述單元1和單元2確實以該順序存在于單鏈氨基酸內還是單元2在單鏈氨基酸中位于單元1之前對于兩個單元被組合以能夠識別靶序列沒有產生差異。適合于本發明的重新設計的大范圍核酸酶可以包含如seqidno.6代表的氨基酸序列,其編碼包含由接頭序列(氨基酸166-203)偶聯并且前面是核定位序列(氨基酸1-10)的兩個亞基(分別是氨基酸11-165和204-360)的單鏈大范圍核酸酶。或者,此類大范圍核酸酶可以由兩個單體單元組成,所述單體單元可以作為異二聚體切割識別位點。該異二聚體大范圍核酸酶還可以包含seqidno5.從氨基酸位置11-165和204-360的氨基酸序列。便利地,可以通過編碼此類大范圍核酸酶的植物可表達的重組(嵌合)基因的表達來提供dsbi酶。為此,包含編碼重新設計的大范圍核酸酶或大范圍核酸酶單體單元的核苷酸序列的dna區可以有效連接植物可表達的啟動子和任選地參與轉錄終止和多腺苷酸化的dna區,并引入植物、植物部分或植物細胞。可以瞬時或穩定引入編碼dsbi酶的重組基因。還可以通過將翻譯成dsbi酶的rna分子引入細胞中來向植物、植物部分或植物細胞中引入所述dsbi酶。或者,所述dsbi酶可以直接作為蛋白質被引入到植物、植物部分或植物細胞中。用于向植物、植物部分、組織或植物細胞中引入dna或rna分子或蛋白質的方法在本申請的其他地方有描述。就本發明的目的而言,術語“植物有效的啟動子”和“植物可表達的啟動子”表示能夠在植物、植物組織、植物器官、植物部分或植物細胞中驅動轉錄的啟動子。這包括植物來源的任何啟動子,也包括非植物來源的任何啟動子,其能夠在植物細胞中指導轉錄。可以用于這方面的啟動子是組成型啟動子,如花椰菜花葉病毒(camv)35s轉錄物的啟動子(hapster等,1988,mol.gen.genet.212:182-190)、camv19s啟動子(美國專利號5,352,605;wo84/02913;benfey等,1989,emboj.8:2195-2202)、地三葉斑點病毒啟動子no4或no7(wo96/06932)、rubisco小亞基啟動子(美國專利號4,962,028)、泛素啟動子(holtorf等,1995,plantmol.biol.29:637-649)、t-dna基因啟動子,如來自農桿菌的章魚堿合酶(ocs)和胭脂堿合酶(nos)啟動子,和其在植物中的組成型表達為本領域技術人員所知的基因的其他啟動子。可以用于這方面的其他啟動子是組織特異性或器官特異性啟動子,優選種子特異性啟動子,如2s白蛋白啟動子(joseffson等,1987,j.biol.chem.262:12196-12201)、菜豆球蛋白啟動子(美國專利號5,504,200;bustos等,1989,plantcell1.(9):839-53)、豆科啟動子(shirsat等,1989,mol.gen.genet.215(2):326-331)、“未知種子蛋白質”(usp)啟動子(baumlein等,1991,mol.gen.genet.225(3):459-67)、油菜籽蛋白啟動子(美國專利號5,608,152;stalberg等,1996,planta199:515-519)、擬南芥油質蛋白啟動子(wo98/45461)、蕓苔bce4啟動子(wo91/13980),和其在植物中的種子特異性表達為本領域技術人員所知的基因的其他啟動子。可以使用的其他啟動子是組織特異性或器官特異性啟動子,像器官原基特異性啟動子(an等,1996,plantcell8:15-30)、莖特異性啟動子(keller等,1988,emboj.7(12):3625-3633)、葉特異性啟動子(hudspeth等,1989,plantmol.biol.12:579-589)、葉肉特異性啟動子(如光誘導的rubisco啟動子)、根特異性啟動子(keller等,1989,genesdev.3:1639-1646)、塊莖特異性啟動子(keil等,1989,emboj.8(5):1323-1330)、脈管組織特異性啟動子(peleman等,1989,gene84:359-369)、雄蕊選擇性啟動子(wo89/10396,wo92/13956)、開裂區帶特異性啟動子(wo97/13865)等。適合于本發明的編碼dsbi酶(重新設計的大范圍核酸酶)的核苷酸序列可以包含seqidno.5的從核苷酸位置3120-3584的核苷酸序列和seqidno.5從核苷酸位置3698-4169的核苷酸序列(不包括接頭和nls)。在單鏈大范圍核酸酶的情況下,這可以包括兩個亞基之間的接頭序列,如由seqidno.5從核苷酸位置3584-3697的核苷酸序列編碼的接頭序列。dsbi酶編碼核苷酸序列中還可以包括編碼核定位信號的核苷酸序列,如seqidno.5從位置3091-3119的核苷酸序列。為了便于克隆和其他重組dna技術,可以有利地在編碼大范圍核酸酶,特別是單鏈大范圍核酸酶的區域中包括在植物中有功能的內含子。可以通過改編gc含量、密碼子選擇、消除不想要的核苷酸序列來優化編碼dsbi酶的dna區用于在植物中表達。可以進一步優化編碼區用于在植物中表達并且合成的編碼區可以具有已經被設計以滿足以下標準的核苷酸序列:a)核苷酸序列編碼如本文所述的有功能的重新設計的歸巢內切核酸酶;b)密碼子選擇適合于靶生物的優選密碼子選擇;c)核苷酸序列具有約50%-約60%的gc含量;d)核苷酸序列不包含選自以下的核苷酸序列:gataat、tataaa、aatata、aatatt、gataaa、aatgaa、aataag、aataaa、aataat、aaccaa、atataa、aatcaa、atacta、ataaaa、atgaaa、aagcat、attaat、atacat、aaaata、attaaa、aattaa、aataca和cataaa;e)核苷酸不包含選自ccaat、attgg、gcaat和attgc的核苷酸序列;f)核苷酸序列不包含選自attta、aaggt、aggta、ggta或gcagg的序列;g)核苷酸序列不包含由選自g或c的7個連續核苷酸組成的gc串;h)核苷酸序列不包含由選自a或t的5個連續核苷酸組成的at串;和i)核苷酸序列不包含編碼leu、ile、val、ser、pro、thr、ala的密碼子,所述密碼子在位置2和3上包含ta或cg對(即所述核苷酸序列不包含密碼子tta、cta、ata、gta、tcg、ccg、acg和gcg)。還將清楚的是,用于描述方法的術語,如“引入dna片段”以及“從細胞再生植物”不表示此類dna片段必需通過轉化技術而被引入。實際上,本領域技術人員將立即清楚的是,還可以通過育種或雜交技術將目的dna分子從一種植物引入到另一種植物中。因此,與本申請相關聯的“引入”涉及通過任何已知手段將遺傳信息置于植物細胞或植物中。這可以通過本領域已知的任何方法完成,用于將rna或dna轉化到植物細胞、組織、原生質體或整株植物中或如下所述通過將所述rna或dna漸滲到植物中來完成。更特別地,“引入”表示穩定整合到植物的基因組中。可以通過本領域已知的任何方法,包括農桿菌介導的轉化,也可以通過直接dna轉移方法將核酸分子引入到植物細胞中。用于向細胞/組織(完整的植物細胞或部分降解的組織或植物細胞)中遞送dna的多種方法為本領域所知,并包括如美國專利號5,384,253中闡明的電穿孔;如美國專利號5,015,580;5,550,318;5,538,880;6,160,208;6,399,861;和6,403,865中闡明的微粒轟擊(基因槍(biolistics));通過粒子轟擊進行的棉花轉化報道于例如wo92/15675中;如美國專利號5,635,055;5,824,877;5,591,616;5,981,840;和6,384,301中闡明的農桿菌介導的轉化;棉花的農桿菌介導的轉化已經描述于例如美國專利5,004,863、美國專利6,483,013和wo2000/71733中;如美國專利號5,508,184中闡明的原生質體轉化、電穿孔、化學輔助的轉化、脂質體介導的轉化(參見例如a.deshayes,等(1985)emboj.4:2731-7.)、碳纖維、碳化硅纖維或硼酸鋁纖維(一般稱為晶須)(參見例如j.brisibe,exp.bot.51(343):187-196(2000);dunwell(1999)methodsmol.biol.111:375-82;和美國專利號5,464,765)、顯微注射(參見例如,tj.reich,efal.(1986)biotechnology4:1001-1004)和病毒介導的轉化(參見例如,s.b.gelvin,(2005)natbiotechnol.23:684-5,wo90/12107,wo03/052108和wo2005/098004)、利用附著到粒子上的異源外源dna轟擊植物細胞、彈道粒子加速、氣溶膠束轉化(美國專利申請號20010026941;美國專利號4,945,050;國際公開號wo91/00915;美國專利申請號2002015066,wo01/038514;全部并入本文作為參考)、lec1轉化、peg轉化和多種其他非粒子直接介導的方法來轉移dna。如本文所用,“直接dna轉移”是將dna引入到植物細胞中的任何方法,其不涉及使用天然農桿菌屬物種(agrobacteriumspp.),且能夠將dna引入到植物細胞中。在一個實施方案中,核酸分子,如修復dna和/或dsbi酶表達構建體通過直接dna轉移,例如通過粒子轟擊引入到植物細胞中。在另一實施方案中,使用農桿菌發生dna分子的引入。在具體的實施方案中,植物細胞包含在胚胎發生愈傷組織,優選脆弱愈傷組織(即植物細胞是愈傷組織細胞)中。術語“愈傷組織”或“胚胎發生愈傷組織”指作為植物組織培養的結果,主要是產生的胚胎發生細胞和細胞群的無組織團塊。脆弱的愈傷組織指具有脆弱質地的愈傷組織,其具有形成枝條和根并最終再生成完整植株的潛力。此類愈傷組織可以進一步區別在于鸚鵡綠/乳脂色、液體培養基中容易分散的細胞團、以及一種結節狀形狀。愈傷組織可以再生自/誘導自多種組織的外植體,例如下胚軸、子葉、不成熟的合子胚、葉、花藥、小孢子母細胞、花瓣、胚珠、根、和分生組織、莖細胞和葉柄。為在棉花植物細胞中靶向基因組修飾的目的轉化胚胎發生愈傷組織描述于美國臨時申請61/493579和ep11004570.5中(并入本文作為參考,特別是第12-15頁,第40-50段,和第31-35頁,實施例2-5)。在預選位點上誘導雙鏈斷裂的能力開啟了若干可能的應用,即插入、替換或缺失一個或多個核苷酸。如果修復dna分子中存在的目的dna待插入到預選位點中,那么這可以通過同源重組或通過非同源末端連接方法發生。雙鏈斷裂還可以用于在預選位點上誘導小缺失或插入的形成,由此可能失活包含預選位點的核苷酸序列的基因或調節元件。預選位點的雙鏈斷裂還將使用修復dna促進該位點附近的dna區替換成目的dna,例如在wo06/105946、wo08/037436或wo08/148559中所述。如果雙鏈dna斷裂誘導伴隨著用作模板的修復dna分子的引入,那么雙鏈斷裂修復可以基本上以三種方式發生。修復dna可以在dsb位點通過在兩端的非同源末端連接整合到基因組dna,或者如果在修復dna中存在與預選位點的上游和/或下游區域具有同源性的一個或兩個側翼區,那么修復dna的整合也可以通過同源重組發生(部分地)。照此,在預選位點的雙鏈斷裂也將促進將那一位點附近的dna區替換成目的dna區,例如在wo06/105946、wo08/037436或wo08/148559中所述。為了通過同源重組在預選位點插入目的dna,該修復dna可以包含至少一個側翼dna區,該側翼dna區具有與預選位點上游或下游的dna區的核苷酸序列相似的核苷酸序列。外源dna也可以包含位于該分子相反端的兩個側翼dna區,并且這些區域分別與預選位點上游和下游的dna區的核苷酸序列具有充分同源性,以允許所述側翼區與所述上游和下游區域之間的重組。如本文所用,“側翼dna區”是修復dna中的dna區域,所述修復dna具有與靶dna序列或預選位點的的上游或下游的dna區域(同源區)分別具有同源性(即高序列同一性)的核苷酸序列。這允許更好地控制插入目的dna。的確,通過同源重組的整合將允許目的dna與植物核基因組在高達核苷酸水平上的精確連接。為了具有充分同源性用于重組,修復dna的側翼dna區可以在長度方面變化并且應至少約10個核苷酸長。然而,側翼區在實際中可以盡可能長(例如高達約100-150kb),如完全細菌人工染色體(bac)。優選地,側翼區將是約50bp-約2000bp。此外,在目的dna側翼的區域不需要與同源區(預選位點側翼的dna區域)相同,并且可以與預選位點側翼的dna區域具有約80%-約100%的序列同一性,優選約95%-約100%的序列同一性。側翼區越長,對于同源性的要求越不嚴格。此外,優選的是在實際中在dsb附近序列同一性盡量高。此外,為了實現交換靶dna序列而不改變毗鄰dna序列的dna序列,側翼dna序列應優選與在預選位點側翼的上游和下游dna區域或與待被交換的靶dna序列相同。同樣的標準適用于彼此帶有同源性的上游和下游區域之間的重組,以通過染色體內同源重組移除間插dna序列。此外,修復dna的側翼區不需要與就在預選位點側翼的區域具有同源性,但是可以與距離預選位點更遠的核基因組的dna區域具有同源性。然后目的dna的插入將導致預選插入位點和dna同源區域之間靶dna的移除。換言之,定位于該同源區域之間的靶dna(即與外源修復dna的側翼區具有同源性的基因組區域)將被取代為定位于修復dna的兩個側翼區之間的目的dna。當修復dna僅由兩個側翼區組成時,即缺乏任何間插序列(目的dna),可以使用這種方法特異地缺失定位于兩個同源區域之間的基因組區域。如本文所用,“預選位點”或“預定位點”表示在植物基因組(例如核基因組)中的特定核苷酸序列,該核苷酸序列定位于靶dna序列中或靠近該靶dna序列,在此位置期望插入、替換或缺失一個或多個核苷酸。本領域技術人員將能夠選擇識別所選靶核苷酸序列的雙鏈dna斷裂誘導("dsbi”)酶或者改造此類dsbi內切核酸酶。或者,使用任何常規轉化方法或通過使用在其基因組中具有dsbi內切核酸酶識別位點的植物株系的常規育種,可以將dsbi酶識別位點引入到該植物基因組中,并且然后可以將任何想要的dna引入到該預選位點。在其他實施方案中,本發明提供dsbi酶,如上文所述非天然存在的dsbi酶在現有原種事件的附近修飾植物細胞的基因組的用途。如本文所用,“定位于附近”指雙鏈dna斷裂誘導的位點,即定位于距離預定位點100bp、250bp、500bp、lkbp、2kbp、3kbp、4kbp、5kbp-10kbp距離的dsbi酶的識別位點,即在基因組dna中待被修飾的位點(靶位點)。待插入的目的dna還可以包含可選擇的或可篩選的標志物,其可以在或不在插入之后移除。如本文所用,“可選擇的或可篩選的標志物”具有本領域通常的含義并且包括,但不限于植物可表達的草銨膦乙酰轉移酶、新霉素磷酸轉移酶、草甘膦氧化酶、草甘膦耐受的epsp酶、腈水解酶基因、突變型乙酰乳酸合酶或乙酰羥酸合酶基因、β-葡萄糖醛酸酶(gus)、r-基因座基因、綠色熒光蛋白等。植物細胞或植物(其中通過側翼dna區的同源重組已經引入可選擇的或可篩選的標志物和剩余的目的dna)的選擇可以例如通過篩選存在于轉化dna中但是定位于側翼dna區之外的序列的缺失來實現。的確,來自側翼dna區之外的轉化dna的序列的存在將表明轉化植物細胞的起源是通過隨機dna插入。為此,可選擇的或可篩選的標志物可以包括在側翼dna區之外的轉化dna分子中,然后這些標志物可以用于鑒定不具有定位于轉化dna之外的可選擇的或可篩選的標志物并且已經由通過側翼dna區的同源重引起的那些植物細胞。或者,轉化dna分子可以含有側翼dna區之外的可選擇的標志物,其允許選擇此類基因的缺失(陰性可選擇的標志物基因)。將清楚的是,本發明的方法允許任何目的dna的插入,所述目的dna包括包含具有特定核苷酸序列標識的核苷酸序列的dna,例如用于隨后的鑒定,或包含(可誘導的)增強子或沉默子的dna,例如以調節現有原種事件的表達。目的dna還可以包含一個或多個植物可表達的目的基因,其包括,但不限于除草劑耐受基因、昆蟲抗性基因、疾病抗性基因、非生物脅迫抗性基因、參與油類生物合成或碳水化合物生物合成的酶、參與纖維素強度和/或長度的酶、參與次生代謝物生物合成的酶。特別注意賦予對抑制酶羥苯丙酮酸二加氧酶(hppd)的除草劑的耐受性的除草劑耐受基因。羥苯丙酮酸二加氧酶是催化對-羥基苯基丙酮酸(hpp)轉化為尿黑酸的反應的酶。如wo96/38567、wo99/24585和wo99/24586、wo2009/144079、wo2002/046387或us6,768,044中所述,可以用編碼天然存在的抗性hppd酶的基因、或編碼突變的或嵌合的hppd酶的基因轉化對hppd抑制劑耐受的植物。也可以通過用編碼某些酶的基因轉化植物來獲得對hppd抑制劑的耐受性,盡管hppd抑制劑對天然hppd酶具有抑制作用,但所述酶使得能夠形成尿黑酸。此類植物和基因描述于wo99/34008和wo02/36787中。如wo2004/024928中所述,除編碼hppd耐受酶的基因之外,也可以通過用編碼具有預苯酸脫氫酶(pdh)活性的酶的基因轉化植物來改善植物對hppd抑制劑的耐受性。此外,通過向其基因組中加入編碼能夠代謝或降解hppd抑制劑的酶(如wo2007/103567和wo2008/150473中顯示的cyp450酶)的基因來產生對hppd抑制劑型除草劑更耐受的植物。在特定實施方案中,本發明還公開了包含嵌合基因的棉花植物細胞、植物部分、植物或種子,所述嵌合基因包含(a)編碼具有hppd活性的蛋白質的核酸序列,其中所述蛋白質在對應于seqidno:19的位置336的位置上具有色氨酸,其中所述蛋白質向所述植物提供對至少1x田間劑量的至少一種hppd抑制劑的耐受性,其有效連接(b)植物可表達的啟動子和任選地(c)翻譯終止和多腺苷酸化區。應理解的是,包含含有(a)編碼具有本發明hppd活性的蛋白質的核酸序列的嵌合基因的棉花植物細胞、植物部分、植物或種子不需要根據上文所述用于靶向修飾植物或植物細胞的基因組的方法產生,但還可以通過利用嵌合基因隨機轉化或通過常規育種方法生成,如該申請其他地方所述。術語“具有hppd活性的蛋白質”指催化將酪氨酸降解產物對羥基苯基丙酮酸(本文中縮寫為hpp)轉化為尿黑酸(本文中縮寫為hg)的反應的蛋白質。可以通過本領域所熟知的多種方法定義具有hppd活性的蛋白質的催化活性。wo2009/144079描述了多種合適的篩選方法。可以利用包含編碼如本文所述在細菌中表達的hppd蛋白質的核酸的嵌合基因來進行初期篩選,如在例如大腸桿菌(e.coli)中的互補測定(wo08/124495)。進一步并且更精細的篩選可以在表達如本文公開的hppd蛋白質的植物細胞或植物中進行。當檢查hppd蛋白質是否向植物,如棉花植物提供對如下文進一步所述的至少1x田間劑量的至少一種hppd抑制劑的耐受性時,也可以使用相同的篩選,其不同是除了hppd底物,還添加所述hppd抑制劑。可以測試的hppd抑制劑包括異氟草、環磺酮、甲基磺草酮、磺酰草吡唑、bicyclopyrone、苯吡唑草酮、莊無忌和磺草酮以及該申請中提及的其他hppd抑制劑。易于實施的篩選方法是(a)確定hppd抑制劑的劑量應該是對應于至少1x、至少2x或甚至至少3x或至少4x所選hppd抑制劑的田間劑量的劑量,所述hppd抑制劑不抑制具有本發明hppd活性的蛋白質,如seqidno:3的蛋白質,其例如用于體外或細胞培養實驗;(b)使各自包含本發明嵌合基因的植物細胞、植物部分或植物承受該劑量,其中具有hppd活性的蛋白質是如上文所述的蛋白質,并隨后(c)分離已經經受住該致死劑量的植物細胞、植物部分或植物。同時或備選地,可以評估并評分利用所述抑制劑處理后所述植物部分或植物的地上部分的損傷,如萎黃病、褪色和/或枯斑的程度。例如在所附實施例或如上所述進行來完成評分。對應于seqidno:19的位置336的位置指hppd蛋白質的氨基酸序列,所述蛋白質包含除seqidno:19以外的氨基酸序列,具有相似或相同的整體結構,并因此也具有對應于seqidno:19的位置336的氨基酸位置,但根據氨基酸序列的長度,可能是在不同位置上。可以通過比對這些蛋白質的序列與如上所述的seqidno:19確定其他hppd蛋白質中的對應位置。在本文所述植物細胞、植物部分、植物或種子的一個實例中,編碼具有hppd活性的蛋白質的所述核酸序列包含seqidno:20從nt949-2025的核苷酸序列或與其具有至少70%、至少80%、至少90%、至少95%、至少98%或至少99%序列同一性的核酸序列,其中所述蛋白質向植物提供對至少1x田間劑量的至少一種hppd抑制劑的耐受性。該定義中的序列包括編碼seqidno:21的氨基酸序列或與seqidno:21具有至少70%、至少80%、至少90%、至少95%、至少98%或至少99%序列同一性的氨基酸序列的那些序列。還包括所述seqidno:20從nt949-2025的片段,只要由此編碼的蛋白質向植物提供對至少1x田間劑量的至少一種hppd抑制劑的耐受性。此類片段包含例如至少300、至少400、至少500、至少600、至少700、至少800、至少900或至少1000個核苷酸。在一個實例中,任何上述片段包括編碼hppd蛋白質的核酸序列,所述蛋白質在等同于seqidno:19的位置336上的位置上包含色氨酸。在另一實例中,所述片段還包括所述色氨酸n和/或c末端的至少前5個、前10個、前20個、前30個氨基酸。因此,在本文所述植物細胞、植物部分、植物或種子的一個實例中,具有hppd活性的蛋白質的所述氨基酸序列包含seqidno:21的氨基酸序列或與其具有至少90%、至少95%、至少96%、至少97%、至少98%或至少99%序列同一性的氨基酸序列,其中所述蛋白質包含上述氨基酸取代并向所述植物提供對至少1x田間劑量的至少一種hppd抑制劑的耐受性。還包括所述seqidno:21的片段,只要具有hppd活性的所述蛋白質包含上述氨基酸取代并向植物提供對至少1x田間劑量的至少一種hppd抑制劑的耐受性。此類片段包含例如至少100個氨基酸、至少150個氨基酸、至少200個氨基酸、至少250個氨基酸、至少300個氨基酸。作為在植物細胞和植物中作為啟動子發揮功能的調控序列,可使用天然表達于植物中的基因的任何啟動子序列,特別是尤其表達于植物葉中的啟動子,如例如細菌、病毒或植物來源的“組成型”啟動子。植物可表達的啟動子可以是組成型啟動子,即能夠在大多數細胞類型中(以時空不依賴型方式)指導高水平表達的啟動子。植物可表達的組成型啟動子的實例包括細菌來源的啟動子,如來自農桿菌的章魚堿合酶(ocs)和胭脂堿合酶(nos)啟動子,還包括病毒來源的啟動子,如花椰菜花葉病毒(camv)35s轉錄物的啟動子(hapster等,1988,mol.gen.genet.212:182-190)或19srnas基因的啟動子(odell等,1985,nature.6;313(6005):810-2;美國專利號5,352,605;wo84/02913;benfey等,1989,emboj.8:2195-2202)、增強的2x35s啟動子(kayatal.,1987,science236:1299–1302;datla等(1993),plantsci94:139–149)、木薯葉脈花葉病毒的啟動子(csvmv;wo97/48819,us7,053,205)、2xcsvmv(wo2004/053135)、環病毒(au689311)啟動子、甘蔗桿狀dna病毒(scbv)啟動子(samac等,2004,transgenicres.13(4):349-61)、玄參花葉病毒(fmv)啟動子(sanger等,1990,plantmolbiol.14(3):433-43)、地三葉斑點病毒啟動子no4或no7(wo96/06932)和如us5,164,316、us5,196,525、us5,322,938、us5,359,142和us5,424,200中所述的增強的35s啟動子。在植物來源的啟動子中,將注意來自玉米和向日葵的植物核酮糖二羧化酶/加氧酶(rubisco)小亞基啟動子(us4,962,028;wo99/25842)、擬南芥組蛋白h4基因的啟動子(chabouté等,1987)、玉米、稻和甘蔗的泛素啟動子(holtorf等,1995,plantmol.biol.29:637-649,us5,510,474)、稻肌動蛋白1啟動子(act-1,us5,641,876)、如在ep0507698a1中所述的組蛋白啟動子、玉米乙醇脫氫酶1啟動子(adh-1)(來自http://www.patentlens.net/daisy/promoters/242.html))。如果其用途與各終止子的用途組合,那么還可以使用來自菊花的小亞基啟動子(outchkourov等,planta,216:1003–1012,2003)。或者,對植物的特定區域、組織或器官特異的啟動子序列可以用于表達本文公開的hppd蛋白質。可以使用的啟動子是組織特異性或器官特異性啟動子,如器官原基特異性啟動子(an等,1996,plantcell8:15-30)、莖特異性啟動子(keller等,1988,emboj.7(12):3625-3633)、葉肉特異性啟動子(如光誘導的rubisco啟動子)、根特異性啟動子(keller等,1989,genesdev.3:1639-1646)、脈管組織特異性啟動子(peleman等,1989,gene84:359-369)等。還可以使用從苯丙氨酸解氨酶(pal)、hmg-coa還原酶(hmg)、幾丁質酶、葡聚糖酶、蛋白酶抑制劑(pi)、pr1家族基因、胭脂堿合酶(nos)和vspb啟動子(us5670349,表3)、hmg2啟動子(us5670349)、蘋果β-半乳糖苷酶(abg1)啟動子和蘋果氨基環丙烷羧酸鹽合酶(acc合酶)啟動子(wo98/45445)有利選擇的誘導型啟動子。根據本發明,與啟動子組合,還可以使用其他調節序列,其定位在啟動子和編碼序列之間,如轉錄激活子(“增強子”),例如在申請wo87/07644中描述的煙草花葉病毒(tmv)或例如carrington&freed1990,j.virol.64:1590-1597描述的煙草蝕刻病毒(tev)的翻譯激活子。增強功能表達并由此增強除草劑耐受性的其他調節序列還可以定位在嵌合基因內。此類調節序列的一個實例是內含子。內含子是在前mrna中存在但通過精確的剪接機制切除后在成熟rna中缺失的間插序列。天然內含子增強基因表達(稱為內含子介導的增強作用(ime)的方法)的能力已經在多種生物,包括哺乳動物、昆蟲、線蟲和植物中已知(wo07/098042,第11-12頁)。ime一般描述為轉錄后機制,其通過穩定轉錄物導致基因表達增加。內含子需要以正常方向定位在啟動子與編碼序列之間。然而,已經描述了一些內含子,以影響翻譯,充當啟動子或位置和方向不依賴型轉錄增強子(chaubet-gigot等,2001,plantmolbiol.45(1):17-30,p27-28)。含有此類內含子的基因的實例包括來自稻肌動蛋白1基因的5’內含子(參見us5641876)、稻肌動蛋白2基因、玉米蔗糖合酶基因(clancy和hannah,2002,plantphysiol.130(2):918-29)、玉米醇脫氫酶-1(adh-1)和bronze-1基因(callis等1987genesdev.1(10):1183-200;mascarenhas等1990,plantmolbiol.15(6):913-20)、玉米熱激蛋白70基因(參見us5593874)、玉米皺縮1基因、馬鈴薯(solanumtuberosum)的光敏性1基因和矮牽牛(petuniahybrida)的熱激蛋白70基因(參見us5659122)、來自苜蓿的替換組蛋白h3基因(keleman等2002transgenicres.11(1):69-72)和擬南芥的任一替換組蛋白h3(組蛋白h3.3-樣)基因(chaubet-gigot等,2001,plantmolbiol.45(1):17-30)。其他合適的調節序列包括5’utrs。如本文所用,5’utr也稱為前導序列,是定位在轉錄起始位點和編碼區的起始密碼子之間的信使rna(mrna)的特定區域。其參與mrna的穩定性和翻譯效率。例如,35s轉錄起始位點下游的牽牛花葉綠素a/b結合蛋白基因的5’非翻譯前導可以用于增加報道基因表達的穩定狀態水平(harpster等,1988,molgengenet.212(1):182-90)。wo95/006742描述了來自編碼熱激蛋白的基因的5’非翻譯前導序列增加轉基因表達的用途。嵌合基因還可以包含在植物細胞中有效的轉錄終止或多腺苷酸化序列。作為轉錄終止或多腺苷酸化序列,可以使用細菌來源的任何對應序列,如例如根癌農桿菌(agrobacteriumtumefaciens)的nos終止子,病毒來源的任何對應序列,如例如camv35s終止子,植物來源的任何對應序列,如例如在公布的專利申請ep0633317a1中描述的組蛋白終止子。與本申請相關聯,可以通過優化編碼待在棉花中表達的蛋白質的序列,由此尤其考慮棉花的密碼子選擇進一步增強在棉花中賦予對至少一種hppd抑制劑的耐受性的嵌合基因的表達。因此,例如可以通過密碼子優化來優化編碼本文公開的hppd蛋白質的核酸序列用于在棉花中表達(例如通過www.entelechon.com可得)。編碼具有本文公開的hppd活性的蛋白質的棉花密碼子優化的核酸序列的實例由sedidno.20從nt949-2025代表。具有本文公開的hppd活性的蛋白質可以由seqidno:20從nt949-2025的核酸序列編碼或具有seqidno:21的氨基酸序列。在一個實例中,所述嵌合基因包含或由seqidno:20從位置88-位置2714的核酸序列組成。如在本文公開的植物細胞、植物部分、植物或種子中觀察到的對至少一種hppd抑制劑的耐受性由具有本文公開的hppd活性的蛋白質引起并被引入到所述植物細胞、植物部分、植物中,其向所述植物細胞、植物部分、植物或種子提供對至少一種hppd抑制劑的所述耐受性。術語“耐受性”或“耐受的”表示表達具有本文公開的hppd活性的蛋白質的植物對物質,特別是抑制hppd蛋白質的除草劑的易感性減少或完全缺失,任選地與植物本身的hppd蛋白質相比。更尤其地,所述術語表示內在耐受性的相對水平,即根據菌株或植物的可見指標表型在存在不同濃度的hppd抑制劑下篩選的具有hppd活性的蛋白質的上述易感性的減少或完全缺失,所述菌株或植物轉化有包含編碼各蛋白質的基因的核酸。所述hppd抑制劑可以選自任何可得的hppd抑制劑。一般地,向不表達hppd酶(其提供對所述hppd抑制劑的耐受性)的植物應用hppd抑制劑導致在應用后不久植物地上部分的嚴重損傷,而在表達本發明hppd蛋白質或相信提供耐受性的其他hppd蛋白質的植物中沒有觀察到損傷或僅較少的損傷。因此,與不表達所述hppd蛋白質或表達不同hppd蛋白質的植物相比,觀察到對所述至少一種hppd抑制劑的耐受性為表達本發明的hppd蛋白質并利用至少一種hppd抑制劑處理的植物提供了農藝學優勢。盡管本發明的植物在除草劑處理后顯示一些較少的損傷,但是所述植物在處理后短時間內恢復。因而,當被處理時,與未處理的植物相比,表達本發明的hppd蛋白質的植物優選不具有減少的農藝學表現,例如不展示減少的產量。目前,大多數市售hppd抑制劑屬于這四個化學家族之一:1)三酮類,例如,磺草酮[即2-[2-氯-4-(甲基磺酰基)苯甲酰基]-1,3-環己二酮],甲基磺草酮[即2-[4-(甲基磺酰基)-2-硝基苯甲酰基]-1,3-環己二酮];環磺酮[即2-[2-氯-4-(甲基磺酰基)-3-[(2,2,2,-三-氟乙氧基)甲基]苯甲酰基]-1,3-環-己二酮];莊無忌[即2-[2-氯-4-(甲基磺酰基)-3-[[(四氫-2-呋喃基)甲氧基]甲基]苯甲酰基]-1,3-環己二酮]];bicyclopyrone[即4-羥基-3-[[2-[(2-甲氧基乙氧基)甲基]-6-(三氟甲基)-3-吡啶基]羰基]二環[3.2.1]辛-3-烯-2-酮];雙環磺草酮[即3-(2-氯-4-甲磺酰基苯甲酰基)-2-苯基硫代二環[3.2.1]辛-2-烯-4-酮];2)異唑類,例如異氟草[即(5-環丙基-4-異唑基)[2-(甲磺酰基)-4-(三氟甲基)苯基]甲酮]或對應的二酮腈類化合物;和3)吡唑啉酮類(pyrazolinones),例如苯吡唑草酮[即[3-(4,5-二氫-3-異唑基)-2-甲基-4-(甲基磺酰基)苯基](5-羥基-1-甲基-1h-吡唑-4-基)甲酮],磺酰草吡唑[(5-羥基-1,3-二甲基吡唑-4-基(2-甲磺酰基-4-三氟甲基苯基(trifluaromethylphenyl))甲酮];pyrazofen[2-[4-(2,4-二氯苯甲酰基)-1,3-二甲基吡唑-5-基氧]苯乙酮]。用于本發明的hppd抑制劑的其他化合物種類是如在pct/ep2011/064820中公開的n-(四唑-5-基)-和n-(三唑-5-基)芳基甲酰胺和在wo2011/035874中公開的n-(1,2,5-二唑-3-基)苯酰胺。在一個實例中,所述至少一種hppd抑制劑選自異氟草、環磺酮、甲基磺草酮、磺酰草吡唑和苯吡唑草酮以及任何其他適用的hppd抑制劑或hppd除草劑,如在本文所列出的那些。與這些指標表型(生長抑制、萎黃病、褪色、一般而言葉損傷、萎蔫、枯斑、除草劑效果等等)相關的劑量應答和劑量應答的相對改變被方便地以例如在不同濃度的除草劑范圍內基于植物損傷(如植物地上部分的損傷,其可以表現為褪色、萎黃病和/或枯斑)、分生組織褪色癥狀等以正常方式的gr50(生長降低50%的濃度)或mic(最小抑制濃度)值來表示,其中值的增加對應于表達的hppd的內在耐受性增加。這些數據可以以例如來自劑量/應答曲線的gr50值來表示,所述曲線具有繪制于x軸上的“劑量”和繪制于y軸上的“百分比殺死(percentagekill)”、“除草劑效果”、“出苗綠色植物的數目”等,其中增加的gr50值對應于表達的hppd的內在耐受性水平增加。除草劑可以在出苗前或出苗后應用,如在所附實施例中所述。或者,可以基于上文提及的相關指征表型,例如在0-100范圍上評價植物其綠色部分或葉的損傷,其中0%表明沒有損傷,而100%表明完全褪色、萎黃病、枯斑和/或萎蔫等。如本文所用,“出苗前”指在從播種的種子生長的幼苗的土壤或生長培養基表面上出苗前,例如恰好在播種種子之前或在播種種子時,或恰好在播種后向其中播種種子的土壤或生長培養基中應用除草劑,例如至少一種hppd抑制劑。如本文所用,“出苗后”指在幼苗出苗后,例如在2-3苗期或之后向植物和土壤或生長培養基應用除草劑。包含如本文公開的嵌合hppd基因的植物細胞、植物部分、植物或種子對至少1x田間劑量的至少一種hppd抑制劑耐受。所述至少一種hppd抑制劑可以在出苗前期和/或出苗后期應用。如本文所用,“1x”表示hppd抑制劑,如hppd抑制型除草劑或包含hppd抑制劑的制劑的正常的單一田間劑量,以ga.i./ha表示,其中a.i.代表活性組分,如商業上所用。根據例如所用的hppd抑制劑、其應用的農作物和應用條件,例如其上生長農作物和期望存在雜草的土壤類型,田間劑量可以變化。1x田間劑量的商品化產品在產品標簽上有說明。根據產品和標簽建議,商品化1x田地率可以在例如18.41ga.i./ha(對于產品impacttm中存在的苯吡唑草酮)與例如138ga.i./ha(對于產品laudissctm中存在的環磺酮)之間變化。在本實施例田地試驗中,產品callistotm中存在的甲基磺草酮以對應于105ga.i./ha的1x田間劑量應用,并且產品impacttm中存在的苯吡唑草酮以對應于18.41ga.i./ha的1x田間劑量應用。用于所選hppd抑制劑的其他示例性1x田間劑量是異氟草(balanceflexxtm):105ga.i./hapyrasulfutole:37.5ga.i./ha環磺酮(2-{2-氯-4-甲磺酰基-3-[(2,2,2-三氟乙氧基)甲基]苯甲酰基}環己烷-1,3-二酮或2-[2-氯-4-(甲磺酰基)-3-[(2,2,2-三氟乙氧基)甲基]苯甲酰基]-1,3-環己二酮;制劑laudissctm):138ga.i./ha。環磺酮的另一示例性1x田間劑量:100ga.i./ha。因此,1.5x田間劑量指150%的1x劑量的應用劑量,2x田間劑量指200%的1x劑量的特定hppd抑制劑或hppd抑制劑制劑等的應用劑量。在另一實例中,本文公開的植物細胞、植物部分、植物或種子對至少2x至少一種hppd抑制劑的田間劑量具有耐受性。在其他實例中,本文公開的植物細胞、植物部分、植物或種子對至少3x或至少4x至少一種hppd抑制劑的田間劑量具有耐受性。在特定實施方案中,本文公開的植物細胞、植物部分、植物或種子對至少1xift、至少2xift或至少4xift的田間劑量具有耐受性。在甚至其他實施方案中,至少1xift、至少2xift或至少4xift的所述劑量在出苗前應用。如本文所用,對特定劑量的hppd抑制劑,如hppd抑制劑型除草劑的耐受性指利用所述除草劑劑量處理后展示最小量損傷的植物。因此,田地中本發明的除草劑耐受植物在至少一種hppd抑制劑處理后7天顯示不高于40%、不高于35%、不高于30%、不高于27%、不高于25%、不高于20%、不高于15%、不高于10%、不高于9%、不高于8%、不高于7%、不高于6%或不高于5%的植物地上部分的可見損傷。可以通過植物地上部分,包括葉和莖的褪色、萎黃病和/或枯斑的程度來評價損傷。因此,耐受性可以表現為在至少一種hppd抑制劑處理后7天顯示不高于40%、不高于35%、不高于30%、不高于27%、不高于25%、不高于20%、不高于15%、不高于10%、不高于9%、不高于8%、不高于7%、不高于6%或不高于5%的萎黃病、褪色和/或枯斑。在本申請的其他地方描述了損傷或植物應答評分。或者,田地中本發明的除草劑耐受植物在至少一種hppd抑制劑處理后21天顯示不高于20%、不高于15%、不高于10%、不高于9%、不高于8%、不高于7%、不高于6%或不高于5%的植物地上部分的可見損傷。換言之并且根據上文,耐受性可以表現為在至少一種hppd抑制劑處理后21天不高于20%、不高于15%、不高于10%、不高于9%、不高于8%、不高于7%、不高于6%或不高于5%的萎黃病、褪色和/或枯斑。在不同的可選項中,田地中本發明的除草劑耐受植物在至少一種hppd抑制劑處理后21天顯示不高于10%、不高于6%、不高于5%、不高于4%或不高于3%的植物地上部分的可見損傷。換言之并且根據上文,耐受性可以表現為在至少一種hppd抑制劑處理后21天不高于10%、不高于6%、不高于5%、不高于4%或不高于3%的萎黃病、褪色和/或枯斑。所述至少一種hppd抑制劑也可以是多于一種hppd抑制劑,如至少2種、至少3種、至少4種、至少5種、至少6種、至少10種或甚至更多種hppd抑制劑。由此,用于每一種上述選項的hppd抑制劑的任何組合是可能的。例如,本發明的植物可以對至少兩種hppd抑制劑耐受,或者本發明的嵌合基因可以賦予對至少兩種hppd抑制劑的耐受性,其中所述至少兩種抑制劑可以包含異氟草和苯吡唑草酮、異氟草和甲基磺草酮、異氟草和pyrasulfutole、異氟草和環磺酮、苯吡唑草酮和甲基磺草酮、苯吡唑草酮和pyrasulfutole、苯吡唑草酮和環磺酮、甲基磺草酮和pyrasulfutole、甲基磺草酮和環磺酮或pyrasulfultole和環磺酮。或者所述至少一種hppd抑制劑可以是至少三種hppd抑制劑,其中所述至少三種抑制劑可以包含異氟草、苯吡唑草酮和甲基磺草酮;異氟草、苯吡唑草酮和pyrasulfutole;異氟草、苯吡唑草酮和環磺酮;異氟草、甲基磺草酮和pyrasulfutole;異氟草、甲基磺草酮和環磺酮;異氟草、甲基磺草酮和pyrasulfutole;異氟草、異氟草和環磺酮;苯吡唑草酮、甲基磺草酮和pyrasulfutol;苯吡唑草酮、甲基磺草酮和環磺酮;苯吡唑草酮、pyrasulfutole和環磺酮或甲基磺草酮、pyrasulfutole和環磺酮。至少四種hppd抑制劑的組合可以包含異氟草、苯吡唑草酮、甲基磺草酮和pyrasulfutole;異氟草、苯吡唑草酮、甲基磺草酮和環磺酮;異氟草、甲基磺草酮、pyrasulfutole和環磺酮或苯吡唑草酮、甲基磺草酮、pyrasulfutole和環磺酮。至少五種hppd抑制劑的示例性組合包含異氟草、苯吡唑草酮、甲基磺草酮、pyrasulfutole和環磺酮。不同hppd抑制劑的耐受性水平還可以不同。例如,盡管本發明的嵌合基因賦予棉花植物對2x一種hppd抑制劑的耐受性,其對另一種可以是1x。在任何情況下,提供對至少1x如要求保護的每一種hppd抑制劑的耐受性。其他除草劑耐受性基因包括編碼酶5-烯醇式丙酮酰莽草酸-3-磷酸合酶(epsps)的基因。此類epsps基因的實例是細菌鼠傷寒沙門氏菌(salmonellatyphimurium)的aroa基因(突變株ct7)(comai等,1983,science221,370-371)、細菌土壤桿菌種(agrobacteriumsp.)的cp4基因(barry等,1992,curr.topicsplantphysiol.7,139-145)、編碼牽牛花epsps的基因(shah等,1986,science233,478-481)、番茄epsps(gasser等,1988,j.biol.chem.263,4280-4289)或牛筋草epsps(wo01/66704)。突變的epsps也可以如描述于例如ep0837944、wo00/66746、wo00/66747或wo02/26995。如美國專利號5,776,760和5,463,175中所述,也可以通過表達編碼草甘膦氧化還原酶的基因來獲得草甘膦耐受性植物。如例如wo02/36782、wo03/092360、wo05/012515和wo07/024782中所述,也可以通過表達編碼草甘膦乙酰轉移酶的基因來獲得草甘膦耐受性植物。例如在wo01/024615或wo03/013226中所述,也可以通過選擇包含上述基因的天然出現突變的植物來獲得草甘膦耐受性植物。賦予草甘膦耐受性的epsps基因描述于例如美國專利申請號11/517,991、10/739,610、12/139,408、12/352,532、11/312,866、11/315,678、12/421,292、11/400,598、11/651,752、11/681,285、11/605,824、12/468,205、11/760,570、11/762,526、11/769,327、11/769,255、11/943801或12/362,774中。賦予草甘膦耐受性的其他基因,如脫羧酶基因描述于例如美國專利申請11/588,811、11/185,342、12/364,724、11/185,560或12/423,926中。在編碼合適的epsps(其賦予對以epsps為靶標的除草劑的耐受性)的dna序列中,將更特別地注意編碼植物epsps,特別是玉米epsps,特別是這樣的玉米epsps的基因,其包含兩個突變,特別是氨基酸位置102處一個突變和氨基酸位置106處一個突變(wo2004/074443),并且其描述于專利申請us6566587,在下文命名為雙突變型玉米epsps或2mepsps(對于編碼該epsps的核酸序列,也參見seqidno:20從nt4602-5939,并且對于所得酶的氨基酸序列,參見seqidno:22)。在編碼epsps的dna序列,并且更特別地編碼上述基因的情況下,編碼將epsps蛋白質靶向葉綠體的轉運肽的序列有利地位于編碼這些酶的序列之前。表達epsps蛋白質的特定目的葉綠體轉運肽是如美國專利5,510,471或5,633,448中所述的“優化的轉運肽”。例如美國專利申請號11/760,602中所述,甚至其他除草劑耐受性基因可以編碼對除草劑解毒的酶或對抑制作用具有抗性的突變型谷氨酰胺合酶。一種此類有效的解毒酶是編碼膦絲菌素乙酰轉移酶的酶(如來自鏈霉菌屬種的bar或pat蛋白)。膦絲菌素乙酰轉移酶例如描述于美國專利號5,561,236;5,648,477;5,646,024;5,273,894;5,637,489;5,276,268;5,739,082;5,908,810和7,112,665中。仍另外的除草劑耐受性基因編碼變體als酶(也稱為乙酰羥酸合成酶,ahas),如例如描述于tranel(特瑞納)和wright(賴特)(2002,weedscience(《雜草科學》)50:700-712)中,而且在美國專利號5,605,011、5,378,824、5,141,870、以及5,013,659中。硫酰脲耐受植物以及咪唑啉酮耐受植物的生產描述于美國專利號5,605,011、5,013,659、5,141,870、5,767,361、5,731,180、5,304,732、4,761,373、5,331,107、5,928,937、以及5,378,824、以及國際公開wo96/33270中。其他的咪唑啉酮耐受性基因還描述于例如wo2004/040012、wo2004/106529、wo2005/020673、wo2005/093093、wo2006/007373、wo2006/015376、wo2006/024351、以及wo2006/060634中。另外的硫酰脲以及咪唑啉酮耐受性基因描述于例如wo07/024782和美國專利申請號61/288958中。昆蟲抗性基因可以包括一種編碼序列,該序列編碼:1)一種來自蘇云金桿菌的殺昆蟲晶體蛋白或它的一個殺昆蟲部分,例如由crickmore(克里克莫爾)等人列舉的殺昆蟲晶體蛋白(1998,microbiologyandmolecularbiologyreviews(《微生物學和分子生物學綜述》),62:807-813),由crickmore(克里克莫爾)等人(2005)在蘇云金桿菌毒素命名法中更新,聯機在:http://www.lifesci.sussex.ac.uk/home/neil_crickmore/bt/),或它的殺昆蟲部分,例如cry蛋白類cry1ab、cry1ac、cry1b、cry1c、cry1d、cry1f、cry2ab、cry3aa、或cry3bb的蛋白或它的殺昆蟲部分(例如ep1999141和wo2007/107302),或者由如例如描述于美國專利申請號12/249,016中的合成基因編碼的這種蛋白;或2)一種來自蘇云金桿菌的晶體蛋白或它的一部分,該部分在一個第二其他的來自蘇云金桿菌的晶體蛋白或它的一部分的存在下是殺昆蟲的,例如由cry34和cry35晶體蛋白構成的二元毒素(moellenbeck(默勒本克)等人,2001,nat.biotechnol.(《自然生物技術》)19:668-72;schnepf(史涅夫)等人2006,appliedenvironm.microbiol.(《應用與環境微生物學》)71,1765-1774)或由cry1a或cry1f蛋白和cry2aa或cry2ab或cry2ae蛋白組成的二元毒素(美國專利申請號12/214,022和ep08010791.5);或3)一種包括來自蘇云金桿菌的不同的殺昆蟲晶體蛋白的部分的雜交殺昆蟲蛋白,例如以上1)的蛋白的雜交體或以上2)的蛋白的雜交體,例如由玉米事件mon98034(wo2007/027777)生產的cry1a.105蛋白;或4)以上1)至3)的任意一種蛋白,其中一些特別是1至10個氨基酸已經被另一氨基酸取代以便獲得一個對目標昆蟲物種的更高殺昆蟲活性、和/或用以擴大所影響的目標昆蟲物種的范圍、和/或由于在克隆或轉化期間引入編碼dna的變化,例如在玉米事件mon863或mon88017中的cry3bb1蛋白、或在玉米事件mir604中的cry3a蛋白;或5)一種來自蘇云金桿菌或蠟樣芽胞桿菌的殺昆蟲分泌蛋白或它的一個殺昆蟲部分,例如列舉在:http://www.lifesci.sussex.ac.uk/home/neil_crickmore/bt/vip.html的營養期殺昆蟲蛋白(vip),例如來自vip3aa蛋白類的蛋白;或6)一種來自蘇云金桿菌或蠟樣芽胞桿菌的分泌蛋白,該分泌蛋白在來自蘇云金桿菌或蠟樣芽胞桿菌的一個第二分泌蛋白的存在下是殺昆蟲的,例如由vip1a和vip2a蛋白構成的二元毒素(wo94/21795);或7)一種包括來自蘇云金桿菌或蠟樣芽胞桿菌的不同的分泌蛋白的部分的雜交殺昆蟲蛋白,例如以上1)中的蛋白的一種雜交體或以上2)中的蛋白的一種雜交體;或8)以上5)至7)的任意一種蛋白,其中一些特別是1至10個氨基酸已經被另一氨基酸取代以便獲得一個對目標昆蟲物種的更高殺昆蟲活性、和/或用以擴大所影響的目標昆蟲物種的范圍、和/或由于在克隆或轉化期間引入編碼dna的變化(盡管仍然編碼一種殺昆蟲蛋白),例如在棉花事件cot102中的vip3aa蛋白;或9)一種來自蘇云金桿菌或蠟樣芽胞桿菌的分泌蛋白,該分泌蛋白在來自蘇云金桿菌的晶體蛋白的存在下是殺昆蟲的,例如由vip3和cry1a或cry1f構成的二元毒素(美國專利申請號61/126083和61/195019)或由vip3蛋白和cry2aa或cry2ab或cry2ae蛋白構成的二元毒素(美國專利申請號12/214,022和ep08010791.5);10)以上9)的一種蛋白,其中一些特別是1至10個氨基酸已經被另一氨基酸取代以便獲得一個對目標昆蟲物種的更高殺昆蟲活性、和/或用以擴大所影響的目標昆蟲物種的范圍、和/或由于在克隆或轉化期間引入編碼dna的變化(盡管仍然編碼一種殺昆蟲蛋白)。如在此使用,“昆蟲抗性基因”,進一步包括轉基因,這些轉基因包含一個在表達時生產雙鏈rna的序列,該雙鏈rna被植物昆蟲有害生物攝取時抑制這種昆蟲有害生物的生長,如例如描述于wo2007/080126、wo2006/129204、wo2007/074405、wo2007/080127和wo2007/035650中。非生物脅迫耐受性基因包括1)在植物細胞或植物中能夠減少聚(adp核糖)聚合酶(parp)基因的表達和/或活性的一種轉基因,如描述于wo00/04173、wo/2006/045633、ep04077984.5或ep06009836.5中。2)能夠減少植物或植物細胞的parg編碼基因的表達和/或活性的一種轉基因,如例如描述于wo2004/090140中。3)編碼一種煙酰胺腺嘌呤二核苷酸補救合成通路的植物功能性酶的一種轉基因,這些酶包括煙酰胺酶、煙酰酸磷酸核糖基轉移酶、煙酸單核苷酸腺嘌呤轉移酶、煙酰胺腺嘌呤二核苷酸合成酶或煙酰胺磷酸核糖基轉移酶,如例如描述于ep04077624.7、wo2006/133827、pct/ep07/002433、ep1999263或wo2007/107326中。涉及碳水化合物生物合成的酶包括描述于以下的那些,例如:ep0571427、wo95/04826、ep0719338、wo96/15248、wo96/19581、wo96/27674、wo97/11188、wo97/26362、wo97/32985、wo97/42328、wo97/44472、wo97/45545、wo98/27212、wo98/40503、wo99/58688、wo99/58690、wo99/58654、wo00/08184、wo00/08185、wo00/08175、wo00/28052、wo00/77229、wo01/12782、wo01/12826、wo02/101059、wo03/071860、wo2004/056999、wo2005/030942、wo2005/030941、wo2005/095632、wo2005/095617、wo2005/095619、wo2005/095618、wo2005/123927、wo2006/018319、wo2006/103107、wo2006/108702、wo2007/009823、wo00/22140、wo2006/063862、wo2006/072603、wo02/034923、ep06090134.5、ep06090228.5、ep06090227.7、ep07090007.1、ep07090009.7、wo01/14569、wo02/79410、wo03/33540、wo2004/078983、wo01/19975、wo95/26407、wo96/34968、wo98/20145、wo99/12950、wo99/66050、wo99/53072、us6,734,341、wo00/11192、wo98/22604、wo98/32326、wo01/98509、wo01/98509、wo2005/002359、us5,824,790、us6,013,861、wo94/04693、wo94/09144、wo94/11520、wo95/35026或wo97/20936,或涉及多聚果糖,尤其是菊粉和果聚糖型的生產的酶,如披露于ep0663956、wo96/01904、wo96/21023、wo98/39460、和wo99/24593中,如披露于wo95/31553、us2002031826、us6,284,479、us5,712,107、wo97/47806、wo97/47807、wo97/47808和wo00/14249中的α-1,4-葡聚糖的生產,如披露于wo00/73422中的α-1,6分支α-1,4-葡聚糖的生產,如披露于例如wo00/47727、wo00/73422、ep06077301.7、us5,908,975和ep0728213的alternan的生產,如例如披露于wo2006/032538、wo2007/039314、wo2007/039315、wo2007/039316、jp2006304779、和wo2005/012529中的透明質酸的生產。也可以根據本發明處理的棉花植物或植物栽培種(可以通過植物生物技術方法如遺傳工程來獲得的)是具有改變的纖維特性的植物,如棉花植物。此類植物可以通過遺傳轉化或通過選擇含有賦予此類改變的纖維特性的突變的植物來獲得,并且包括:a)如描述于wo98/00549中含有纖維素合酶基因的改變形式的植物,如棉花植物b)如描述于wo2004/053219中含有rsw2或rsw3同源核酸的改變形式的植物,如棉花植物c)如描述于wo01/17333中具有蔗糖磷酸合酶升高的表達的植物,如棉花植物d)如描述于wo02/45485中具有蔗糖合酶升高的表達的植物,如棉花植物e)如描述于wo2005/017157中或如描述于ep08075514.3或美國專利申請號61/128,938中的植物,如棉花植物,其中例如通過下調纖維選擇性β-1,3-葡聚糖酶改變了纖維細胞基礎上胞間連絲的門控計時。如描述于wo2006/136351中,具有例如通過表達包括nodc和幾丁質合酶基因在內的n-乙酰葡糖胺轉移酶基因而改變的反應性的纖維的植物,如棉花植物。本發明的實施方案還提供編碼如本文所述的重新設計的大范圍核酸酶的嵌合基因,其中所述嵌合基因包含有效連接編碼蛋白質的dna區的植物可表達的啟動子,所述蛋白質包含對應于i-crei的氨基酸序列的氨基酸序列作為支架(seqidno16),其在位置32處包含s;位置33處包含y;位置38處包含q;位置80處包含q;位置40處包含s;位置42處包含t;位置77處包含r;位置68處包含y;位置70處包含q;位置75處包含h;位置44處包含t;位置24處包含i;位置26處包含q;位置28處包含k和位置30處包含n,和/或其中所述嵌合基因包含有效連接編碼第二種蛋白質的dna區的植物可表達的啟動子,所述蛋白質包含對應于i-crei的氨基酸序列的氨基酸序列作為支架(seqidno16),其在位置70處包含s;位置44處包含q;位置24處包含k;位置26處包含a;位置28處包含k;位置30處包含n;位置32處包含s;位置33處包含y;位置38處包含q;位置80處包含q;位置40處包含s;位置42處包含t;位置77處包含q和位置68處包含y,如包含seqid5或seqid6的氨基酸序列的蛋白質(可以通過比對來確定重新設計的大范圍核酸酶關于i-crei的對應氨基酸位置)。本領域技術人員將理解,除了核基因組,本發明的方法還可以應用于修飾例如葉綠體基因組或線粒體基因組,由此dsb誘導在預定位點上,并且可以進一步通過向內切核酸酶提供正確靶信號而得到增強。應理解的是,本發明的方法可以應用于任何植物(被子植物或裸子植物),包括但不限于棉花、蕓苔、油菜、大豆、蔬菜、馬鈴薯、浮萍屬、煙草屬、擬南芥、苜蓿、大麥、黃豆、玉米、棉花、亞麻、黍、豌豆、油菜、稻、黑麥、紅花、高粱、大豆、向日葵、煙草、草坪草(turfgrass)、小麥、蘆筍、甜菜根和甜菜、綠花椰菜、卷心菜、胡蘿卜、花椰菜、芹菜、黃瓜、茄子、萵苣、洋蔥、油菜、胡椒、馬鈴薯、西葫蘆、蘿卜、菠菜、南瓜、甘蔗、番茄、胡瓜、杏仁、蘋果、杏、香蕉、黑莓、藍莓、可可、櫻桃、椰子、酸果蔓、棗、葡萄、葡萄柚、番石榴、獼猴桃、檸檬、酸橙、芒果、甜瓜、油桃、橙子、木瓜、西番蓮果、桃、花生、梨、菠蘿、阿月渾子、李子、覆盆子、草莓、橘子、核桃和西瓜。在特定實施方案中,所述植物是棉花。如本文所用,棉花指任何現有的棉花品種。例如,棉花植物細胞可以來自用于培養棉花的品種。最常用的棉花品種是海島棉(gossypiumbarbadense)、陸地棉(g.hirsutum)、亞洲棉(g.arboreum)和草棉(g.herbaceum)。其他品種包括阿非利加棉(g.africanum)和雷蒙德氏棉(g.raimondii)。相同方法應用于本文所述的棉花植物、棉花植物部分和棉花種子。本文公開的實例棉花植物包括胚胎發生愈傷組織可以衍生的那些,例如coker312、coker310、coker5acalasj-5、gsc25110、fibermax819、siokra1-3、t25、gsa75、acalasj2、acalasj4、acalasj5、acalasj-c1、acalab1644、acalab1654-26、acalab1654-43、acalab3991、acalagc356、acalagc510、acalagam1、acalac1、acalaroyale、acalamaxxa、acalaprema、acalab638、acalab1810、acalab2724、acalab4894、acalab5002、nonacala"picker"siokra、"stripper"品種fc2017、coker315、stoneville506、stoneville825、dp50、dp61、dp90、dp77、des119、mcn235、hbx87、hbx191、hbx107、fc3027、chembreda1、chembreda2、chembreda3、chembreda4、chembredb1、chembredb2、chembredb3、chembredc1、chembredc2、chembredc3、chembredc4、paymaster145、hs26、hs46、sicala、pimas6oroblancopima、fibermaxfm5013、fibermaxfm5015、fibermaxfm5017、fibermaxfm989、fibermaxfm832、fibermaxfm966、fibermaxfm958、fibermaxfm989、fibermaxfm958、fibermaxfm832、fibermaxfm991、fibermaxfm819、fibermaxfm800、fibermaxfm960、fibermaxfm966、fibermaxfm981、fibermaxfm5035、fibermaxfm5044、fibermaxfm5045、fibermaxfm5013、fibermaxfm5015、fibermaxfm5017或fibermaxfm5024以及具有其衍生基因型的植物。這些適合引入本文公開的嵌合基因。本發明的目標還提供根據本發明的方法產生的植物細胞、植物部分和植物,如果實、種子、胚、生殖組織、分生組織區、愈傷組織、葉、根、芽、花、纖維、脈管組織、配子體、孢子體、花粉和小孢子,其特征在于它們在基因組中包含特定修飾(插入、替換和/或缺失)或它們包含本發明的嵌合hddp基因。通過傳統育種方法產生的包含dna修飾事件的植物的配子、種子、胚(合子或體細胞的)、后代或雜合體也包括在本發明的范圍內。此類植物可以含有插入到靶序列處或替代靶序列的目的dna或可以具有缺失(甚至是單個核苷酸)的特定dna序列,并且與其祖先植物的區別將僅在于在交換后存在該異源dna或dna序列或缺失特定缺失的序列。或者,此類植物含有本發明的嵌合hppd基因。將清楚的是,本發明的植物細胞、植物部分和植物可以來自如上文列出的任何植物,包括所有棉花植物。在特定實施方案中,本文所述的植物細胞是非繁殖的植物細胞或不能再生成植物的植物細胞或不能通過從無機物,如水、二氧化碳和無機鹽通過光合作用合成碳水化合物和蛋白質維持其自身的植物細胞。種子由種皮包被的胚胎發生植物與儲存營養物形成。它是裸子植物和被子植物的成熟胚珠的產物,其在受精并且母本植物中生長至某一程度后發生。本文公開的種子保留母本的區別性特征,如通過自交或雜交獲得的種子,例如雜合種子(通過將兩個近交的親本株系雜交獲得)。本文所述的植物細胞和植物,如通過本文所述的方法獲得的那些可以進一步用于本領域熟知的育種程序,如雜交、自交和回交。育種程序可以涉及雜交,以產生f1(第一代)代,隨后是自交若干代(產生f2、f3等)。所述育種程序還涉及回交(bc)步驟,由此后代與親本株系之一進行回交,稱為回歸親本。“漸滲”表示基因通過自然手段,即通過包含本文所述嵌合基因的植物與不包含所述嵌合基因的植物雜交整合到植物的基因組中。可以選擇包含嵌合基因的那些后代。因此,本文還公開的是用于產生包含引入的性狀(即,基因組修飾或本發明的hppd基因)的植物的方法,其包括將本文公開的植物與另一植物或自身雜交并選擇包含引入的性狀的后代的步驟。本文所述和/或通過本文公開的方法獲得的植物細胞和植物還可以進一步用于隨后的轉化方法中,例如以引入其他嵌合基因。本發明的植物和種子可以進一步被化合物,如選自以下列表的化合物處理:水果/蔬菜:除草劑:莠去津(atrazine)、除草定(bromacil)、氟丙嘧草酯(butafenacil)、敵草隆(diuron)、吡氟禾草靈(fluazifop)、草銨膦、草甘膦、吡氯黃隆(halosulfuron)、甲基吡氯黃隆(halosulfuron-methyl)、indaziflam、利谷隆(linuron)、賽克津(metribuzin)、對草快(paraquat)、拿草特(propyzamide)、稀禾定(sethoxydim)、西瑪津(simazine)、氟樂靈(trifluralin)。殺蟲劑:齊墩螨素(abamectin)、滅螨醌(acequinocyl)、吡蟲清(acetamiprid)、涕滅威(aldicarb)、艾扎丁(azadirachtin)、丙硫克百威(benfuracarb)、聯苯肼酯(bifenazate)、噻嗪酮(buprofezin)、甲萘威(carbaryl)、蟲螨威(carbofuran)、chlorantraniliprole(rynaxypyr)、毒死蜱(chlorpyrifos)、chromafenozide、噻蟲胺(clothianidin)、cyanopyrafen、cyantraniliprole(cyazypyr)、丁氟螨酯(cyflumetofen)、β-氟氯氰菊酯(beta-cyfluthrin)、γ-氯氟氰菊酯(gamma-cyhalothrin)、λ-氯氟氰菊酯(lambda-cyhalothrin)、α-氯氰菊酯(alpha-cypermethrin)、溴氰菊酯(deltamethrin)、殺螨硫隆(diafenthiuron)、呋蟲胺(dinotefuran)、甲氨基阿維菌素-benzoate(emamectin-benzoate)、高氰戊菊酯(esfenvalerate)、fenamiphos、殺螨錫(fenbutatin-oxid)、氟啶蟲酰胺(flonicamid)、fluacrypyrim、flubendiamide、fluensulfone、氟吡菌酰胺(fluopyram)、flupyradifurone、imicyafos、吡蟲啉(imidacloprid)、二唑蟲(indoxacarb)、氰氟蟲胺(metaflumizone)、滅蟲威(methiocarb)、甲氧苯酰肼(methoxyfenozide)、雙苯氟脲(novaluron)、拒嗪酮(pymetrozine)、新喹唑啉(pyrifluquinazon)、pyriproxifen、spinetoram、艾克敵105(spinosad)、螺螨酯(spirodiclofen)、螺甲螨酯(spiromesifen)、螺蟲乙酯(spirotetramat)、sulfoxaflor、噻蟲啉(thiacloprid)、噻蟲嗪(thiamethoxam)、硫雙威(thiodicarb)、殺蟲隆(triflumuron)、1-(3-氯吡啶-2-基)-n-[4-氰基-2-甲基-6-(甲基氨基甲酰基)苯基]-3-{[5-(三氟甲基)-2h-四唑-2-基]甲基}-1h-吡唑-5-甲酰胺、1-(3-氯吡啶-2-基)-n-[4-氰基-2-甲基-6-(甲基氨基甲酰基)苯基]-3-{[5-(三氟甲基)-1h-四唑-1-基]甲基}-1h-吡唑-5-甲酰胺、1-{2-氟-4-甲基-5-[(2,2,2-三氟乙基)亞磺酰基]苯基}-3-(三氟甲基)-1h-1,2,4-三唑-5-胺、(1e)-n-[(6-氯吡啶-3-基)甲基]-n'-氰基-n-(2,2-二氟乙基)乙脒、堅強芽孢桿菌(bacillusfirmus)、堅強芽孢桿菌菌株i-1582、蘇云金芽孢桿菌、枯草芽孢桿菌(bacillussubtilis)、枯草芽孢桿菌菌株gb03、枯草芽孢桿菌菌株qst713、金龜子綠僵菌(metarhiziumanisopliae)、金龜子綠僵菌菌株f52。水果/蔬菜殺真菌劑:ametoctradin、amisulbrom、腈嘧菌酯(azoxystrobin)、苯噻菌胺(benthiavalicarb)、n-[9-(二氯亞甲基)-1,2,3,4-四氫-1,4-亞甲基萘甲叉-5-基]-3-(二氟甲基)-1-甲基-1h-吡唑-4-甲酰胺(benzovindiflupyr、benzodiflupyr)、bixafen、啶酰菌胺(boscalid)、克菌丹(captan)、多菌靈(carbendazim)、百菌清(chlorothalonil)、銅(copper)、氰霜唑(cyazofamid)、環氟菌胺(cyflufenamid)、清菌脲(cymoxanil)、環唑醇(cyproconazole)、環丙嘧啶(cyprodinil)、醚唑(difenoconazole)、dimetomorph、二噻農(dithianon)、氧唑菌(epoxiconazole)、唑酮菌(famoxadone)、咪唑菌酮(fenamidone)、環酰菌胺(fenhexamid)、fenpyrazamine、氟啶胺(fluazinam)、氟菌(fludioxonil)、氟吡菌胺(fluopicolide)、氟吡菌酰胺、氟嘧菌酯(fluoxastrobin)、喹唑菌酮(fluquinconazole)、fluthianil、氟唑菌酰胺(fluxapyroxad)、滅菌丹(folpet)、藻菌磷(fosetyl)、異丙定(iprodione)、異丙菌胺(iprovalicarb)、isopyrazam、異噻菌胺(isotianil)、醚菌酯(kresoxim-methyl)、代森錳鋅(mancozeb)、雙炔酰菌胺(mandipropamid)、meptyldinocap、甲霜靈/精甲霜靈(metalaxyl/mefenoxam)、代森聯(metiram)、苯菌酮(metrafenone)、腈菌唑(myclobutanil)、戊菌唑(penconazole)、氟唑菌苯胺(penflufen)、吡噻菌胺(penthiopyrad)、phosphonicacid(h3po3)、啶氧菌胺(picoxystrobin)、百維靈(propamocarb)、丙環唑(propiconazole)、甲基代森鋅(propineb)、丙氧喹啉(proquinazid)、丙硫菌唑(prothioconazole)、唑菌胺酯(pyraclostrobin)、二甲嘧菌胺(pyrimethanil)、pyriofenone、1-(4-{4-[(5r)-5-(2,6-二氟苯基)-4,5-二氫-1,2唑-3-基]-1,3-噻唑-2-基}哌啶-1-基)-2-[5-甲基-3-(三氟甲基)-1h-吡唑-1-基]乙酮、喹氧靈(quinoxyfen)、sedaxane、螺茂胺(spiroxamine)、sulphur、戊唑醇(tebuconazole)、甲基托布津(thiophanate-methyl)、福美雙(thiram)、唑菌醇(triadimenol)、肟菌酯(trifloxystrobin)。谷類:除草劑:2,4-d、磺氨黃隆(amidosulfuron)、氟丁酰草胺(beflubutamid)、滅草松(bentazon)、治草醚(bifenox)、溴苯腈(bromoxynil)、乙基氟酮唑草(carfentrazone-ethyl)、綠麥隆(chlorotoluron)、綠黃隆(chlorsulfuron)、吲哚酮草酯(cinidon-ethyl)、clodinafop-propargyl、二氯皮考啉酸(clopyralid)、麥草畏(dicamba)、2,4-滴丙酸(dichlorprop)、2,4-滴丙酸-p(dichlorprop-p)、甲基氯甲草(diclofop-methyl)、吡氟草胺(diflufenican)、唑禾草靈(fenoxaprop)、雙氟磺草胺(florasulam)、氟酮黃隆鈉(flucarbazone-sodium)、氟噻草胺(flufenacet)、甲基氟啶黃隆鈉(flupyrsulfuron-methyl-sodium)、氟草煙(fluroxypyr)、呋草酮(flurtamone)、草甘膦、咪草啶酸(imazamox)、咪草酯(imazamethabenz)、碘黃隆(iodosulfuron)、碘苯腈(ioxynil)、異丙隆(isoproturon)、異惡草胺(isoxaben)、mcpa、mcpb、mecoprop-p、甲磺胺黃隆(mesosulfuron)、metsulfuron)、胺硝草(pendimethalin)、picolinafen、pinoxaden、丙苯磺隆(propoxycarbazone)、芐草丹(prosulfocarb)、pyraflufen、磺酰草吡唑、吡唑磺草胺(pyroxsulam)、乙黃黃隆(sulfosulfuron)、thiencarbazone、噻黃隆(thifensulfuron)、traloxydim、野麥畏(triallat)、醚苯黃隆(triasulfuron)、苯黃隆(tribenuron)、氟樂靈、三氟甲黃隆(tritosulfuron)(安全劑:mefenpyr、mefenpyr-diethyl、cloquintocet)。殺真菌劑:腈嘧菌酯、n-[9-(二氯亞甲基)-1,2,3,4-四氫-1,4-亞甲基萘甲叉-5-基]-3-(二氟甲基)-1-甲基-1h-吡唑-4-甲酰胺(benzovindiflupyr)、benzodiflupyr、bixafen、啶酰菌胺、多菌靈、萎銹靈(carboxin)、百菌清、環氟菌胺、環唑醇、環丙嘧啶、醚唑、醚菌胺(dimoxystrobin)、烯唑醇(diniconazole)、氧唑菌、苯銹啶(fenpropidin)、丁苯嗎啉(fenpropimorph)、氟菌、氟吡菌酰胺、氟嘧菌酯、喹唑菌酮、氟硅唑(flusilazole)、fluthianil、粉唑醇(flutriafol)、氟唑菌酰胺、烯菌靈(imazalil)、環戊唑醇(ipconazole)、isopyrazam、醚菌酯、mefenoxam、甲霜靈(metalaxyl)、環戊唑菌(metconazole)、叉氨苯酰胺(metominostrobin)、苯菌酮、腈菌唑、氟唑菌苯胺、吡噻菌胺、啶氧菌胺、丙氯靈(prochloraz)、丙環唑、丙氧喹啉、丙硫菌唑、唑菌胺酯、pyriofenone、喹氧靈、sedaxane、硅噻菌胺(silthiofam)、螺茂胺、戊唑醇、涕必靈(thiabendazole)、甲基托布津、唑菌醇、肟菌酯、戊叉唑菌(triticonazole)、堅強芽孢桿菌、堅強芽孢桿菌菌株i-1582、枯草芽孢桿菌、枯草芽孢桿菌菌株gb03、枯草芽孢桿菌菌株qst713。殺蟲劑:吡蟲清、艾扎丁、丙硫克百威、氟氯菊酯(bifenthrin)、chlorantraniliprole(rynaxypyr)、chlorpyriphos、噻蟲胺、cyantraniliprole(cyazypyr)、β-氟氯氰菊酯、α-氯氰菊酯、溴氰菊酯、殺螨硫隆、樂果(dimethoate)、dinetofuran、銳勁特(fipronil)、fluensulfone、氟吡菌酰胺、flupyradifurone、imicyafos、吡蟲啉、氰氟蟲胺、metamidophos、滅蟲威、甲拌磷(phorate)、抗蚜威(pirimicarb)、拒嗪酮、新喹唑啉、螺蟲乙酯、sulfoxaflor、七氟菊酯(tefluthrin)、噻蟲啉、噻蟲嗪、硫雙威、transfluthirn、殺蟲隆、1-(3-氯吡啶-2-基)-n-[4-氰基-2-甲基-6-(甲基氨基甲酰基)苯基]-3-{[5-(三氟甲基)-2h-四唑-2-基]甲基}-1h-吡唑-5-甲酰胺、1-(3-氯吡啶-2-基)-n-[4-氰基-2-甲基-6-(甲基氨基甲酰基)苯基]-3-{[5-(三氟甲基)-1h-四唑-1-基]甲基}-1h-吡唑-5-甲酰胺、1-{2-氟-4-甲基-5-[(2,2,2-三氟乙基)亞硫酰基]苯基}-3-(三氟甲基)-1h-1,2,4-三唑-5-胺、(1e)-n-[(6-氯吡啶-3-基)甲基]-n'-氰基-n-(2,2-二氟乙基)乙脒、堅強芽孢桿菌、堅強芽孢桿菌菌株i-1582、枯草芽孢桿菌、枯草芽孢桿菌菌株gb03、枯草芽孢桿菌菌株qst713、金龜子綠僵菌f52。玉米:除草劑/植物生長調節劑(pgrs):苯草醚(aclonifen)、莠去津、甲草胺(alachlor)、滅草松、bicyclopyrone、溴苯腈、乙草胺(acetochlor)、苯草醚、麥草畏、二氯皮考啉酸、dimethenamid-p、雙氟磺草胺、氟噻草胺、氟草煙、甲酰胺黃隆(foramsulfuron)、草銨膦、草甘膦、isoxadifen、異氟草、(s-)metolachlor、甲基磺草酮、煙黃隆(nicosulfuron)、胺硝草、烯草胺(pethoxamid)、氟嘧黃隆(primisulfuron)pyroxasulfon、玉嘧黃隆(rimsulfuron)、磺草酮、環磺酮、特丁津(terbuthylazin)、thiencarbazone、thifensulfuron-methyl、苯吡唑草酮、saflufenacil(安全劑:isoxadifen-ethyl)。殺蟲劑:齊墩螨素、吡蟲清、艾扎丁、丙硫克百威、氟氯菊酯、蟲螨威、chlorantraniliprole(rynaxypyr)、毒死蜱、噻蟲胺、cyantraniliprole(cyazypyr)、(β-)氟氯氰菊酯、λ-氯氟氰菊酯、(α)-氯氰菊酯、溴氰菊酯、呋蟲胺、ethiprole,銳勁特、flubendiamide、fluensulfone、氟吡菌酰胺、flupyradifurone、二唑蟲、imicyafos、吡蟲啉、氟丙氧脲(lufenuron)、氰氟蟲胺、滅蟲威、拒嗪酮、新喹唑啉、spinetoram、艾克敵105、螺甲螨酯、螺蟲乙酯、sulfoxaflor、七氟菊酯、特丁磷(terbufos)、噻蟲嗪、硫雙威、殺蟲隆、tebupirimphos、噻蟲啉、1-(3-氯吡啶-2-基)-n-[4-氰基-2-甲基-6-(甲基氨基甲酰基)苯基]-3-{[5-(三氟甲基)-2h-四唑-2-基]甲基}-1h-吡唑-5-甲酰胺、1-(3-氯吡啶-2-基)-n-[4-氰基-2-甲基-6-(甲基氨基甲酰基)苯基]-3-{[5-(三氟甲基)-1h-四唑-1-基]甲基}-1h-吡唑-5-甲酰胺、1-{2-氟-4-甲基-5-[(2,2,2-三氟乙基)亞磺酰基]苯基}-3-(三氟甲基)-1h-1,2,4-三唑-5-胺、(1e)-n-[(6-氯吡啶-3-基)甲基]-n'-氰基-n-(2,2-二氟乙基)乙脒、堅強芽孢桿菌、堅強芽孢桿菌菌株i-1582、枯草芽孢桿菌、枯草芽孢桿菌菌株gb03、枯草芽孢桿菌菌株qst713、金龜子綠僵菌f52。殺真菌劑:腈嘧菌酯、n-[9-(二氯亞甲基)-1,2,3,4-四氫-1,4-亞甲基萘甲叉-5-基]-3-(二氟甲基)-1-甲基-1h-吡唑-4-甲酰胺(benzovindiflupyr、benzodiflupyr)、bixafen、啶酰菌胺、多菌靈、chenopodiumquinjoa、環唑醇、醚菌胺、氧唑菌、咪唑菌酮、銳勁特、氟吡菌酰胺、fluthianil、氟嘧菌酯、氟硅唑、粉唑醇、氟唑菌酰胺、環戊唑醇、isopyrazam、代森錳鋅、mefenoxam、甲霜靈、叉氨苯酰胺、環戊唑菌、腈菌唑、氟唑菌苯胺、吡噻菌胺、啶氧菌胺、丙環唑、丙硫菌唑、唑菌胺酯、saponin、sedaxane、戊唑醇、福美雙、唑菌醇、肟菌酯、福美鋅(ziram)、堅強芽孢桿菌、堅強芽孢桿菌菌株i-1582、枯草芽孢桿菌、枯草芽孢桿菌菌株gb03、枯草芽孢桿菌菌株qst713、短小芽孢桿菌(bacilluspumulis)、短小芽孢桿菌菌株gb34。稻:除草劑:莎稗磷(anilofos)、四唑黃隆(azimsulfuron)、benfuresate、芐嘧黃隆(bensulfuron)、噻草平(bentazone)、雙環磺草酮、吡草酮(benzofenap)、bispyribac、溴丁酰草胺(bromobutide)、丁草胺(butachlor)、唑草胺(cafenstrole)、乙基氟酮唑草、異惡草酮(clomazone)、稗草胺(clomeprop)、芐草隆(cumyluron)、cyhalofop、香草隆(daimuron)、哌草丹(dimepiperate)、禾草畏(esprocarb)、乙氧嘧黃隆(ethoxysulfuron)、唑禾草靈、fenoxasulfone)、四唑草胺(fentrazamide)、氟吡黃隆(flucetosulfuron)、氟噻草胺、吡氯黃隆、啶咪黃隆(imazosulfuron)、茚草酮(indanofan)、ipfencarbazone)、苯噻草胺(mefenacet)、甲基磺草酮、metamifop、metazosulfuron、草達滅(molinate)、萘丙胺(naproanilide)、orthosulfamuron、炔丙唑草(oxadiargyl)、oxadiazone、氯嗪草(oxaziclomefone)、五氟磺草胺(penoxsulam)、戊唑草(pentoxazone)、丙草胺(pretilachlor)、profoxydim、敵稗(propanil)、propyrisulfuron、雙唑草腈(pyraclonil)、pyrazolate、吡嘧黃隆(pyrazosulfuron)、芐草唑、稗草畏(pyributicarb)、pyriftalid、肟啶草(pyriminobac-methyl)、pyrimisulfan、二氯喹啉酸(quinclorac)、莊無忌、噻醚草胺(thenylchlor)、殺草丹(thiobencarb)、triafamone。殺蟲劑:吡蟲清、艾扎丁、丙硫克百威、噻嗪酮、蟲螨威、chlorantraniliprole(rynaxypyr)、chlorpyriphos、chromafenozide、噻蟲胺、cyantraniliprole(cyazypyr)、氯氰菊酯、溴氰菊酯、diazinon、呋蟲胺、甲氨基阿維菌素-benzoate、ethiprole、etofenprox、fenobucarb、銳勁特、flubendiamide、fluensulfone、氟吡菌酰胺、flupyradifurone、imicyafos、吡蟲啉、isoprocarb、氰氟蟲胺、拒嗪酮、新喹唑啉、spinetoram、艾克敵105、螺蟲乙酯、sulfoxaflor、噻蟲啉、噻蟲嗪、1-(3-氯吡啶-2-基)-n-[4-氰基-2-甲基-6-(甲基氨基甲酰基)苯基]-3-{[5-(三氟甲基)-2h-四唑-2-基]甲基}-1h-吡唑-5-甲酰胺、1-(3-氯吡啶-2-基)-n-[4-氰基-2-甲基-6-(甲基氨基甲酰基)苯基]-3-{[5-(三氟甲基)-1h-四唑-1-基]甲基}-1h-吡唑-5-甲酰胺、1-{2-氟-4-甲基-5-[(2,2,2-三氟乙基)亞磺酰基]苯基}-3-(三氟甲基)-1h-1,2,4-三唑-5-胺、(1e)-n-[(6-氯吡啶-3-基)甲基]-n'-氰基-n-(2,2-二氟乙基)乙脒、堅強芽孢桿菌、堅強芽孢桿菌菌株i-1582、枯草芽孢桿菌、枯草芽孢桿菌菌株gb03、枯草芽孢桿菌菌株qst713、金龜子綠僵菌f52。殺真菌劑:腈嘧菌酯、n-[9-(二氯亞甲基)-1,2,3,4-四氫-1,4-亞甲基萘甲叉-5-基]-3-(二氟甲基)-1-甲基-1h-吡唑-4-甲酰胺(benzovindiflupyr、benzodiflupyr)、bixafen、多菌靈、carpropamid、百菌清、copper-oxychloride、環唑醇、diclocymet、醚唑、edifenphos、氧唑菌、ferimzone、氟菌、氟吡菌酰胺、氟嘧菌酯、氟硅唑、fluthianil、flutolanil、氟唑菌酰胺、furametpyr、gentamycin、hexaconazole、hymexazol、環戊唑醇、iprobenfos(ibp)、異丙定、isoprothiolane、異噻菌胺、kasugamycin、代森錳鋅、mefenoxam、甲霜靈、叉氨苯酰胺、腈菌唑、orysastrobin、pencycuron、氟唑菌苯胺、phthalide、probenazole、丙氯靈、百維靈、丙環唑、甲基代森鋅、丙硫菌唑、pyroquilon、sedaxane、simconazole、鏈霉素(streptomycin)、sulphur、戊唑醇、thifluzamide、甲基托布津、福美雙、tiadinil、tricyclazole、肟菌酯、validamycin、堅強芽孢桿菌、堅強芽孢桿菌菌株i-1582、枯草芽孢桿菌、枯草芽孢桿菌菌株gb03、枯草芽孢桿菌菌株qst713。棉花:除草劑:氟酮唑草、clethodim、敵草隆、吡氟禾草靈(fluazifop-butyl)、flumioxazin、fluometuron、草銨膦、草甘膦、msma、norflurazon、oxyfluorfen、胺硝草、prometryn、pyrithiobac-sodium、tepraloxydim、thidiazuron、trifloxysulfuron、氟樂靈。殺蟲劑:齊墩螨素、acephate、吡蟲清、涕滅威、艾扎丁、氟氯菊酯、chlorantraniliprole(rynaxypyr)、毒死蜱、噻蟲胺、cyantraniliprole(cyazypyr)、(β-)氟氯氰菊酯、γ-氯氟氰菊酯、λ-氯氟氰菊酯、氯氰菊酯、溴氰菊酯、殺螨硫隆、呋蟲胺、甲氨基阿維菌素-benzoate、氟啶蟲酰胺、flubendiamide、fluensulfone、氟吡菌酰胺、flupyradifurone、imicyafos、吡蟲啉、二唑蟲、氰氟蟲胺、拒嗪酮、pyridalyl、新喹唑啉、spinetoram、艾克敵105、螺甲螨酯、spirotetramat、sulfoxaflor、噻蟲啉、噻蟲嗪、硫雙威、殺蟲隆、1-(3-氯吡啶-2-基)-n-[4-氰基-2-甲基-6-(甲基氨基甲酰基)苯基]-3-{[5-(三氟甲基)-2h-四唑-2-基]甲基}-1h-吡唑-5-甲酰胺、1-(3-氯吡啶-2-基)-n-[4-氰基-2-甲基-6-(甲基氨基甲酰基)苯基]-3-{[5-(三氟甲基)-1h-四唑-1-基]甲基}-1h-吡唑-5-甲酰胺、1-{2-氟-4-甲基-5-[(2,2,2-三氟乙基)亞磺酰基]苯基}-3-(三氟甲基)-1h-1,2,4-三唑-5-胺、(1e)-n-[(6-氯吡啶-3-基)甲基]-n'-氰基-n-(2,2-二氟乙基)乙脒、堅強芽孢桿菌、堅強芽孢桿菌菌株i-1582、枯草芽孢桿菌、枯草芽孢桿菌菌株gb03、枯草芽孢桿菌菌株qst713、金龜子綠僵菌f52。殺真菌劑:腈嘧菌酯、n-[9-(二氯亞甲基)-1,2,3,4-四氫-1,4-亞甲基萘甲叉-5-基]-3-(二氟甲基)-1-甲基-1h-吡唑-4-甲酰胺(benzovindiflupyr、benzodiflupyr)、bixafen、啶酰菌胺、多菌靈、百菌清、copper、環唑醇、醚唑、醚菌胺、氧唑菌、咪唑菌酮、氟啶胺、氟吡菌酰胺、氟嘧菌酯、氟唑菌酰胺、環戊唑醇、異丙定、isopyrazam、異噻菌胺、代森錳鋅、maneb、mefenoxam、甲霜靈、叉氨苯酰胺、pencycuron、氟唑菌苯胺、吡噻菌胺、啶氧菌胺、甲基代森鋅、丙硫菌唑、唑菌胺酯、quintozene、sedaxane、戊唑醇、tetraconazole、甲基托布津、唑菌醇、肟菌酯、堅強芽孢桿菌、堅強芽孢桿菌菌株i-1582、枯草芽孢桿菌、枯草芽孢桿菌菌株gb03、枯草芽孢桿菌菌株qst713。大豆:除草劑:甲草胺、噻草平、chlorimuron-ethyl、cloransulam-methyl、唑禾草靈、吡氟禾草靈、fomesafen、草銨膦、草甘膦、咪草啶酸、imazaquin、imazethapyr、(s-)metolachlor、賽克津、胺硝草、tepraloxydim、氟樂靈。殺蟲劑:吡蟲清、艾扎丁、chlorantraniliprole(rynaxypyr)、噻蟲胺、cyantraniliprole(cyazypyr)、(β-)氟氯氰菊酯、γ-氯氟氰菊酯、λ-氯氟氰菊酯、溴氰菊酯、呋蟲胺、甲氨基阿維菌素-benzoate、ethiprole、銳勁特、氟啶蟲酰胺、flubendiamide、fluensulfone、氟吡菌酰胺、flupyradifurone、imicyafos、吡蟲啉、氰氟蟲胺、methomyl、新喹唑啉、拒嗪酮、spinetoram、艾克敵105、螺螨酯、螺甲螨酯、spirotetramat、sulfoxaflor、噻蟲啉、噻蟲嗪、硫雙威、殺蟲隆、1-(3-氯吡啶-2-基)-n-[4-氰基-2-甲基-6-(甲基氨基甲酰基)苯基]-3-{[5-(三氟甲基)-2h-四唑-2-基]甲基}-1h-吡唑-5-甲酰胺、1-(3-氯吡啶-2-基)-n-[4-氰基-2-甲基-6-(甲基氨基甲酰基)苯基]-3-{[5-(三氟甲基)-1h-四唑-1-基]甲基}-1h-吡唑-5-甲酰胺、1-{2-氟-4-甲基-5-[(2,2,2-三氟乙基)亞磺酰基]苯基}-3-(三氟甲基)-1h-1,2,4-三唑-5-胺、(1e)-n-[(6-氯吡啶-3-基)甲基]-n'-氰基-n-(2,2-二氟乙基)乙脒、堅強芽孢桿菌、堅強芽孢桿菌菌株i-1582、枯草芽孢桿菌、枯草芽孢桿菌菌株gb03、枯草芽孢桿菌菌株qst713、金龜子綠僵菌f52、rhizobia。殺真菌劑:腈嘧菌酯、n-[9-(二氯亞甲基)-1,2,3,4-四氫-1,4-亞甲基萘甲叉-5-基]-3-(二氟甲基)-1-甲基-1h-吡唑-4-甲酰胺(benzovindiflupyr、benzodiflupyr)、bixafen、啶酰菌胺、多菌靈、萎銹靈、chenopodiumquinoasaponins、百菌清、copper、環唑醇、醚唑、醚菌胺、氧唑菌、氟啶胺、氟菌、氟吡菌胺、氟吡菌酰胺、氟嘧菌酯、喹唑菌酮、粉唑醇、fluthianil、氟唑菌酰胺、環戊唑醇、isopyrazam、異丙定、異噻菌胺、代森錳鋅、maneb、mefenoxam、甲霜靈、環戊唑菌、叉氨苯酰胺、腈菌唑、氟唑菌苯胺、吡噻菌胺、啶氧菌胺、丙環唑、甲基代森鋅、丙硫菌唑、唑菌胺酯、sedaxane、戊唑醇、tetraconazole、甲基托布津、福美雙、唑菌醇、肟菌酯、枯草芽孢桿菌、枯草芽孢桿菌菌株gb03、枯草芽孢桿菌菌株qst713、短小芽孢桿菌(bacilluspumilis)、短小芽孢桿菌gb34。甜菜:除草劑:chloridazon、二氯皮考啉酸、cycloxydim、desmedipham、ethofumesate、吡氟禾草靈、lenacil、metamitron、phenmedipham、quinmerac、quizalofop、tepraloxydim、野麥畏(triallate)、氟胺黃隆(triflusulfuron)。殺蟲劑:吡蟲清、涕滅威、艾扎丁、噻蟲胺、dinetofuran、溴氰菊酯、蟲螨威、chlorantraniliprole(rynaxypyr)、cyantraniliprole(cyazypyr)、(β-)氟氯氰菊酯、γ-氯氟氰菊酯、λ-氯氟氰菊酯、氯氰菊酯、銳勁特、fluensulfone、氟吡菌酰胺、flupyradifurone、imicyafos、吡蟲啉、氰氟蟲胺、滅蟲威、拒嗪酮、新喹唑啉、螺蟲乙酯、sulfoxaflor、七氟菊酯、噻蟲啉、噻蟲嗪、1-(3-氯吡啶-2-基)-n-[4-氰基-2-甲基-6-(甲基氨基甲酰基)苯基]-3-{[5-(三氟甲基)-2h-四唑-2-基]甲基}-1h-吡唑-5-甲酰胺、1-(3-氯吡啶-2-基)-n-[4-氰基-2-甲基-6-(甲基氨基甲酰基)苯基]-3-{[5-(三氟甲基)-1h-四唑-1-基]甲基}-1h-吡唑-5-甲酰胺、1-{2-氟-4-甲基-5-[(2,2,2-三氟乙基)亞磺酰基]苯基}-3-(三氟甲基)-1h-1,2,4-三唑-5-胺、(1e)-n-[(6-氯吡啶-3-基)甲基]-n'-氰基-n-(2,2-二氟乙基)乙脒、堅強芽孢桿菌、堅強芽孢桿菌菌株i-1582、枯草芽孢桿菌、枯草芽孢桿菌菌株gb03、枯草芽孢桿菌菌株qst713、金龜子綠僵菌f52。殺真菌劑:腈嘧菌酯、n-[9-(二氯亞甲基)-1,2,3,4-四氫-1,4-亞甲基萘甲叉-5-基]-3-(二氟甲基)-1-甲基-1h-吡唑-4-甲酰胺(benzovindiflupyr、benzodiflupyr)、bixafen、多菌靈、百菌清、copper、環唑醇、醚唑、氧唑菌、fefenoxam、苯銹啶、丁苯嗎啉、氟吡菌酰胺、氟硅唑、fluthianil、粉唑醇、氟唑菌酰胺、hymexazol、環戊唑醇、isopyrazam、醚菌酯、代森錳鋅、maneb、甲霜靈、腈菌唑、氟唑菌苯胺、丙氯靈、丙環唑、丙硫菌唑、戊唑醇、唑菌胺酯、喹氧靈、sedaxane、sulphur、tetraconazole、甲基托布津、福美雙、肟菌酯、堅強芽孢桿菌、堅強芽孢桿菌菌株i-1582、枯草芽孢桿菌、枯草芽孢桿菌菌株gb03、枯草芽孢桿菌菌株qst713。蕓苔:除草劑:clethodim、二氯皮考啉酸、氯甲草、ethametsulfuron、吡氟禾草靈、草銨膦、草甘膦、metazachlor、quinmerac、quizalofop、tepraloxydim、氟樂靈。殺真菌劑/pgrs:腈嘧菌酯、n-[9-(二氯亞甲基)-1,2,3,4-四氫-1,4-亞甲基萘甲叉-5-基]-3-(二氟甲基)-1-甲基-1h-吡唑-4-甲酰胺(benzovindiflupyr、benzodiflupyr)、bixafen、啶酰菌胺、多菌靈、萎銹靈、chlormequat-chloride、coniothryriumminitans、環唑醇、環丙嘧啶、醚唑、dimethomorph、醚菌胺、氧唑菌、唑酮菌、氟啶胺、氟菌、氟吡菌胺、氟吡菌酰胺、氟嘧菌酯、喹唑菌酮、氟硅唑、fluthianil、粉唑醇、氟唑菌酰胺、異丙定、isopyrazam、mefenoxam、mepiquat-chloride、甲霜靈、環戊唑菌、叉氨苯酰胺、paclobutrazole、氟唑菌苯胺、吡噻菌胺、啶氧菌胺、丙氯靈、丙硫菌唑、唑菌胺酯、sedaxane、戊唑醇、tetraconazole、甲基托布津、福美雙、唑菌醇、肟菌酯、堅強芽孢桿菌、堅強芽孢桿菌菌株i-1582、枯草芽孢桿菌、枯草芽孢桿菌菌株gb03、枯草芽孢桿菌菌株qst713,短小芽孢桿菌,短小芽孢桿菌菌株gb34。殺蟲劑:吡蟲清、涕滅威、艾扎丁、蟲螨威、chlorantraniliprole(rynaxypyr)、噻蟲胺、cyantraniliprole(cyazypyr)、(β-)氟氯氰菊酯、γ-氯氟氰菊酯、λ-氯氟氰菊酯、氯氰菊酯、溴氰菊酯、樂果、dinetofuran、ethiprole、氟啶蟲酰胺、flubendiamide、fluensulfone、氟吡菌酰胺,flupyradifurone、tau-fluvalinate、imicyafos、吡蟲啉、氰氟蟲胺、滅蟲威、拒嗪酮、新喹唑啉、spinetoram、艾克敵105、螺蟲乙酯、sulfoxaflor、噻蟲啉、噻蟲嗪、1-(3-氯吡啶-2-基)-n-[4-氰基-2-甲基-6-(甲基氨基甲酰基)苯基]-3-{[5-(三氟甲基)-2h-四唑-2-基]甲基}-1h-吡唑-5-甲酰胺、1-(3-氯吡啶-2-基)-n-[4-氰基-2-甲基-6-(甲基氨基甲酰基)苯基]-3-{[5-(三氟甲基)-1h-四唑-1-基]甲基}-1h-吡唑-5-甲酰胺、1-{2-氟-4-甲基-5-[(2,2,2-三氟乙基)亞磺酰基]苯基}-3-(三氟甲基)-1h-1,2,4-三唑-5-胺、(1e)-n-[(6-氯吡啶-3-基)甲基]-n'-氰基-n-(2,2-二氟乙基)乙脒、堅強芽孢桿菌、堅強芽孢桿菌菌株i-1582、枯草芽孢桿菌、枯草芽孢桿菌菌株gb03、枯草芽孢桿菌菌株qst713、金龜子綠僵菌f52。本文進一步公開的是從本文公開的包含本發明的hppd基因的棉花植物或種子獲得的纖維或油類。本文進一步公開的是紗、織物或充填劑,其包含本文公開的纖維以及包含本發明的種子的至少部分的粉。在另一方面,本申請公開了用于獲得對至少1x田間劑量的至少一種hppd抑制劑耐受的棉花植物或植物細胞的方法,其包括引入嵌合基因,所述嵌合基因包含(a)編碼具有hppd活性的蛋白質的核酸序列,其中所述蛋白質在對應于seqidno:19的位置336的位置上具有色氨酸,其中所述蛋白質向所述植物提供對至少1x田間劑量的至少一種hppd抑制劑的耐受性,其有效連接(b)植物可表達的啟動子和任選地翻譯終止和多腺苷酸化區。所述方法還包括從植物細胞培養植物。本文還公開用于在棉花植物附近或在本發明的棉花植物的田地上控制雜草的方法,其包括向所述棉花植物的附近或棉花植物田地上應用至少1x田間劑量的至少一種hppd抑制劑。在特定實施方案中,所述至少一種hppd抑制劑在出苗前應用并且在同一種抑制劑或另外至少一種hppd抑制劑在出苗后應用。例如,可用至少1x出苗前劑量的例如ift,隨后用至少1x出苗后劑量的相同的或另外的hppd抑制劑,例如mst或tbt處理植物或植物田地。如本文所用,術語“雜草”指例如田地上不想要的植物或除有意種植植物外的植物,其不想要地在目的植物之間生長并且可能抑制所述目的植物的生長和發育。術語“控制雜草”因此包括抑制雜草生長和殺死雜草。目的植物附近包括其周圍的區域,特別是植物所有生長階段的根覆蓋的區域。通過在棉花田地或棉花植物附近噴灑包含所述hppd抑制劑的溶液向棉花田地或棉花植物附近應用至少一種hppd抑制劑,以便獲得想要的田間劑量的所述至少一種hppd抑制劑的濃度。在特定實施方案中,所述至少一種hppd抑制劑在出苗前應用并且同一種抑制劑或另外至少一種hppd抑制劑在出苗后應用。例如,可用至少1x出苗前劑量的ift和至少1x出苗后劑量的相同的或另外的hppd抑制劑,如mst處理植物或植物田地。在一個實例中,至少一種hppd抑制劑以對所述雜草有毒的劑量應用。該劑量一般是1x,但在個別情況下可以更高,如1,5x、2x、4x或甚至更高。如上文早已說明,對應于1x的劑量率可以根據許多因素而變化。在任何情況下,對于給定情況的劑量率可以增加。如本文所用,“對雜草有毒的”hppd抑制劑的量或濃度與術語“有效量”或“有效濃度”可互換使用。“有效量”和“有效濃度”是足以殺死或抑制雜草生長的量或濃度,但所述量不殺死或嚴重抑制本文公開的棉花植物、植物組織、植物細胞和種子的生長。通常,有效量的至少一種hppd抑制劑是常規用于農業生產系統以殺死目的雜草的量,其表述為“田間劑量”。在該方面,對于棉花,1x的田間劑量根據上文已經示例的所選hppd抑制劑而變化。在一個實例中,所述至少一種hppd抑制劑以至少1.5x的田間劑量應用。在其他實例中,所述至少一種hppd抑制劑以至少2x的田間劑量應用。在仍另一實例中,所述至少一種hppd抑制劑以至少3x或至少4x的田間劑量應用。所述至少一種hppd抑制劑可以在出苗后,也可以出苗前,或出苗后和出苗前應用。本文還公開的是產生包含嵌合基因的棉花種子或棉花纖維的方法,所述嵌合基因包含(a)編碼具有hppd活性的蛋白質的核酸序列,其中所述蛋白質在對應于seqidno:19的位置336的位置上具有色氨酸,其中所述蛋白質向從所述種子生長的棉花植物提供對至少1x田間劑量的至少一種hppd抑制劑的耐受性,所述核酸序列有效連接(b)植物可表達的啟動子;和任選地(c)翻譯終止和多腺苷酸化區,所述方法包括提供本文公開的植物或通過上文所述方法獲得的植物,其中所述植物產生所述種子并且所述嵌合基因包含在所述種子中;并從所述棉花植物中分離所述種子或所述纖維。在一個實例中,具有hppd活性的所述蛋白質與seqidno:21具有至少95%的序列同一性,包含seqidno:21或由seqidno:19組成。具有hppd功能的蛋白質的其他合適的實例公開于本說明書的其他地方。本申請還公開了用于生產包含加工本文公開的纖維的織物的方法。進一步公開的是本文公開的嵌合基因,如這樣的嵌合基因用于(i)在棉花植物中獲得對至少1x田間劑量的至少一種hppd抑制劑的耐受性,或(ii)產生對至少1x田間劑量的至少一種hppd抑制劑的耐受的棉花植物的用途,所述嵌合基因包含(a)編碼具有hppd活性的蛋白質的核酸序列,其中所述蛋白質在對應于seqidno:19的位置336的位置上具有色氨酸,其中所述蛋白質向植物提供對至少1x田間劑量的至少一種hppd抑制劑的耐受性,所述核酸序列有效連接(b)植物可表達的啟動子;和任選地(c)翻譯終止和多腺苷酸化區。本文還公開的是本文公開的棉花纖維用于生產紗、織物或填充劑材料的用途。本文進一步公開的是本文公開的種子,如棉花種子用于生產種子油或膳食的用途。嵌合基因是通過有效連接不相關基因的片段或其他核酸序列構建的人工基因。換言之,“嵌合基因”表示通常不見于植物種類的基因或指任一基因,其中該基因的啟動子或一個或多個其他調節區在自然界中不與部分或全部轉錄的核酸相結合,即對轉錄核酸而言是異源。更特別地,嵌合基因是人工的,即非天然存在的基因,其通過待轉錄的核酸序列的有效連接產生。術語“異源”指來自不同來源的兩條或多條核酸或蛋白質序列之間的關系。例如,啟動子對于有效連接的核酸序列,如編碼序列是異源的,如果該組合不通常見于自然界中。此外,特定序列對于其插入的細胞或生物而言可以是“異源的”(即不天然存在于該特定細胞或生物中)。例如,本文公開的嵌合基因是異源核酸。表述“有效連接”表示,嵌合基因的所述元件彼此以這樣的方式連接,它們的功能是協調的并且允許表達編碼序列,即它們是在功能上相連接的。例如,當啟動子能夠保證所述其他核苷酸序列的轉錄和最終表達時,其與另一核苷酸序列在功能上相接。兩個蛋白質編碼核苷酸序列,例如轉運肽編碼核酸序列和編碼具有hppd活性的蛋白質的核酸序列彼此在功能上連接或有效連接,如果它們以可以形成第一個蛋白質和第二個蛋白質或多肽的融合蛋白質的方式相互連接。如本文所用,“包含”將被解釋為詳細說明如提及的陳述的特征、整數、步驟或組分的存在,但是并不排除一個或多個特征、整數、步驟或組分、或其組的存在或添加。因此,例如包含核苷酸或氨基酸的序列的核酸或蛋白質可以包含比實際引用的序列更多的核苷酸或氨基酸,即被包埋在更大的核酸或蛋白質中。包含功能上或結構上定義的dna區域的嵌合基因可以包含額外的dna區域等。如本文所用,“植物部分”包括任何植物器官或植物組織,包括但不限于果實、種子、胚、分生組織區、愈傷組織、葉、根、芽、花、配子體、孢子體、花粉和小孢子。出于本發明的目的,兩條相關的核苷酸或氨基酸序列的“序列同一性”(表述為百分比)指在這兩條最佳比對序列中的具有相同殘基的位置的數目(x100)除以所比較的位置的數目。空位(即比對中的位置,其中殘基在一條序列中存在但是在另一條序列中不存在)被視為具有不相同殘基的位置。通過needleman和wunsch算法進行兩條序列的最佳比對(needleman和wunsch1970)。可以使用標準軟件程序,如是wisconsin軟件包版本10.1部分的gap(geneticscomputergroup,madison,wisconsin,usa),使用具有空位產生罰分50和空位延伸罰分3的默認評分矩陣)方便地進行以上計算機輔助序列比對。核酸可以是dna或rna,單鏈或雙鏈的。可以化學合成或通過在體外或甚至在體內的生物表達產生核酸。可以使用適當保護的核糖核苷亞磷酰胺和常規的dna/rna合成儀來化學合成核酸。rna合成試劑的供應商是proligo(hamburg,germany)、dharmaconresearch(lafayette,co,usa)、piercechemical(perbioscience的部門,rockford,il,usa)、glenresearch(sterling,va,usa)、chemgenes(ashland,ma,usa)和cruachem(glasgow,uk)。與本公開內容的嵌合基因相關的是,dna包括cdna和基因組dna。如本文所用,術語“蛋白質”或“多肽”描述由多于30個氨基酸組成的分子組,然而術語“肽”描述由高達30個氨基酸組成的分子。蛋白質和肽可以進一步形成二聚體、三聚體和更多低聚體,即由多于一個(多)肽分子組成。形成此類二聚體、三聚體等的蛋白質或肽分子可以相同或不相同。相應的高級結構因此稱為同二聚體或異二聚體、同三聚體或異三聚體等。術語“蛋白質”和“肽”也指天然修飾的蛋白質或肽,其中例如通過糖基化、乙酰化、磷酸化等實現所述修飾。此類修飾為本領域所熟知。將清楚的是,當rna分子的核苷酸序列通過參考相應的dna分子的核苷酸序列進行定義時,核苷酸序列中的胸腺嘧啶(t)應該被尿嘧啶(u)所替換。參考rna還是dna分子從本申請的上下文中將會是清楚的。以下非限制性實施例描述了重新設計的大范圍核酸酶將目的dna引入到緊靠ghb119原種事件的用途和包含賦予對至少1x田間劑量的至少一種hppd抑制劑的耐受性的hppd基因的棉花植物的產生。除非在實施例中另行說明,所有的重組dna技術是根據如描述于sambrook(薩姆布魯克)等人,(1989)molecularcloning:alaboratorymanual,secondedition(《分子克隆:實驗室手冊》,第二版),coldspringharborlaboratorypress(冷泉港實驗室出版社),紐約和在ausubel(奧蘇貝爾)等人,(1994)currentprotocolsinmolecularbiology,currentprotocols,usa(《美國,當前技術方案,分子生物學的當前技術方案》)中卷1和卷2的標準方案來進行的。用于植物分子工作的標準材料和方法描述于由r.d.d.croy(克羅伊)撰寫的plantmolecularbiologylabfax(《植物分子生物學實驗》)(1993)中,由biosscientificpublicationsltd(uk)(bios科學出版有限公司(英國))和blackwellscientificpublications,uk(布萊克威爾科學出版物,英國)聯合出版。其他用于標準分子生物技術的參考文獻包括sambrook(薩姆布魯克)和russell(拉塞爾)(2001),molecularcloning:alaboratorymanual,thirdedition(《分子克隆:實驗手冊,第三版》),冷泉港實驗室出版社,紐約,brown(布朗),molecularbiologylabfax,secondedition,(《分子生物學實驗,第二版》),學術出版社(英國)(1998)。用于聚合酶鏈式反應的標準材料和方法可以在dieffenbach(迪芬巴赫)和dveksler(德福柯思樂)(1995),pcrprimer:alaboratorymanual,(《pcr引物:實驗室手冊》)coldspringharborlaboratorypress(冷泉港實驗室出版社)(1995)中,以及在mcpherson(麥克弗森)等人(2000),pcr-basics:frombackgroundtobench,firstedition,(《pcr基礎:從背景到平臺,第一版》)(2000)springerverlag(施普林格),德國中找到。在此提到的所有專利、專利申請和出版物,都出于所有目的通過引用以其全文并入本文。在說明書和實施例中,參考以下序列:seqidno.1:bay-5/6大范圍核酸酶的識別序列(正義)seqidno.2:bay-5/6大范圍核酸酶的識別序列(seqidno.1的反向互補序列)seqidno.3:含有bay-5/6識別位點的ghb119的3’側翼序列seqidno.4:ghb119的5’側翼序列seqidno.5:大范圍核酸酶表達載體pcv193seqidno.6:cot-5/6單鏈大范圍核酸酶的氨基酸序列seqidno.7:修復dna載體pcv211seqidno.8:pf-hppd(w336)的氨基酸序列seqidno.9:2mepsps的氨基酸序列seqidno.10:cry2a探針的核苷酸序列seqidno.11:hppd探針的核苷酸序列seqidno.12:2mepsps探針的核苷酸序列seqidno.13:pcr引物ib527seqidno.14:pcr引物ib616seqidno.15:pcr引物ib617seqidno.16:i-crei的氨基酸序列seqidno.17:pcr引物ib589seqidno.18:pcr引物vds406seqidno.19:野生型(wt)熒光假單胞菌hppd蛋白質的氨基酸序列seqidno.20:載體ptif16的t-dna的核酸序列,包含本發明的嵌合基因seqidno.21:編碼熒光假單胞菌hppd蛋白質的氨基酸序列,其中氨基酸位置336上的甘氨酸已經被色氨酸替換seqidno.22:玉米的2mepsps蛋白質的氨基酸序列seqidno.23:載體ptsih09的t-dna的核酸序列,包含本發明的嵌合基因在命名為“bcs11-2012-wo_st25”的105千字節(在microsoft中測定大小)的文件中含有的序列表含有從seqidno.1至seqidno:23的23條序列,通過電子投遞而被提交并并入本文作為參考。實施例本文描述的所有重新設計的大范圍核酸酶及其表達載體已經由precisionbiosciencesinc.,104t.w.alexanderdrive,researchtrianglepark,nc27713設計。實施例1:載體構建使用標準重組dna技術,構建以下dna載體,其包含以下有效連接的元件(在圖3中概述地描繪):修復dna載體pcv211(seqidno:7)●nt2252-4310:ghb119上游cot-5/6識別位點:cot-5/6識別位點上游的事件ghb119的3'側翼基因組dna。●4354-4771:p35s2c:p35s2啟動子序列。●nt4772-4840:5'cab22l:包括矮牽牛的葉綠素a/b結合蛋白基因的前導序列的序列(harpster等,1988)。●nt4842-5213:tpotpy-1pa:優化的轉運肽衍生物(位置55改變為tyr)的編碼序列,含有玉米(玉米)和向日葵(helianthusannuus)(向日葵)的rubisco小亞基基因的序列,適合于棉花密碼子選擇。●nt5214-6290(incl終止):hppdpfw336-1pa:通過用色氨酸替換氨基酸甘氨酸336而修飾的熒光假單胞菌菌株a32的4-羥苯基丙酮酸雙加氧酶基因的編碼序列(boudec等,1999),適合于棉花密碼子選擇。●nt6316-6976:3'histonat:包括擬南芥組蛋白h4基因的3′非翻譯區的序列(chabouté等,1987)。●nt7060-7976:ph4a748:包括擬南芥組蛋白h4基因的啟動子區的序列(chabouté等,1987)。●nt8026-8488:intron1h3at:擬南芥組蛋白h3.iii變體的基因ii的第一個內含子(chaubet等,1992)。●nt8495-8866:tpotpc:優化的轉運肽的編碼序列,含有玉米(玉米)和向日葵(向日葵)的rubisco小亞基基因的序列(lebrun等,1996)。●nt8867-10204:2mepsps:玉米的雙突變型5-烯醇式-丙酮酰莽草酸-3-磷酸合酶基因的編碼序列(lebrun等,1997)。●nt10228-10888:3'histonat:包括擬南芥組蛋白h4基因的3′非翻譯區的序列(chabouté等,1987)。nt10971-12521:fgdghb119:下游cot-5/6識別位點:cot-5/6識別位點下游的事件ghb119的3'側翼基因組dna。cot-5/6大范圍核酸酶表達載體pcv193(seqidno:5)●nt2241-2599:p35s2c(片段):thep35s2c啟動子片段。●nt2602-3083:p35s2c:p35s啟動子序列。●nt3091-4169:cot-5/6-sc:cot-5/6單鏈大范圍核酸酶編碼dna區。●nt4173-4433:3'nos:包括來自ptit37的t-dna的胭脂堿合酶基因的。實施例2:培養基和緩沖液如下文在實施例3和4中所述,在胚胎發生愈傷組織生成和轉化期間使用的培養基和緩沖液:共培養基質:具有1/2濃度ms鹽的m100,+100μmas+100mg/ll-半胱氨酸(l-半胱氨酸總是新鮮配制并且高壓滅菌后添加),ph=5.2m100基質:ms鹽、b5維生素、mes0.5g/l、mgcl2.6h2o0.94g/l、gelrite2g/l、葡萄糖30g/l,ph5.8100q基質:m100基質+0.2m甘露醇+0.2m山梨醇,ph5.8m104基質:=m100基質+1g/lkno3,ph5.8m700基質:stewarts鹽+維生素、mgcl2.6h2o0.47g/l、gelrite1g/l、植物瓊脂2.25g/l、蔗糖20g/l、ph6.8m702基質:stewarts鹽+維生素、mgcl2.6h2o0.71g/l、gelrite1.5g/l、植物瓊脂5g/l、蔗糖5g/l、ph6.8ac:活性碳2g/las:乙酰丁香酮實施例3:脆弱的胚胎發生愈傷組織的生成在黑暗中在26-28℃將來自coker312的棉花種子在不含激素的固體萌發培養基m100上萌發7-10天。接下來,通過將來自幼苗的下胚軸外植體在固體培養基m100(不含激素)上溫育來進行胚胎發生愈傷組織的誘導。約2個月后,當在下胚軸的剖面處損傷的愈傷組織開始顯示出快速增殖時,為了富集并且維持胚胎發生愈傷組織的進一步傳代培養在具有活性碳(2g/l)的固體m100培養基上進行。在26-28℃胚胎發生愈傷組織的誘導和維持發生在暗光條件下(強度:1-7μmolm-2sec-1;光周期:16h光/8h黑暗)。(基本上如美國臨時申請61/493579和ep11004570.5中所述,并入本文作為參考,特別是第31-35頁,實施例2-5)。實施例4:通過粒子轟擊轉化棉花胚胎發生愈傷組織遵循以下程序來使用粒子轟擊轉化棉花胚胎發生愈傷組織●在轟擊之前,將向其中引入dsb誘導的靶向修飾的靶株系的實施例3的脆弱棉花胚胎發生愈傷組織(ec)在傳代培養后2-3周進行收集,并且通過büchnerfilter,在具有0.2m甘露醇和0.2m山梨醇的100q基質上的濾紙頂部上以薄層進行平板培養約4-約6小時。●在100q基質上預質壁分離(preplasmolysis)約4-約6小時后,用0.5pmolpcv211+0.5pmolpcv193或0.75pmolpcv211+0.5pmolpcv193或0.75pmolpcv211+0.75pmolpcv193轟擊ec。●轟擊條件:○金顆粒直徑:0.3-3μm○爆破薄膜:1350psi○距靶組織的距離:9cm○真空室約27(按hg計)○bioradpps_1000/he生物彈射粒子遞送系統●轟擊后,將濾器轉移至具有0.2m甘露醇的m100基質上或者不含選擇劑的m100基質上。●在26-28℃在暗光條件下在非選擇性基質上1-4天后,將濾器轉移至具有1mm草甘膦的選擇性m100基質上。●約2-3周后,增殖愈傷組織選自濾器,并且進一步作為小堆傳代培養至具有1mm草甘膦的選擇性m100基質上。按約每3周一次的傳代培養間隔進行約6周的傳代培養時期后,在26-28℃在暗光條件下在選擇性m100基質上,可以選擇轉化的ec/體細胞胚。●用于鑒定靶向修飾事件的基于pcr分析的分子篩選在轉化的ec/體細胞胚水平上進行。●在26-28℃,在光條件下(強度:40-70μmolm-2sec-1;光周期:16h光/8h黑暗),通過將ec/體細胞胚在具有活性碳(ac)和相應選擇劑的m104上進行平板培養,從靶向修飾事件啟動植物再生。●約一個月之后,將0.5-1cm的單獨胚轉移至具有ac和1mm草甘膦的m104上的濾紙頂部上。●在26-28℃,在光條件下(強度:40-70μmolm-2sec-1;光周期:16h光/8h黑暗),將進一步良好萌發的胚轉移至非選擇性萌發基質m702上。●一至兩個月后,在26-28℃在光條件下(強度:40-70μmolm-2sec-1;光周期:16h光/8h黑暗),將進一步發育的胚轉移至m700基質上,用于發育成小苗。(基本上如美國臨時申請61/493579和ep11004570.5中所述,并入本文作為參考,特別是第31-35頁,實施例2-5)實施例5:通過同源重組緊靠ghb119轉基因事件靶向修飾基因組區域使用如實施例4中所述的粒子轟擊將pcv211修復dna載體和pcv193cot-5/6大范圍核酸酶編碼載體引入半合子ghb119植物的胚胎發生愈傷組織中(如在wo2008/151780所述)。首先,篩選轉化體的草甘膦抗性,因為這表明了修復dnapcv211的插入。隨后利用引物對ib527xib616(分別為seqidno.13和14,概述地表示于圖3),使用來自invitrogen的最終mgcl2濃度為2mm的elongase酶混合物和以下循環參數:在94℃下2分鐘變性,隨后是35個循環的94°-30秒鐘,58°-30秒鐘,68°-9分鐘,和68℃下7分鐘的最終延伸步驟,通過pcr使這些進行高通量分子分析,以鑒定可能正確的基因靶向事件。9640bp的pcr產物的存在表明通過至少一側同源重組(通過如圖3中連譜號所示的2072bp同源區域)的正確疊加基因靶向事件。可能正確的疊加還可以通過利用引物對ib527xib617(分別為seqidno13和17)通過pcr進行鑒定,其然后應該產生3893bp的pcr產物(圖3)。隨后的pcr產物的序列分析允許驗證至少一側正確的同源重組。因為pcr產物的缺失也可以起因于差的dna質量或靶位點上大的插入或缺失,所以也可以在嚴謹條件下使用分別識別epsps基因(seqidno9)、cry2ae基因(seqidno10)和hppd基因(seqidno11)的探針,在draiii/stui消化的基因組dna上通過dna印跡來分析pcr陽性或陰性的大量草甘膦抗性愈傷組織。如果有正確的疊加事件,那么使用所有三種探針應該導致11kb帶的鑒定(也參見圖3)。使用pcr已經鑒定了總計至少27個推測的正確疊加事件(總計1479個鑒定的glyr事件的約1.8%),其中至少13個已經被如上所述的dna印跡驗證為正確的基因靶向事件。評價一些驗證的疊加事件其原始ghb119事件的bar和cry2ae基因的功能性。通過leafstrip測試,pat和cry2ae蛋白質的表達可以在那些事件中得以驗證。此外,pcr片段ib527xib617和ib589xvds406(分別為seqidno.17和seqidno.18)(其橫跨兩個重組位點(參見圖3))的序列分析顯示在靶基因座上epsps和hppd的正確插入。實施例6:傳遞疊加至下一代為了測試所有四個轉基因是否的確作為疊加傳遞到下一代并且不獨立地分離,評價包含疊加的植物與野生型植物的雜交后代中轉基因的存在。表2:靶向插入事件(即對疊加事件為雜合的)與野生型植物的雜交分離數據產生約50%wt和約50%4基因。epsphppdcry2aebarwtwtwtwt1個拷貝1個拷貝1個拷貝1個拷貝2個拷貝2個拷貝2個拷貝2-3個拷貝1個拷貝1個拷貝1個拷貝1個拷貝1個拷貝1個拷貝1個拷貝1個拷貝2個拷貝2個拷貝2個拷貝2個拷貝wtwtwtwtwtwtwtwt1個拷貝1個拷貝1個拷貝1個拷貝1個拷貝1個拷貝1個拷貝1個拷貝1個拷貝1個拷貝1個拷貝1個拷貝wtwtwtwt1個拷貝1個拷貝1個拷貝1個拷貝1個拷貝1個拷貝失敗1個拷貝表3:自交后代(即對疊加事件為純合的)與野生型植物的分離數據產生約25%wt和約75%4基因。因此,表2和表3顯示所述疊加事件的確作為單個遺傳單位遺傳。實施例7:疊加中的轉基因的表達和功能性使用q-pcr分析和蛋白質印跡評價大量疊加事件的轉基因表達。觀察到所有四個轉基因的表達,雖然有時候具有不同的表達水平(其在兩種檢測方法之間是相關的)。同樣,在溫室來自疊加事件的后代中評價對hppd抑制劑型除草劑的耐受性。植物在2xtbt處理后展示一些輕度褪色,但隨后恢復。實施例8:生成包含嵌合hppd基因的棉花植物使用常規重組dna技術構建ptif16和ptsih09t-dna表達載體,兩者均包含分別處于35s和csvmv啟動子控制下的hppd編碼嵌合基因,和epsps編碼嵌合基因,具有以下有效連接的dna片段:ptif16(seqidno.20)-hppd嵌合基因:a)p35s2:包括花椰菜花葉病毒35s轉錄物的啟動子區的序列(odell等,1985):seqidno:20的nt位置88-506b)5'cab22l:包括矮牽牛的葉綠素a/b結合蛋白基因的前導序列的序列(harpster等,1988):seqidno:20的nt位置507-576c)tpotpy-1pa:含有玉米(玉米)和向日葵(向日葵)的rubisco小亞基基因的序列的優化轉運肽衍生物(位置55改變成tyr)的編碼序列(lebrun等,1996),適用于棉花密碼子選擇:seqidno:20的nt位置577-948d)hppdpfw336-1pa:通過用色氨酸替換氨基酸甘氨酸336而修飾的熒光假單胞菌菌株a32的4-羥苯基丙酮酸雙加氧酶基因的編碼序列(boudec等,2001),適用于棉花密碼子選擇:seqidno:20的nt位置949-2025(編碼見于nt1954-1956的位置336上的色氨酸的密碼子)e)3'histonat:包括擬南芥組蛋白h4基因的3’非翻譯區的序列(chabouté等,1987):seqidno:20的nt位置2026–2714-epsps嵌合基因:a)ph4a748abc:包括擬南芥組蛋白h4基因的啟動子區的序列(chabouté等,1987):seqidno:20的nt位置2791-3750b)內含子1h3at:擬南芥組蛋白h3.iii變體的基因ii的第一個內含子(chaubet等,1992):seqidno:20的nt位置3751-4229c)tpotpc:優化的轉運肽的編碼序列,含有玉米(玉米)和向日葵(向日葵)的rubisco小亞基基因的序列(lebrun等,1996):seqidno:20的nt位置4230–4601d)2mepsps:玉米(玉米)的雙突變型5-烯醇式-丙酮酰莽草酸-3-磷酸合酶基因的編碼序列(lebrun等,1997):seqidno:20的nt位置4602–5939e)3'histonat:包括擬南芥組蛋白h4基因的3′非翻譯區的序列(chabouté等,1987):seqidno:20的nt位置5940–6630ptsih09(seqidno.23)-hppd嵌合基因:a)pcsvmvxyz:包括木薯葉脈花葉病毒的啟動子區的序列(verdaguer等,1996):seqidno:23的nt位置2735-2223b)tpotpy-1pa:含有玉米(玉米)和向日葵(向日葵)的rubisco小亞基基因的序列的優化轉運肽衍生物(位置55改變成tyr)的編碼序列(lebrun等,1996),適用于棉花密碼子選擇:seqidno:23的nt位置2214-1845c)hppdpfw336-1pa:通過用色氨酸替換氨基酸甘氨酸336而修飾的熒光假單胞菌菌株a32的4-羥苯基丙酮酸雙加氧酶基因的編碼序列(boudec等,2001),適用于棉花密碼子選擇:seqidno:23的nt位置1842-766d)3'histonat:包括擬南芥組蛋白h4基因的3’非翻譯區的序列(chabouté等,1987):seqidno:23的nt位置749-83-epsps嵌合基因:a)ph4a748abc:包括擬南芥組蛋白h4基因的啟動子區的序列(chabouté等,1987):seqidno:23的nt位置2834-3750b)內含子1h3at:擬南芥組蛋白h3.iii變體的基因ii的第一個內含子(chaubet等,1992):seqidno:23的nt位置3790-4255c)tpotpc:優化的轉運肽的編碼序列,含有玉米(玉米)和向日葵(向日葵)的rubisco小亞基基因的序列(lebrun等,1996):seqidno:23的nt位置4269-4640d)2mepsps:玉米(玉米)的雙突變型5-烯醇式-丙酮酰莽草酸-3-磷酸合酶基因的編碼序列(lebrun等,1997):seqidno:23的nt位置4641-5978e)3'histonat:包括擬南芥組蛋白h4基因的3′非翻譯區的序列(chabouté等,1987):seqidno:23的nt位置5999-6665將t-dna載體ptif16和ptsih09引入到根癌農桿菌c58c1rif(peha101)中并根據本領域已知的方法使用壯觀霉素和鏈霉素選擇轉化體。根據本領域已知的方法使用農桿菌菌株轉化棉花品種“coker312”并且在體外選擇轉基因植物對草甘膦(1.0-1.5mm)的耐受性并使用rt-pcr分析拷貝數。使含有轉基因的t0代植物自交并將所得t1代用于溫室中的除草劑耐受性測試。實施例9:在溫室中評估除草劑耐受性為了分析除草劑耐受性,將ptif16和ptsih09事件的100粒種子的t1分離群體播種在溫室中。在生長期用不同田間劑量的不同hppd抑制劑處理出苗植物。在13和24dpa(應用后天數),評分植物在0-100范圍內的表型(“損傷分數”),其中0代表沒有損傷(即,對應于未處理的野生型植物),而100代表最大損傷(即在所有地上部分展示損傷)(表4)。實施例10:在田地中評估除草劑耐受性在美國兩個地方即california(ca)和tennessee(tn)進行除草劑耐受性的田地試驗,每個地方兩次重復。將在coker312背景中對ptif16事件純合的植物與對照植物(來自同一株系或coker312植株的wt分離體)一起播種在約40株植物的一塊地中并在2-4葉期用商業濃度(至少1x)的廣譜hppd抑制劑型除草劑以出苗后處理的方式進行處理(除了在ca,在出苗前應用,而在tn,在出苗后應用的balanceflex)。處理后7、21和28天(dat)在0-100范圍(其中0對應于沒有萎黃病/褪色/枯斑,而100表示植物死亡)內對植物應答評分(考慮萎黃病、褪色和枯斑的程度)來評價耐受性。將兩個地方的結果平均(表5)。盡管wt植物響應除草劑處理展示明顯的萎黃病、褪色和枯斑,但是對ptif16事件純合的植物對所測試的所有hppdi除草劑都耐受。表5:對包含嵌合熒光假單胞菌(pf)-hddp-336w基因的ptif16事件純合(hom)和單性(azygous)(wt)的棉花植物的植物應答(萎黃病、褪色和枯斑)。*tbt處理ca比其他處理應用早1周,**僅balanceflex處理captif16純合的植物對balanceflexx(異氟草,105ga.i./ha)的出苗前處理也完全耐受。實施例11:田地中的出苗前和出苗后除草劑耐受性與上文類似,在argentina,利用一個ptif16事件和兩個ptshi09事件(純合子)相比于野生型coker312株系進行田地試驗。在處理后7、14和21天(dat),評價對2x和4xbalancepro(ift)和4xcallisto(mst)的出苗前耐受性。測試對2xift、4xift、2xmst、2xtbt、4xtbt2x苯吡唑草酮(top)和4x草甘膦的出苗后耐受性。結果(植物應答)描繪于下文的表6和表7中。表6:反映為植物應答(萎黃病、褪色和枯斑)的田地中出苗前除草劑耐受性。表7:反映為植物應答(萎黃病、褪色和枯斑)的田地中出苗后除草劑耐受性。序列表<110>拜爾作物科學公司拜爾作物科學股份公司bayercropsciencelpd'halluin,kathleendittgen,janjansens,stefanmeulewaeter,frankmoser,halweterings,koenwilde,thomashain,ruedigerbickers,udotrolinder,lindahenniger,gary<120>修飾植物基因組的方法和手段<130>bcs11-2012<160>23<170>patentin版本3.5<210>1<211>22<212>dna<213>人工序列<220><223>ghb119<400>1taaaattatttacaagtgttta22<210>2<211>22<212>dna<213>人工序列<220><223>ghb119<400>2taaacacttgtaaataatttta22<210>3<211>3687<212>dna<213>人工序列<220><223>ghb119<400>3ccagtactaaaatccagatcatgcatggacctgcaggtcgacggccgagtactgttttat60ttttaacaggaatttgagtcacgcaatttctaatacttgttcaaattgatttgaaaaaaa120aaattgaataactcaaataatgcaattcaattaacttgaaattcgaacaattttttattt180ttattttttaaattgaatcaagttttacttacccgtaattatttgaatggatcaagttga240gttaaattcaaccttttttttctttttaagaattggaccataactcaacttagctcgtaa300atatgttgtaaccgttgagccatcagtgtaatattaactaccttgtttgtaactcctaca360caagtcgtatagtctattagcggttgctttgagtaattctatgtttagatgtcatatatc420gtattatattattcgagttttagtgtgattaatgaatataaatacttgtcaacgtagata480tactaaatttttttaggattttatgtattcgggagtcatatatatatatcatattttcat540atttacgaatgagttgaatacgagtttcgaatacatatttaaaagaataaataaaatgtt600ggtaacatagggttggtgggctttgataatgggctaatggcaatggatttgcacatacac660ccctaaccaccttcattttggttgattttttaaatattttttcttgtgtgctgaataaat720aaataaataaataaaacgaatcgaatccatgtgtttggactatctaaaaaatgaaacttt780aacttaatggaattctatttttttttattttatgattaatttaaatttttataatataga840aataagttttttatatttaaaataattaaaataacttaaaatttttatataaaaagtatt900tttttcaaaaaataccattttaaaaagttattaatggttttttatttactttgaaaaatt960attttagaaaaaaatttcaaatttatttttaataattttattattatacattctcattat1020atctgttatttttaacacaatacttaaaatcatctatagtccctctttaactgcgtttca1080acgcactcgaactcacattctcctatattaacgataatgccatactaatcaaattaagac1140ttagtcgacaaaaatacttttaaactttaccattatgaatgaatccaaaaatatatccat1200taaaaaaaattcaacatgctacatgattgtttgagatttgacttagtttgtttttggacc1260ttaacaatcaatcatatctataactatatctttatttagaatttaaagggtttgttttga1320tgacacctaagcatagatataaaatataatttaaagtactgaagttgagaatttactttt1380aaaggcttttcagaaaaaaataaaaaaataaaggattggattattagacaacaatgcaat1440gttattaaattaagtatcgggacgattgagtcttaattagattagtattagtgttgttat1500cagtgtgaaatgatgtgggttcgagtatgctttagagcattatcctcctatttaaggatt1560aggaaggggttatgggtagttttagatattgtatccaaaatagcaaatgatattgatgtt1620caaaaaaaaaattaaaagtttgggattaaatttagaagattatttgtataaattaaaata1680caaaactttttttttatcataattaaggggtatatatcaaaattatatataaattatgat1740ttaatattcaatttgatatacgaactttgattttatacaatttaatacataaatattaaa1800tttagctcaattttcacaaataattaacatcgtactcgatgtagcatcattttaggaaaa1860aaattatatacctaacatgaattttatttgattgatttagtttttaaaatatttacaatt1920aaatcaaaatcaaaattatatatatataattgaaacaaaattatagtttctacattaggc1980aaatcatttataaatatttttgtgttatattttacgtgtttaaaatgatataaaattata2040caaaaacaatattaaatgttaataatttgtttaatatgaattttgagagcactattttta2100tgccgtgcctaattaaaattatttacaagtgtttatgttgatattttaattatgtttcat2160ataaatttgtcgtaatagtttaattgttaaatctaaataaaagtatttaaaaatagatat2220tatattttttatgatatatataaatatcatgaaactttagaatttatggacaaaaacatc2280actatccactagttaatcatttctacaaacgacatttatttatttaaaatggtcagaaaa2340attgtgacctaccaggaatgaggatttgattggagggatgtaaaccaagttgatattttt2400ttctcacttgtgaaaaagatagtgtttggctggaattttatgtatgataaatttgatagg2460gacttgcacttgagattattttctgggttcttcatgcttttataataacttgactatcaa2520attctctctttggattctacgacgttcatatctatttgtatggtggcttcgagtaggaat2580ttttttaaaagtatttgtgtttgatatttacatatgtttttatacaatgtttagaactac2640ttatagtccatttcatcccttaagtaggtacgccttcctacattgacaacaatatcgata2700ctaatcgaactaaaactcaatcaacgcccatattcgacacttcaatttggataataagta2760gagtattttatagatatttgatttatatgtaaatgatttttttaatattttttgagagta2820ttttaaaattcgtgttttattaattttcatattatttagattcttaggattgaaaaaaaa2880tttataaatttgaaaaactctctcgtggaaaagaattacaaaataatttaacccctaata2940aaaatataacaaccatttttttatgtcaatcactacgttattcatcgcatcattacttaa3000cgtatcatcaaagatttgatgtaaaaattaaattcaagggctcaagccatgagagcttca3060atgaaaatattcattgctaacttttgattgaaattttaattgaatataattttttcttgt3120gagcttaacggatattttaaattaccttacaaaaactgtttaaaacttaaaactgtataa3180taataaaacttatattctctaacaacatataaatctataatttgtagtttgtcccctaaa3240cttgtccaacaatatcaaattaatgtctgaattttttatcgagttggtacgtctattcta3300acaccttaatttatgctgatgcgacactaccagccaatcaaatatgacacataataacct3360tcaatatgagatgtgatcaattaagaagtttagaaatctttagaaattatgatttatatt3420cccatcatccccacaaccatatcttacctcttaaaaatatgctttctaaaaatatgattt3480gatttgctgtgtgaaaaaaaaaaggcaaaagaaaatatctaagaaataaatcaataattg3540agagaggtcaaaagacgatattatccttttgatttgctggtctctagaaagcaaaagatg3600ggggattagggtaggcagaataaaaaaaaaaatctagggaaagaagatcccactttaacc3660aatgaacgacagcccacctcattagag3687<210>4<211>1635<212>dna<213>人工序列<220><223>側翼區<400>4ttttaaatcaaaatccgccaaactgctgtcgtctcattggatattacgataatatcatta60atttaattttgaagtattttttgttttcttaaatgtaacgagtaacacaattaaaatata120aatataaatataatacaaatggactaaaaatgggtataatccgaactaacaggattaaaa180ctcacatgataaaatttggaaacctatacgtataattgcctataataagtctcgaactaa240ccctccaaatattggtattatcctttggaatgtattgtaacctcacactaccctaaacat300atcataaggttgtattctatctcttttattcagaaaataaaaacaaattcttatatatca360acatcaaatgttggagacacgtaaatttttaataataaaaattaataaaaattttaacag420aaataattaatttactctttaatttaacatacaaaaattaattttctcgaaaatataatc480tatttcttaaattgcgtgttcattcatcaattcttttcttcaaggtatctcttactcaaa540cgtgtaaagggctaaaagcaactaaaatagctcataagtacaaatgaatcttggatgtac600attgtacagtagaccggtctacggcctggtggcctggcctcgattaaatgggctcattgt660tctggagttaggtttagacccaaattttatattaaataaattttgttttttaattaattt720ataaaataaataaataatagcgtgaaattaaatttcggttttttaaaattttataaataa780taaaatgagttatttttaggggtgagtattttatcaaatcgaatgaaaaatatttgattt840agttcagttaatgagtcatattttatcatcttaactcgttttaaatattttccaaatcaa900gtcgagtcaaataaatttattcaagtcaaattaaaaaataaataaactggtcatattgaa960atcttgttgacgatacgactaaaattcaagttaaaaaaacgcaaatatcatatatatttg1020aacactttttaaaagaaaataggaaaaaatatatggtaattagtatgataaacttatctt1080aataatttatttgttcaagacatcatataatttagcatttttatatattttaaatatttt1140aaaaaataatttaatttttaatatattttataaagaaatttgaaaaatattttataaaac1200taacgagtaaaaatgatccaaagagtatttacatcaatttattattctaattgaatttta1260caattcgaattaaactccaaaaaattgaattgcttattgtaaaattgaacgtcaataatg1320acaatgaatcgcaaagcaagagagaaaaataaaacacacaaaattttacgtggaaaccct1380ttcgagaaaaaatcacaggcagagggaagaaaattctccatgtcaaattcgaatgataca1440agataagagacgattacgtctatttataagtttaaaaaccttattctaatcaaagtcaaa1500tagaagtgatgcagtaagattaaaaactttgtgcagccttcgccctaacagatcccccta1560tcttgctaaatggctccatggcgatcgctctagaggatctgcgatctagtaacatagatg1620acaccgcgcgcgata1635<210>5<211>5000<212>dna<213>人工序列<220><223>bay-5/6表達載體<220><221>cds<222>(3090)..(4169)<400>5gacgaaagggcctcgtgatacgcctatttttataggttaatgtcatgataataatggttt60cttagacgtcaggtggcacttttcggggaaatgtgcgcggaacccctatttgtttatttt120tctaaatacattcaaatatgtatccgctcatgagacaataaccctgataaatgcttcaat180aatattgaaaaaggaagagtatgagtattcaacatttccgtgtcgcccttattccctttt240ttgcggcattttgccttcctgtttttgctcacccagaaacgctggtgaaagtaaaagatg300ctgaagatcagttgggtgcacgagtgggttacatcgaactggatctcaacagcggtaaga360tccttgagagttttcgccccgaagaacgttttccaatgatgagcacttttaaagttctgc420tatgtggcgcggtattatcccgtattgacgccgggcaagagcaactcggtcgccgcatac480actattctcagaatgacttggttgagtactcaccagtcacagaaaagcatcttacggatg540gcatgacagtaagagaattatgcagtgctgccataaccatgagtgataacactgcggcca600acttacttctgacaacgatcggaggaccgaaggagctaaccgcttttttgcacaacatgg660gggatcatgtaactcgccttgatcgttgggaaccggagctgaatgaagccataccaaacg720acgagcgtgacaccacgatgcctgtagcaatggcaacaacgttgcgcaaactattaactg780gcgaactacttactctagcttcccggcaacaattaatagactggatggaggcggataaag840ttgcaggaccacttctgcgctcggcccttccggctggctggtttattgctgataaatctg900gagccggtgagcgtgggtctcgcggtatcattgcagcactggggccagatggtaagccct960cccgtatcgtagttatctacacgacggggagtcaggcaactatggatgaacgaaatagac1020agatcgctgagataggtgcctcactgattaagcattggtaactgtcagaccaagtttact1080catatatactttagattgatttaaaacttcatttttaatttaaaaggatctaggtgaaga1140tcctttttgataatctcatgaccaaaatcccttaacgtgagttttcgttccactgagcgt1200cagaccccgtagaaaagatcaaaggatcttcttgagatcctttttttctgcgcgtaatct1260gctgcttgcaaacaaaaaaaccaccgctaccagcggtggtttgtttgccggatcaagagc1320taccaactctttttccgaaggtaactggcttcagcagagcgcagataccaaatactgttc1380ttctagtgtagccgtagttaggccaccacttcaagaactctgtagcaccgcctacatacc1440tcgctctgctaatcctgttaccagtggctgctgccagtggcgataagtcgtgtcttaccg1500ggttggactcaagacgatagttaccggataaggcgcagcggtcgggctgaacggggggtt1560cgtgcacacagcccagcttggagcgaacgacctacaccgaactgagatacctacagcgtg1620agctatgagaaagcgccacgcttcccgaagggagaaaggcggacaggtatccggtaagcg1680gcagggtcggaacaggagagcgcacgagggagcttccagggggaaacgcctggtatcttt1740atagtcctgtcgggtttcgccacctctgacttgagcgtcgatttttgtgatgctcgtcag1800gggggcggagcctatggaaaaacgccagcaacgcggcctttttacggttcctggcctttt1860gctggccttttgctcacatgttctttcctgcgttatcccctgattctgtggataaccgta1920ttaccgcctttgagtgagctgataccgctcgccgcagccgaacgaccgagcgcagcgagt1980cagtgagcgaggaagcggaagagcgcccaatacgcaaaccgcctctccccgcgcgttggc2040cgattcattaatgcagctggcacgacaggtttcccgactggaaagcgggcagtgagcgca2100acgcaattaatgtgagttagctcactcattaggcaccccaggctttacactttatgcttc2160cggctcgtatgttgtgtggaattgtgagcggataacaatttcacacaggaaacagctatg2220accatgattacgccaagcttgagaggcggtttgcgtattggctagagcagcttgccaaca2280tggtggagcacgacactctcgtctactccaagaatatcaaagatacagtctcagaagacc2340aaagggctattgagacttttcaacaaagggtaatatcgggaaacctcctcggattccatt2400gcccagctatctgtcacttcatcaaaaggacagtagaaaaggaaggtggcacctacaaat2460gccatcattgcgataaaggaaaggctatcgttcaagatgcctctgccgacagtggtccca2520aagatggacccccacccacgaggagcatcgtggaaaaagaagacgttccaaccacgtctt2580caaagcaagtggattgatgtgaacatggtggagcacgacactctcgtctactccaagaat2640atcaaagatacagtctcagaagaccaaagggctattgagacttttcaacaaagggtaata2700tcgggaaacctcctcggattccattgcccagctatctgtcacttcatcaaaaggacagta2760gaaaaggaaggtggcacctacaaatgccatcattgcgataaaggaaaggctatcgttcaa2820gatgcctctgccgacagtggtcccaaagatggacccccacccacgaggagcatcgtggaa2880aaagaagacgttccaaccacgtcttcaaagcaagtggattgatgtgatatctccactgac2940gtaagggatgacgcacaatcccactatccttcgcaagacccttcctctatataaggaagt3000tcatttcatttggagaggacacgctgaaatcaccagtctctctctacaaatctatctctc3060tcgagctttcgcagatctgtcgaaccaccatggcaccgaagaagaagcgcaag3113metalaprolyslyslysarglys15gtgcatatgaacaccaagtacaacaaggagttcctgctctacctggcg3161valhismetasnthrlystyrasnlysglupheleuleutyrleuala101520ggcttcgtggacggggacggctccatcatcgcccagatcaagccgaac3209glyphevalaspglyaspglyserileilealaglnilelysproasn25303540cagtcctacaagttcaagcatcagctgtccctcaccttcaccgtcacc3257glnsertyrlysphelyshisglnleuserleuthrphethrvalthr455055cagaagacacagcgccgttggttcctcgacaagctggtggacgagatc3305glnlysthrglnargargtrppheleuasplysleuvalaspgluile606570ggagtgggctacgtgtacgaccagggcagcgtctcccactaccgcctg3353glyvalglytyrvaltyraspglnglyservalserhistyrargleu758085tcccagatcaagcctctgcacaacttcctgacccagctccagcccttc3401serglnilelysproleuhisasnpheleuthrglnleuglnprophe9095100ctgaagctcaagcagaagcaggccaacctcgtgctgaagatcatcgag3449leulysleulysglnlysglnalaasnleuvalleulysileileglu105110115120cagctgccctccgccaaggaatccccggacaagttcctggaggtgtgc3497glnleuproseralalysgluserproasplyspheleugluvalcys125130135acgtgggtggaccagatcgcggccctcaacgacagcaagacccgcaag3545thrtrpvalaspglnilealaalaleuasnaspserlysthrarglys140145150acgacctcggagacggtgcgggcggtcctggactccctcccaggatcc3593thrthrsergluthrvalargalavalleuaspserleuproglyser155160165gtgggaggtctatcgccatctcaggcatccagcgccgcatcctcggct3641valglyglyleuserproserglnalaserseralaalaserserala170175180tcctcaagcccgggttcagggatctccgaagcactcagagctggagca3689serserserproglyserglyileserglualaleuargalaglyala185190195200actaagtccaaggaattcctgctctacctggcgggcttcgtcgacggg3737thrlysserlysglupheleuleutyrleualaglyphevalaspgly205210215gacggctccatcaaggccgcgatcaagccgaaccagtcctacaagttc3785aspglyserilelysalaalailelysproasnglnsertyrlysphe220225230aagcatcagctgtccctcaccttccaggtcacgcagaagacacagcgc3833lyshisglnleuserleuthrpheglnvalthrglnlysthrglnarg235240245cgttggttcctcgacaagctggtggacgagatcggggtgggctacgtg3881argtrppheleuasplysleuvalaspgluileglyvalglytyrval250255260tacgactccggcagcgtctccgactaccagctgtcccagatcaagcct3929tyraspserglyservalserasptyrglnleuserglnilelyspro265270275280ctgcacaacttcctgacccagctccagcccttcctgaagctcaagcag3977leuhisasnpheleuthrglnleuglnpropheleulysleulysgln285290295aagcaggccaacctcgtgctgaagatcatcgagcagctgccctccgcc4025lysglnalaasnleuvalleulysileilegluglnleuproserala300305310aaggaatccccggacaagttcctggaggtgtgcacctgggtggaccag4073lysgluserproasplyspheleugluvalcysthrtrpvalaspgln315320325atcgccgctctgaacgactccaagacccgcaagaccacttccgagacc4121ilealaalaleuasnaspserlysthrarglysthrthrsergluthr330335340gtccgcgccgttctagacagtctctccgagaagaagaagtcgtccccc4169valargalavalleuaspserleuserglulyslyslysserserpro345350355360tagcatgccgttcaaacatttggcaataaagtttcttaagattgaatcctgttgccggtc4229ttgcgatgattatcatataatttctgttgaattacgttaagcatgtaataattaacatgt4289aatgcatgacgttatttatgagatgggtttttatgattagagtcccgcaattatacattt4349aatacgcgatagaaaacaaaatatagcgcgcaaactaggataaattatcgcgcgcggtgt4409catctatgttactagatcgggcccgggaataaaatatctttattttcattacatctgtgt4469gttggttttttgtgtgaatcgatagtactaacatacgctctccatcaaaacaaaacgaaa4529caaaacaaactagcaaaataggctgtccccagtgcaagtgcaggtgccagaacatttcgg4589taccgagctcgaattcactggccgtcgttttacaacgtcgtgactgggaaaaccctggcg4649ttacccaacttaatcgccttgcagcacatccccctttcgccagctggcgtaatagcgaag4709aggcccgcaccgatcgcccttcccaacagttgcgcagcctgaatggcgaatggcgcctga4769tgcggtattttctccttacgcatctgtgcggtatttcacaccgcatatggtgcactctca4829gtacaatctgctctgatgccgcatagttaagccagccccgacacccgccaacacccgctg4889acgcgccctgacgggcttgtctgctcccggcatccgcttacagacaagctgtgaccgtct4949ccgggagctgcatgtgtcagaggttttcaccgtcatcaccgaaacgcgcga5000<210>6<211>360<212>prt<213>人工序列<220><223>合成的構建體<400>6metalaprolyslyslysarglysvalhismetasnthrlystyrasn151015lysglupheleuleutyrleualaglyphevalaspglyaspglyser202530ileilealaglnilelysproasnglnsertyrlysphelyshisgln354045leuserleuthrphethrvalthrglnlysthrglnargargtrpphe505560leuasplysleuvalaspgluileglyvalglytyrvaltyraspgln65707580glyservalserhistyrargleuserglnilelysproleuhisasn859095pheleuthrglnleuglnpropheleulysleulysglnlysglnala100105110asnleuvalleulysileilegluglnleuproseralalysgluser115120125proasplyspheleugluvalcysthrtrpvalaspglnilealaala130135140leuasnaspserlysthrarglysthrthrsergluthrvalargala145150155160valleuaspserleuproglyservalglyglyleuserprosergln165170175alaserseralaalaserseralaserserserproglyserglyile180185190serglualaleuargalaglyalathrlysserlysglupheleuleu195200205tyrleualaglyphevalaspglyaspglyserilelysalaalaile210215220lysproasnglnsertyrlysphelyshisglnleuserleuthrphe225230235240glnvalthrglnlysthrglnargargtrppheleuasplysleuval245250255aspgluileglyvalglytyrvaltyraspserglyservalserasp260265270tyrglnleuserglnilelysproleuhisasnpheleuthrglnleu275280285glnpropheleulysleulysglnlysglnalaasnleuvalleulys290295300ileilegluglnleuproseralalysgluserproasplyspheleu305310315320gluvalcysthrtrpvalaspglnilealaalaleuasnaspserlys325330335thrarglysthrthrsergluthrvalargalavalleuaspserleu340345350serglulyslyslysserserpro355360<210>7<211>13220<212>dna<213>人工序列<220><223>修復dna載體<220><221>cds<222>(5214)..(6290)<220><221>cds<222>(8867)..(10204)<400>7gtggcacttttcggggaaatgtgcgcggaacccctatttgtttatttttctaaatacatt60caaatatgtatccgctcatgagacaataaccctgataaatgcttcaataatattgaaaaa120ggaagagtatgagtattcaacatttccgtgtcgcccttattcccttttttgcggcatttt180gccttcctgtttttgctcacccagaaacgctggtgaaagtaaaagatgctgaagatcagt240tgggtgcacgagtgggttacatcgaactggatctcaacagcggtaagatccttgagagtt300ttcgccccgaagaacgttttccaatgatgagcacttttaaagttctgctatgtggcgcgg360tattatcccgtattgacgccgggcaagagcaactcggtcgccgcatacactattctcaga420atgacttggttgagtactcaccagtcacagaaaagcatcttacggatggcatgacagtaa480gagaattatgcagtgctgccataaccatgagtgataacactgcggccaacttacttctga540caacgatcggaggaccgaaggagctaaccgcttttttgcacaacatgggggatcatgtaa600ctcgccttgatcgttgggaaccggagctgaatgaagccataccaaacgacgagcgtgaca660ccacgatgcctgtagcaatggcaacaacgttgcgcaaactattaactggcgaactactta720ctctagcttcccggcaacaattaatagactggatggaggcggataaagttgcaggaccac780ttctgcgctcggcccttccggctggctggtttattgctgataaatctggagccggtgagc840gtggatctcgcggtatcattgcagcactggggccagatggtaagccctcccgtatcgtag900ttatctacacgacggggagtcaggcaactatggatgaacgaaatagacagatcgctgaga960taggtgcctcactgattaagcattggtaactgtcagaccaagtttactcatatatacttt1020agattgatttaaaacttcatttttaatttaaaaggatctaggtgaagatcctttttgata1080atctcatgaccaaaatcccttaacgtgagttttcgttccactgagcgtcagaccccgtag1140aaaagatcaaaggatcttcttgagatcctttttttctgcgcgtaatctgctgcttgcaaa1200caaaaaaaccaccgctaccagcggtggtttgtttgccggatcaagagctaccaactcttt1260ttccgaaggtaactggcttcagcagagcgcagataccaaatactgtccttctagtgtagc1320cgtagttaggccaccacttcaagaactctgtagcaccgcctacatacctcgctctgctaa1380tcctgttaccagtggctgctgccagtggcgataagtcgtgtcttaccgggttggactcaa1440gacgatagttaccggataaggcgcagcggtcgggctgaacggggggttcgtgcacacagc1500ccagcttggagcgaacgacctacaccgaactgagatacctacagcgtgagctatgagaaa1560gcgccacgcttcccgaagggagaaaggcggacaggtatccggtaagcggcagggtcggaa1620caggagagcgcacgagggagcttccagggggaaacgcctggtatctttatagtcctgtcg1680ggtttcgccacctctgacttgagcgtcgatttttgtgatgctcgtcaggggggcggagcc1740tatggaaaaacgccagcaacgcggcctttttacggttcctggccttttgctggccttttg1800ctcacatgttctttcctgcgttatcccctgattctgtggataaccgtattaccgcctttg1860agtgagctgataccgctcgccgcagccgaacgaccgagcgcagcgagtcagtgagcgagg1920aagcggaagagcgcccaatacgcaaaccgcctctccccgcgcgttggccgattcattaat1980gcagctggcacgacaggtttcccgactggaaagcgggcagtgagcgcaacgcaattaatg2040tgagttagctcactcattaggcaccccaggctttacactttatgcttccggctcgtatgt2100tgtgtggaattgtgagcggataacaatttcacacaggaaacagctatgaccatgattacg2160ccaagcgcgcaattaaccctcactaaagggaacaaaagctggagcttgcaagcttcacgt2220gatatcccatcggaggaagatctttaattaattttatttttaacaggaatttgagtcacg2280caatttctaatacttgttcaaattgatttgaaaaaaaaaattgaataactcaaataatgc2340aattcaattaacttgaaattcgaacaattttttatttttattttttaaattgaatcaagt2400tttacttacccgtaattatttgaatggatcaagttgagttaaattcaaccttttttttct2460ttttaagaattggaccataactcaacttagctcgtaaatatgttgtaaccgttgagccat2520cagtgtaatattaactaccttgtttgtaactcctacacaagtcgtatagtctattagcgg2580ttgctttgagtaattctatgtttagatgtcatatatcgtattatattattcgagttttag2640tgtgattaatgaatataaatacttgtcaacgtagatatactaaatttttttaggatttta2700tgtattcgggagtcatatatatatatcatattttcatatttacgaatgagttgaatacga2760gtttcgaatacatatttaaaagaataaataaaatgttggtaacatagggttggtgggctt2820tgataatgggctaatggcaatggatttgcacatacacccctaaccaccttcattttggtt2880gattttttaaatattttttcttgtgtgctgaataaataaataaataaataaaacgaatcg2940aatccatgtgtttggactatctaaaaaatgaaactttaacttaatggaattctatttttt3000tttattttatgattaatttaaatttttataatatagaaataagttttttatatttaaaat3060aattaaaataacttaaaatttttatataaaaagtatttttttcaaaaaataccattttaa3120aaagttattaatggttttttatttactttgaaaaattattttagaaaaaaatttcaaatt3180tatttttaataattttattattatacattctcattatatctgttatttttaacacaatac3240ttaaaatcatctatagtccctctttaactgcgtttcaacgcactcgaactcacattctcc3300tatattaacgataatgccatactaatcaaattaagacttagtcgacaaaaatacttttaa3360actttaccattatgaatgaatccaaaaatatatccattaaaaaaaattcaacatgctaca3420tgattgtttgagatttgacttagtttgtttttggaccttaacaatcaatcatatctataa3480ctatatctttatttagaatttaaagggtttgttttgatgacacctaagcatagatataaa3540atataatttaaagtactgaagttgagaatttacttttaaaggcttttcagaaaaaaataa3600aaaaataaaggattggattattagacaacaatgcaatgttattaaattaagtatcgggac3660gattgagtcttaattagattagtattagtgttgttatcagtgtgaaatgatgtgggttcg3720agtatgctttagagcattatcctcctatttaaggattaggaaggggttatgggtagtttt3780agatattgtatccaaaatagcaaatgatattgatgttcaaaaaaaaaattaaaagtttgg3840gattaaatttagaagattatttgtataaattaaaatacaaaactttttttttatcataat3900taaggggtatatatcaaaattatatataaattatgatttaatattcaatttgatatacga3960actttgattttatacaatttaatacataaatattaaatttagctcaattttcacaaataa4020ttaacatcgtactcgatgtagcatcattttaggaaaaaaattatatacctaacatgaatt4080ttatttgattgatttagtttttaaaatatttacaattaaatcaaaatcaaaattatatat4140atataattgaaacaaaattatagtttctacattaggcaaatcatttataaatatttttgt4200gttatattttacgtgtttaaaatgatataaaattatacaaaaacaatattaaatgttaat4260aatttgtttaatatgaattttgagagcactatttttatgccgtgcctaattaaaattatt4320tacgcgatcgctacgtacctgcagggcggccgcaacatggtggagcacgacactctcgtc4380tactccaagaatatcaaagatacagtctcagaagaccaaagggctattgagacttttcaa4440caaagggtaatatcgggaaacctcctcggattccattgcccagctatctgtcacttcatc4500aaaaggacagtagaaaaggaaggtggcacctacaaatgccatcattgcgataaaggaaag4560gctatcgttcaagatgcccctgccgacagtggtcccaaagatggacccccacccacgagg4620agcatcgtggaaaaagaagacgttccaaccacgtcttcaaagcaagtggattgatgtgat4680atctccactgacgtaagggatgacgcacaatcccactatccttcgcaagacccttcctct4740atataaggaagttcatttcatttggagaggactcgagctcatttctctattacttcagcc4800ataacaaaagaactcttttctcttcttattaaaccaaaaccatggctagtatttcatctt4860cagttgccactgtttcacgtaccgcaccagcccaagctaatatggtggctccgttcacag4920gtcttaagtcaaatgctgcatttccaaccactaagaaggctaatgacttctcaacacttc4980cgagcaacggtgggagagtacagtatatgcaagtctggccagcctatggcaacaagaagt5040ttgagaccttatcctacttacctccgctatctatggctcccactgtgatgatggccagct5100ctgccacagctgtggcaccatttcaagggttgaagagtactgctagtttgccagttgctc5160ggagatcttccagatcgctggggaatgttagcaacggaggtaggattagatgcatg5216met1gctgatctatacgagaatcctatgggactcatgggtttcgagttcatt5264alaaspleutyrgluasnprometglyleumetglypheglupheile51015gagtttgcttctcctacacctgggacccttgaacccatctttgagatt5312gluphealaserprothrproglythrleugluproilephegluile202530atggggtttactaaggtagctacacatcgcagtaagaacgtgcaccta5360metglyphethrlysvalalathrhisargserlysasnvalhisleu354045tatcggcaaggagagattaacctgatcttgaacaacgaacctaactct5408tyrargglnglygluileasnleuileleuasnasngluproasnser50556065atagcaagctactttgctgccgaacatggaccatccgtttgtggcatg5456ilealasertyrphealaalagluhisglyproservalcysglymet707580gcatttcgagttaaggattcccagaaagcctacaatagggcattagaa5504alapheargvallysaspserglnlysalatyrasnargalaleuglu859095cttggagctcagcctattcacattgacactggacctatggaattgaat5552leuglyalaglnproilehisileaspthrglyprometgluleuasn100105110cttccagccatcaaaggcatcggtggtgcaccgttgtatcttatcgac5600leuproalailelysglyileglyglyalaproleutyrleuileasp115120125agattcggagagggcagttctatctacgacatagatttcgtctatctt5648argpheglygluglyserseriletyraspileaspphevaltyrleu130135140145gaaggcgtggaacgaaatcctgtcggtgctggcttgaaggttattgat5696gluglyvalgluargasnprovalglyalaglyleulysvalileasp150155160catttgacacacaacgtctatcgtggacgaatggtatactgggctaac5744hisleuthrhisasnvaltyrargglyargmetvaltyrtrpalaasn165170175ttctatgagaaactcttcaactttagggaagctagatactttgacatc5792phetyrglulysleupheasnpheargglualaargtyrpheaspile180185190aaaggagagtatactggtctcacatcaaaggctatgagcgctccagat5840lysglyglutyrthrglyleuthrserlysalametseralaproasp195200205ggtatgataaggattccactcaacgaagagtcaagcaaaggtgcagga5888glymetileargileproleuasnglugluserserlysglyalagly210215220225caaatcgaagagtttcttatgcagttcaatggtgaaggcattcagcat5936glnilegluglupheleumetglnpheasnglygluglyileglnhis230235240gtcgcttttctgactgatgatcttgtgaaaacctgggatgcattgaag5984valalapheleuthraspaspleuvallysthrtrpaspalaleulys245250255aagataggtatgagattcatgactgcacctcccgatacttactatgag6032lysileglymetargphemetthralaproproaspthrtyrtyrglu260265270atgctagaaggacgcctgcctgatcatggggaacccgtggaccaactg6080metleugluglyargleuproasphisglygluprovalaspglnleu275280285caagccaggggtatacttctcgatggttcttcggttgagggtgataag6128glnalaargglyileleuleuaspglyserservalgluglyasplys290295300305aggctcctgttacaaatcttttccgagactctcatgggacccgttttc6176argleuleuleuglnilephesergluthrleumetglyprovalphe310315320tttgagttcattcagcgtaaaggggacgatggcttcggagaatggaac6224pheglupheileglnarglysglyaspaspglypheglyglutrpasn325330335ttcaaagcacttttcgaatcgatagagagggatcaagttcgtagaggg6272phelysalaleuphegluserilegluargaspglnvalargarggly340345350gttttgaccgctgattaaaagctagcacgcgtggcgcgcccccgatcc6320valleuthralaasp355gcgtttgtgttttctgggtttctcacttaagcgtctgcgttttacttttgtattgggttt6380ggcgtttagtagtttgcggtagcgttcttgttatgtgtaattacgctttttcttcttgct6440tcagcagtttcggttgaaatataaatcgaatcaagtttcactttatcagcgttgttttaa6500attttggcattaaattggtgaaaattgcttcaattttgtatctaaatagaagagacaaca6560tgaaattcgacttttgacctcaaatcttcgaacatttatttcctgatttcacgatggatg6620aggataacgaaagggcggttcctatgtccgggaaagttcccgtagaagacaatgagcaaa6680gctactgaaacgcggacacgacgtcgcattggtacggatatgagttaaaccgactcaatt6740cctttattaagacataaaccgattttggttaaagtgtaacagtgagctgatataaaaccg6800aaacaaaccggtacaagtttgattgagcaacttgatgacaaacttcagaattttggttat6860tgaatgaaaatcatagtctaatcgtaaaaaatgtacagaagaaaagctagagcagaacaa6920agattctatattctggttccaatttatcatcgctttaacgtccctcagatttgatcgggg6980aattcgatatcattaccctgttatccctaaagcttattaatataacttcgtatagcatac7040attatacgaagttatgtttgtcgaggagaaatatgagtcgaggcatggatacactaagtt7100cccctgaagtgagcatgatctttgatgctgagatgattcccagagcaagatagtttgtgc7160tgcaagtgacacaattgtaatgaaaccaccactcaacgaatttacttgtggctttgacat7220gtcgtgtgctctgtttgtatttgtgagtgccggttggtaattatttttgttaatgtgatt7280ttaaaacctcttatgtaaatagttactttatctattgaagtgtgttcttgtggtctatag7340tttctcaaagggaaattaaaatgttgacatcccatttacaattgataacttggtatacac7400aaactttgtaaatttggtgatatttatggtcgaaagaaggcaatacccattgtatgttcc7460aatatcaatatcaatacgataacttgataatactaacatatgattgtcattgtttttcca7520gtatcaatatacattaagctactacaaaattagtataaatcactatattataaatctttt7580tcggttgtaacttgtaattcgtgggtttttaaaataaaagcatgtgaaaattttcaaata7640atgtgatggcgcaattttattttccgagttccaaaatattgccgcttcattaccctaatt7700tgtggcgccacatgtaaaacaaaagacgattcttagtggctatcactgccatcacgcgga7760tcactaatatgaaccgtcgattaaaacagatcgacggtttatacatcattttattgtaca7820cacggatcgatatctcagccgttagatttaatatgcgatctgattgctcaaaaaatagac7880tctccgtctttgcctataaaaacaatttcacatctttctcacccaaatctactcttaacc7940gttcttcttcttctacagacatcaatttctctcgactctagaggatccaagcttatcgat8000ttcgaacccctcaggcgaagaacaggtatgatttgtttgtaattagatcaggggtttagg8060tctttccattactttttaatgttttttctgttactgtctccgcgatctgattttacgaca8120atagagtttcgggttttgtcccattccagtttgaaaataaaggtccgtcttttaagtttg8180ctggatcgataaacctgtgaagattgagtctagtcgatttattggatgatccattcttca8240tcgtttttttcttgcttcgaagttctgtataaccagatttgtctgtgtgcgattgtcatt8300acctagccgtgtatcgagaactagggttttcgagtcaattttgccccttttggttatatc8360tggttcgataacgattcatctggattagggttttaagtggtgacgtttagtattccaatt8420tcttcaaaatttagttatggataatgaaaatccccaattgactgttcaatttcttgttaa8480atgcgcagatcacaatggcttcgatctcctcctcagtcgcgaccgttagccggaccgccc8540ctgctcaggccaacatggtggctccgttcaccggccttaagtccaacgccgccttcccca8600ccaccaagaaggctaacgacttctccacccttcccagcaacggtggaagagttcaatgta8660tgcaggtgtggccggcctacggcaacaagaagttcgagacgctgtcgtacctgccgccgc8720tgtctatggcgcccaccgtgatgatggcctcgtcggccaccgccgtcgctccgttccagg8780ggctcaagtccaccgccagcctccccgtcgcccgccgctcctccagaagcctcggcaacg8840tcagcaacggcggaaggatccggtgcatggccggcgccgaggagatcgtgctg8893metalaglyalaglugluilevalleu360365cagcccatcaaggagatctccggcaccgtcaagctgccggggtccaag8941glnproilelysgluileserglythrvallysleuproglyserlys370375380tcgctttccaaccggatcctcctactcgccgccctgtccgaggggaca8989serleuserasnargileleuleuleualaalaleusergluglythr385390395acagtggttgataacctgctgaacagtgaggatgtccactacatgctc9037thrvalvalaspasnleuleuasnsergluaspvalhistyrmetleu400405410415ggggccttgaggactcttggtctctctgtcgaagcggacaaagctgcc9085glyalaleuargthrleuglyleuservalglualaasplysalaala420425430aaaagagctgtagttgttggctgtggtggaaagttcccagttgaggat9133lysargalavalvalvalglycysglyglylyspheprovalgluasp435440445gctaaagaggaagtgcagctcttcttggggaatgctggaatcgcaatg9181alalysglugluvalglnleupheleuglyasnalaglyilealamet450455460cggtccttgacagcagctgttactgctgctggtggaaatgcaacttac9229argserleuthralaalavalthralaalaglyglyasnalathrtyr465470475gtgcttgatggagtaccaagaatgagggagagacccattggcgacttg9277valleuaspglyvalproargmetarggluargproileglyaspleu480485490495gttgtcggattgaagcagcttggtgcagatgttgattgtttccttggc9325valvalglyleulysglnleuglyalaaspvalaspcyspheleugly500505510actgactgcccacctgttcgtgtcaatggaatcggagggctacctggt9373thraspcysproprovalargvalasnglyileglyglyleuprogly515520525ggcaaggtcaagctgtctggctccatcagcagtcagtacttgagtgcc9421glylysvallysleuserglyserileserserglntyrleuserala530535540ttgctgatggctgctcctttggctcttggggatgtggagattgaaatc9469leuleumetalaalaproleualaleuglyaspvalgluilegluile545550555attgataaattaatctccattccgtacgtcgaaatgacattgagattg9517ileasplysleuileserileprotyrvalglumetthrleuargleu560565570575atggagcgttttggtgtgaaagcagagcattctgatagctgggacaga9565metgluargpheglyvallysalagluhisseraspsertrpasparg580585590ttctacattaagggaggtcaaaaatacaagtcccctaaaaatgcctat9613phetyrilelysglyglyglnlystyrlysserprolysasnalatyr595600605gttgaaggtgatgcctcaagcgcaagctatttcttggctggtgctgca9661valgluglyaspalaserseralasertyrpheleualaglyalaala610615620attactggagggactgtgactgtggaaggttgtggcaccaccagtttg9709ilethrglyglythrvalthrvalgluglycysglythrthrserleu625630635cagggtgatgtgaagtttgctgaggtactggagatgatgggagcgaag9757glnglyaspvallysphealagluvalleuglumetmetglyalalys640645650655gttacatggaccgagactagcgtaactgttactggcccaccgcgggag9805valthrtrpthrgluthrservalthrvalthrglyproproargglu660665670ccatttgggaggaaacacctcaaggcgattgatgtcaacatgaacaag9853propheglyarglyshisleulysalaileaspvalasnmetasnlys675680685atgcctgatgtcgccatgactcttgctgtggttgccctctttgccgat9901metproaspvalalametthrleualavalvalalaleuphealaasp690695700ggcccgacagccatcagagacgtggcttcctggagagtaaaggagacc9949glyprothralaileargaspvalalasertrpargvallysgluthr705710715gagaggatggttgcgatccggacggagctaaccaagctgggagcatct9997gluargmetvalalaileargthrgluleuthrlysleuglyalaser720725730735gttgaggaagggccggactactgcatcatcacgccgccggagaagctg10045valglugluglyproasptyrcysileilethrproproglulysleu740745750aacgtgacggcgatcgacacgtacgacgaccacaggatggcgatggct10093asnvalthralaileaspthrtyraspasphisargmetalametala755760765ttctcccttgccgcctgtgccgaggtccccgtcaccatccgggaccct10141pheserleualaalacysalagluvalprovalthrileargasppro770775780gggtgcacccggaagaccttccccgactacttcgatgtgctgagcact10189glycysthrarglysthrpheproasptyrpheaspvalleuserthr785790795ttcgtcaagaattaagctctagaactagtggatcccccgatccgcgtttgtgttt10244phevallysasn800tctgggtttctcacttaagcgtctgcgttttacttttgtattgggtttggcgtttagtag10304tttgcggtagcgttcttgttatgtgtaattacgctttttcttcttgcttcagcagtttcg10364gttgaaatataaatcgaatcaagtttcactttatcagcgttgttttaaattttggcatta10424aattggtgaaaattgcttcaattttgtatctaaatagaagagacaacatgaaattcgact10484tttgacctcaaatcttcgaacatttatttcctgatttcacgatggatgaggataacgaaa10544gggcggttcctatgtccgggaaagttcccgtagaagacaatgagcaaagctactgaaacg10604cggacacgacgtcgcattggtacggatatgagttaaaccgactcaattcctttattaaga10664cataaaccgattttggttaaagtgtaacagtgagctgatataaaaccgaaacaaaccggt10724acaagtttgattgagcaacttgatgacaaacttcagaattttggttattgaatgaaaatc10784atagtctaatcgtaaaaaatgtacagaagaaaagctagagcagaacaaagattctatatt10844ctggttccaatttatcatcgctttaacgtccctcagatttgatcgggaaacataacttcg10904tatagcatacattatacgaagttatcaaaacgtcgtgagacagtttggttaacttacaag10964tgtttatgttgatattttaattatgtttcatataaatttgtcgtaatagtttaattgtta11024aatctaaataaaagtatttaaaaatagatattatattttttatgatatatataaatatca11084tgaaactttagaatttatggacaaaaacatcactatccactagttaatcatttctacaaa11144cgacatttatttatttaaaatggtcagaaaaattgtgacctaccaggaatgaggatttga11204ttggagggatgtaaaccaagttgatatttttttctcacttgtgaaaaagatagtgtttgg11264ctggaattttatgtatgataaatttgatagggacttgcacttgagattattttctgggtt11324cttcatgcttttataataacttgactatcaaattctctctttggattctacgacgttcat11384atctatttgtatggtggcttcgagtaggaatttttttaaaagtatttgtgtttgatattt11444acatatgtttttatacaatgtttagaactacttatagtccatttcatcccttaagtaggt11504acgccttcctacattgacaacaatatcgatactaatcgaactaaaactcaatcaacgccc11564atattcgacacttcaatttggataataagtagagtattttatagatatttgatttatatg11624taaatgatttttttaatattttttgagagtattttaaaattcgtgttttattaattttca11684tattatttagattcttaggattgaaaaaaaatttataaatttgaaaaactctctcgtgga11744aaagaattacaaaataatttaacccctaataaaaatataacaaccatttttttatgtcaa11804tcactacgttattcatcgcatcattacttaacgtatcatcaaagatttgatgtaaaaatt11864aaattcaagggctcaagccatgagagcttcaatgaaaatattcattgctaacttttgatt11924gaaattttaattgaatataattttttcttgtgagcttaacggatattttaaattacctta11984caaaaactgtttaaaacttaaaactgtataataataaaacttatattctctaacaacata12044taaatctataatttgtagtttgtcccctaaacttgtccaacaatatcaaattaatgtctg12104aattttttatcgagttggtacgtctattctaacaccttaatttatgctgatgcgacacta12164ccagccaatcaaatatgacacataataaccttcaatatgagatgtgatcaattaagaagt12224ttagaaatctttagaaattatgatttatattcccatcatccccacaaccatatcttacct12284cttaaaaatatgctttctaaaaatatgatttgatttgctgtgtgaaaaaaaaaaggcaaa12344agaaaatatctaagaaataaatcaataattgagagaggtcaaaagacgatattatccttt12404tgatttgctggtctctagaaagcaaaagatgggggattagggtaggcagaataaaaaaaa12464aaatctagggaaagaagatcccactttaaccaatgaacgacagcccacctcattagagcc12524taggatccagattccaagggaagcttggccttgacggccgtacccaattcgccctatagt12584gagtcgtattacgcgcgctcactggccgtcgttttacaacgtcgtgactgggaaaaccct12644ggcgttacccaacttaatcgccttgcagcacatccccctttcgccagctggcgtaatagc12704gaagaggcccgcaccgatcgcccttcccaacagttgcgcagcctgaatggcgaatgggac12764gcgccctgtagcggcgcattaagcgcggcgggtgtggtggttacgcgcagcgtgaccgct12824acacttgccagcgccctagcgcccgctcctttcgctttcttcccttcctttctcgccacg12884ttcgccggctttccccgtcaagctctaaatcgggggctccctttagggttccgatttagt12944gctttacggcacctcgaccccaaaaaacttgattagggtgatggttcacgtagtgggcca13004tcgccctgatagacggtttttcgccctttgacgttggagtccacgttctttaatagtgga13064ctcttgttccaaactggaacaacactcaaccctatctcggtctattcttttgatttataa13124gggattttgccgatttcggcctattggttaaaaaatgagctgatttaacaaaaatttaac13184gcgaattttaacaaaatattaacgcttacaatttag13220<210>8<211>358<212>prt<213>人工序列<220><223>合成的構建體<400>8metalaaspleutyrgluasnprometglyleumetglyphegluphe151015ilegluphealaserprothrproglythrleugluproilepheglu202530ilemetglyphethrlysvalalathrhisargserlysasnvalhis354045leutyrargglnglygluileasnleuileleuasnasngluproasn505560serilealasertyrphealaalagluhisglyproservalcysgly65707580metalapheargvallysaspserglnlysalatyrasnargalaleu859095gluleuglyalaglnproilehisileaspthrglyprometgluleu100105110asnleuproalailelysglyileglyglyalaproleutyrleuile115120125aspargpheglygluglyserseriletyraspileaspphevaltyr130135140leugluglyvalgluargasnprovalglyalaglyleulysvalile145150155160asphisleuthrhisasnvaltyrargglyargmetvaltyrtrpala165170175asnphetyrglulysleupheasnpheargglualaargtyrpheasp180185190ilelysglyglutyrthrglyleuthrserlysalametseralapro195200205aspglymetileargileproleuasnglugluserserlysglyala210215220glyglnilegluglupheleumetglnpheasnglygluglyilegln225230235240hisvalalapheleuthraspaspleuvallysthrtrpaspalaleu245250255lyslysileglymetargphemetthralaproproaspthrtyrtyr260265270glumetleugluglyargleuproasphisglygluprovalaspgln275280285leuglnalaargglyileleuleuaspglyserservalgluglyasp290295300lysargleuleuleuglnilephesergluthrleumetglyproval305310315320phepheglupheileglnarglysglyaspaspglypheglyglutrp325330335asnphelysalaleuphegluserilegluargaspglnvalargarg340345350glyvalleuthralaasp355<210>9<211>445<212>prt<213>人工序列<220><223>合成的構建體<400>9metalaglyalaglugluilevalleuglnproilelysgluileser151015glythrvallysleuproglyserlysserleuserasnargileleu202530leuleualaalaleusergluglythrthrvalvalaspasnleuleu354045asnsergluaspvalhistyrmetleuglyalaleuargthrleugly505560leuservalglualaasplysalaalalysargalavalvalvalgly65707580cysglyglylyspheprovalgluaspalalysglugluvalglnleu859095pheleuglyasnalaglyilealametargserleuthralaalaval100105110thralaalaglyglyasnalathrtyrvalleuaspglyvalproarg115120125metarggluargproileglyaspleuvalvalglyleulysglnleu130135140glyalaaspvalaspcyspheleuglythraspcysproprovalarg145150155160valasnglyileglyglyleuproglyglylysvallysleusergly165170175serileserserglntyrleuseralaleuleumetalaalaproleu180185190alaleuglyaspvalgluilegluileileasplysleuileserile195200205protyrvalglumetthrleuargleumetgluargpheglyvallys210215220alagluhisseraspsertrpaspargphetyrilelysglyglygln225230235240lystyrlysserprolysasnalatyrvalgluglyaspalaserser245250255alasertyrpheleualaglyalaalailethrglyglythrvalthr260265270valgluglycysglythrthrserleuglnglyaspvallyspheala275280285gluvalleuglumetmetglyalalysvalthrtrpthrgluthrser290295300valthrvalthrglyproproargglupropheglyarglyshisleu305310315320lysalaileaspvalasnmetasnlysmetproaspvalalametthr325330335leualavalvalalaleuphealaaspglyprothralaileargasp340345350valalasertrpargvallysgluthrgluargmetvalalailearg355360365thrgluleuthrlysleuglyalaservalglugluglyproasptyr370375380cysileilethrproproglulysleuasnvalthralaileaspthr385390395400tyraspasphisargmetalametalapheserleualaalacysala405410415gluvalprovalthrileargaspproglycysthrarglysthrphe420425430proasptyrpheaspvalleuserthrphevallysasn435440445<210>10<211>554<212>dna<213>人工序列<220><223>探針<400>10ttgagtctccttctggtactcctggtggtcttagggcttacatggtttctgttcataaca60ggaagaacaacatctacgctgttcatgagaacggtactatgattcatcttgctcctgagg120attacaccggtttcaccatctcccccatccacgccacccaggtcaataatcagaccagga180ccttcatctccgagaagttcggcaaccagggcgactccctgaggttcgagcagtccaaca240ccaccgccaggtacaccctgaggggcaacggcaactcctacaacctgtacctcagggtgt300cctccctcggcaactccaccatcagggtcaccatcaacggcagggtgtacaccgcctcca360acgtgaacaccaccaccaacaacgacggcgtcaacgacaacggcgctaggttcctggaca420tcaacatgggcaacgtcgtggcctccgacaacaccaacgtgcccctggacatcaacgtga480catttaactccggcacccagttcgagctgatgaacatcatgttcgtgccaactaacctcc540cacccatctactga554<210>11<211>828<212>dna<213>人工序列<220><223>探針<400>11accgaagaaccatcgagaagtatacccctggcttgcagttggtccacgggttccccatga60tcaggcaggcgtccttctagcatctcatagtaagtatcgggaggtgcagtcatgaatctc120atacctatcttcttcaatgcatcccaggttttcacaagatcatcagtcagaaaagcgaca180tgctgaatgccttcaccattgaactgcataagaaactcttcgatttgtcctgcacctttg240cttgactcttcgttgagtggaatccttatcataccatctggagcgctcatagcctttgat300gtgagaccagtatactctcctttgatgtcaaagtatctagcttccctaaagttgaagagt360ttctcatagaagttagcccagtataccattcgtccacgatagacgttgtgtgtcaaatga420tcaataaccttcaagccagcaccgacaggatttcgttccacgccttcaagatagacgaaa480tctatgtcgtagatagaactgccctctccgaatctgtcgataagatacaacggtgcacca540ccgatgcctttgatggctggaagattcaattccataggtccagtgtcaatgtgaataggc600tgagctccaagttctaatgccctattgtaggctttctgggaatccttaactcgaaatgcc660atgccacaaacggatggtccatgttcggcagcaaagtagcttgctatagagttaggttcg720ttgttcaagatcaggttaatctctccttgccgatataggtgcacgttcttactgcgatgt780gtagctaccttagtaaaccccataatctcaaagatgggttcaagggtc828<210>12<211>745<212>dna<213>人工序列<220><223>探針<400>12cggcgtgatgatgcagtagtccggcccttcctcaacagatgctcccagcttggttagctc60cgtccggatcgcaaccatcctctcggtctcctttactctccaggaagccacgtctctgat120ggctgtcgggccatcggcaaagagggcaaccacagcaagagtcatggcgacatcaggcat180cttgttcatgttgacatcaatcgccttgaggtgtttcctcccaaatggctcccgcggtgg240gccagtaacagttacgctagtctcggtccatgtaaccttcgctcccatcatctccagtac300ctcagcaaacttcacatcaccctgcaaactggtggtgccacaaccttccacagtcacagt360ccctccagtaattgcagcaccagccaagaaatagcttgcgcttgaggcatcaccttcaac420ataggcatttttaggggacttgtatttttgacctcccttaatgtagaatctgtcccagct480atcagaatgctctgctttcacaccaaaacgctccatcaatctcaatgtcatttcgacgta540cggaatggagattaatttatcaatgatttcaatctccacatccccaagagccaaaggagc600agccatcagcaaggcactcaagtactgactgctgatggagccagacagcttgaccttgcc660accaggtagccctccgattccattgacacgaacaggtgggcagtcagtgccaaggaaaca720atcaacatctgcaccaagctgcttc745<210>13<211>25<212>dna<213>人工序列<220><223>引物<400>13tccagtactaaaatccagatcatgc25<210>14<211>22<212>dna<213>人工序列<220><223>引物<400>14tgatgcgatgaataacgtagtg22<210>15<211>20<212>dna<213>人工序列<220><223>pcr引物<400>15ccctggcttgcagttggtcc20<210>16<211>163<212>prt<213>chlamydomonasreinhardtii<400>16metasnthrlystyrasnlysglupheleuleutyrleualaglyphe151015valaspglyaspglyserileilealaglnilelysproasnglnser202530tyrlysphelyshisglnleuserleuthrpheglnvalthrglulys354045thrglnargargtrppheleuasplysleuvalaspgluileglyval505560glytyrvalargaspargglyservalserasptyrileleuserglu65707580ilelysproleuhisasnpheleuthrglnleuglnpropheleulys859095leulysglnlysglnalaasnleuvalleulysileilegluglnleu100105110proseralalysgluserproasplyspheleugluvalcysthrtrp115120125valaspglnilealaalaleuasnaspserlysthrarglysthrthr130135140sergluthrvalargalavalleuaspserleuserglulyslyslys145150155160serserpro<210>17<211>20<212>dna<213>人工序列<220><223>pcr引物<400>17tataaatgccggcgcgtagc20<210>18<211>20<212>dna<213>人工序列<220><223>pcr引物<400>18cgacagccatcagagacgtg20<210>19<211>358<212>prt<213>熒光假單胞菌<400>19metalaaspleutyrgluasnprometglyleumetglyphegluphe151015ilegluphealaserprothrproglythrleugluproilepheglu202530ilemetglyphethrlysvalalathrhisargserlysasnvalhis354045leutyrargglnglygluileasnleuileleuasnasngluproasn505560serilealasertyrphealaalagluhisglyproservalcysgly65707580metalapheargvallysaspserglnlysalatyrasnargalaleu859095gluleuglyalaglnproilehisileaspthrglyprometgluleu100105110asnleuproalailelysglyileglyglyalaproleutyrleuile115120125aspargpheglygluglyserseriletyraspileaspphevaltyr130135140leugluglyvalgluargasnprovalglyalaglyleulysvalile145150155160asphisleuthrhisasnvaltyrargglyargmetvaltyrtrpala165170175asnphetyrglulysleupheasnpheargglualaargtyrpheasp180185190ilelysglyglutyrthrglyleuthrserlysalametseralapro195200205aspglymetileargileproleuasnglugluserserlysglyala210215220glyglnilegluglupheleumetglnpheasnglygluglyilegln225230235240hisvalalapheleuthraspaspleuvallysthrtrpaspalaleu245250255lyslysileglymetargphemetthralaproproaspthrtyrtyr260265270glumetleugluglyargleuproasphisglygluprovalaspgln275280285leuglnalaargglyileleuleuaspglyserservalgluglyasp290295300lysargleuleuleuglnilephesergluthrleumetglyproval305310315320phepheglupheileglnarglysglyaspaspglypheglyglugly325330335asnphelysalaleuphegluserilegluargaspglnvalargarg340345350glyvalleuthralaasp355<210>20<211>8617<212>dna<213>人工的<220><223>載體<220><221>cds<222>(949)..(2025)<223>hppdpfw336<220><221>cds<222>(4602)..(5939)<223>2mepsps<400>20aattacaacggtatatatcctgccagtactgggccccctcgagggcgatcgctacgtacc60tgcaggcccgggttaattaagcggccgcaacatggtggagcacgacactctcgtctactc120caagaatatcaaagatacagtctcagaagaccaaagggctattgagacttttcaacaaag180ggtaatatcgggaaacctcctcggattccattgcccagctatctgtcacttcatcaaaag240gacagtagaaaaggaaggtggcacctacaaatgccatcattgcgataaaggaaaggctat300cgttcaagatgcccctgccgacagtggtcccaaagatggacccccacccacgaggagcat360cgtggaaaaagaagacgttccaaccacgtcttcaaagcaagtggattgatgtgatatctc420cactgacgtaagggatgacgcacaatcccactatccttcgcaagacccttcctctatata480aggaagttcatttcatttggagaggactcgagctcatttctctattacttcagccataac540aaaagaactcttttctcttcttattaaaccaaaaccatggctagtatttcatcttcagtt600gccactgtttcacgtaccgcaccagcccaagctaatatggtggctccgttcacaggtctt660aagtcaaatgctgcatttccaaccactaagaaggctaatgacttctcaacacttccgagc720aacggtgggagagtacagtatatgcaagtctggccagcctatggcaacaagaagtttgag780accttatcctacttacctccgctatctatggctcccactgtgatgatggccagctctgcc840acagctgtggcaccatttcaagggttgaagagtactgctagtttgccagttgctcggaga900tcttccagatcgctggggaatgttagcaacggaggtaggattagatgcatggctgat957metalaasp1ctatacgagaatcctatgggactcatgggtttcgagttcattgagttt1005leutyrgluasnprometglyleumetglypheglupheilegluphe51015gcttctcctacacctgggacccttgaacccatctttgagattatgggg1053alaserprothrproglythrleugluproilephegluilemetgly20253035tttactaaggtagctacacatcgcagtaagaacgtgcacctatatcgg1101phethrlysvalalathrhisargserlysasnvalhisleutyrarg404550caaggagagattaacctgatcttgaacaacgaacctaactctatagca1149glnglygluileasnleuileleuasnasngluproasnserileala556065agctactttgctgccgaacatggaccatccgtttgtggcatggcattt1197sertyrphealaalagluhisglyproservalcysglymetalaphe707580cgagttaaggattcccagaaagcctacaatagggcattagaacttgga1245argvallysaspserglnlysalatyrasnargalaleugluleugly859095gctcagcctattcacattgacactggacctatggaattgaatcttcca1293alaglnproilehisileaspthrglyprometgluleuasnleupro100105110115gccatcaaaggcatcggtggtgcaccgttgtatcttatcgacagattc1341alailelysglyileglyglyalaproleutyrleuileaspargphe120125130ggagagggcagttctatctacgacatagatttcgtctatcttgaaggc1389glygluglyserseriletyraspileaspphevaltyrleuglugly135140145gtggaacgaaatcctgtcggtgctggcttgaaggttattgatcatttg1437valgluargasnprovalglyalaglyleulysvalileasphisleu150155160acacacaacgtctatcgtggacgaatggtatactgggctaacttctat1485thrhisasnvaltyrargglyargmetvaltyrtrpalaasnphetyr165170175gagaaactcttcaactttagggaagctagatactttgacatcaaagga1533glulysleupheasnpheargglualaargtyrpheaspilelysgly180185190195gagtatactggtctcacatcaaaggctatgagcgctccagatggtatg1581glutyrthrglyleuthrserlysalametseralaproaspglymet200205210ataaggattccactcaacgaagagtcaagcaaaggtgcaggacaaatc1629ileargileproleuasnglugluserserlysglyalaglyglnile215220225gaagagtttcttatgcagttcaatggtgaaggcattcagcatgtcgct1677gluglupheleumetglnpheasnglygluglyileglnhisvalala230235240tttctgactgatgatcttgtgaaaacctgggatgcattgaagaagata1725pheleuthraspaspleuvallysthrtrpaspalaleulyslysile245250255ggtatgagattcatgactgcacctcccgatacttactatgagatgcta1773glymetargphemetthralaproproaspthrtyrtyrglumetleu260265270275gaaggacgcctgcctgatcatggggaacccgtggaccaactgcaagcc1821gluglyargleuproasphisglygluprovalaspglnleuglnala280285290aggggtatacttctcgatggttcttcggttgagggtgataagaggctc1869argglyileleuleuaspglyserservalgluglyasplysargleu295300305ctgttacaaatcttttccgagactctcatgggacccgttttctttgag1917leuleuglnilephesergluthrleumetglyprovalphepheglu310315320ttcattcagcgtaaaggggacgatggcttcggagaatggaacttcaaa1965pheileglnarglysglyaspaspglypheglyglutrpasnphelys325330335gcacttttcgaatcgatagagagggatcaagttcgtagaggggttttg2013alaleuphegluserilegluargaspglnvalargargglyvalleu340345350355accgctgattaaaagctagcacgcgtggcgcgcccccgatccgcgtttgtgt2065thralaasptttctgggtttctcacttaagcgtctgcgttttacttttgtattgggtttggcgtttagt2125agtttgcggtagcgttcttgttatgtgtaattacgctttttcttcttgcttcagcagttt2185cggttgaaatataaatcgaatcaagtttcactttatcagcgttgttttaaattttggcat2245taaattggtgaaaattgcttcaattttgtatctaaatagaagagacaacatgaaattcga2305cttttgacctcaaatcttcgaacatttatttcctgatttcacgatggatgaggataacga2365aagggcggttcctatgtccgggaaagttcccgtagaagacaatgagcaaagctactgaaa2425cgcggacacgacgtcgcattggtacggatatgagttaaaccgactcaattcctttattaa2485gacataaaccgattttggttaaagtgtaacagtgagctgatataaaaccgaaacaaaccg2545gtacaagtttgattgagcaacttgatgacaaacttcagaattttggttattgaatgaaaa2605tcatagtctaatcgtaaaaaatgtacagaagaaaagctagagcagaacaaagattctata2665ttctggttccaatttatcatcgctttaacgtccctcagatttgatcggggaattcgatat2725cattaccctgttatccctaaagcttattaatataacttcgtatagcatacattatacgaa2785gttatgtttgtcgaggagaaatatgagtcgaggcatggatacactaagttcccctgaagt2845gagcatgatctttgatgctgagatgattcccagagcaagatagtttgtgctgcaagtgac2905acaattgtaatgaaaccaccactcaacgaatttacttgtggctttgacatgtcgtgtgct2965ctgtttgtatttgtgagtgccggttggtaattatttttgttaatgtgattttaaaacctc3025ttatgtaaatagttactttatctattgaagtgtgttcttgtggtctatagtttctcaaag3085ggaaattaaaatgttgacatcccatttacaattgataacttggtatacacaaactttgta3145aatttggtgatatttatggtcgaaagaaggcaatacccattgtatgttccaatatcaata3205tcaatacgataacttgataatactaacatatgattgtcattgtttttccagtatcaatat3265acattaagctactacaaaattagtataaatcactatattataaatctttttcggttgtaa3325cttgtaattcgtgggtttttaaaataaaagcatgtgaaaattttcaaataatgtgatggc3385gcaattttattttccgagttccaaaatattgccgcttcattaccctaatttgtggcgcca3445catgtaaaacaaaagacgattcttagtggctatcactgccatcacgcggatcactaatat3505gaaccgtcgattaaaacagatcgacggtttatacatcattttattgtacacacggatcga3565tatctcagccgttagatttaatatgcgatctgattgctcaaaaaatagactctccgtctt3625tgcctataaaaacaatttcacatctttctcacccaaatctactcttaaccgttcttcttc3685ttctacagacatcaatttctctcgactctagaggatccaagcttatcgatttcgaacccc3745tcaggcgaagaacaggtatgatttgtttgtaattagatcaggggtttaggtctttccatt3805actttttaatgttttttctgttactgtctccgcgatctgattttacgacaatagagtttc3865gggttttgtcccattccagtttgaaaataaaggtccgtcttttaagtttgctggatcgat3925aaacctgtgaagattgagtctagtcgatttattggatgatccattcttcatcgttttttt3985cttgcttcgaagttctgtataaccagatttgtctgtgtgcgattgtcattacctagccgt4045gtatcgagaactagggttttcgagtcaattttgccccttttggttatatctggttcgata4105acgattcatctggattagggttttaagtggtgacgtttagtattccaatttcttcaaaat4165ttagttatggataatgaaaatccccaattgactgttcaatttcttgttaaatgcgcagat4225cacaatggcttcgatctcctcctcagtcgcgaccgttagccggaccgcccctgctcaggc4285caacatggtggctccgttcaccggccttaagtccaacgccgccttccccaccaccaagaa4345ggctaacgacttctccacccttcccagcaacggtggaagagttcaatgtatgcaggtgtg4405gccggcctacggcaacaagaagttcgagacgctgtcgtacctgccgccgctgtctatggc4465gcccaccgtgatgatggcctcgtcggccaccgccgtcgctccgttccaggggctcaagtc4525caccgccagcctccccgtcgcccgccgctcctccagaagcctcggcaacgtcagcaacgg4585cggaaggatccggtgcatggccggcgccgaggagatcgtgctgcagcccatc4637metalaglyalaglugluilevalleuglnproile360365370aaggagatctccggcaccgtcaagctgccggggtccaagtcgctttcc4685lysgluileserglythrvallysleuproglyserlysserleuser375380385aaccggatcctcctactcgccgccctgtccgaggggacaacagtggtt4733asnargileleuleuleualaalaleusergluglythrthrvalval390395400gataacctgctgaacagtgaggatgtccactacatgctcggggccttg4781aspasnleuleuasnsergluaspvalhistyrmetleuglyalaleu405410415aggactcttggtctctctgtcgaagcggacaaagctgccaaaagagct4829argthrleuglyleuservalglualaasplysalaalalysargala420425430gtagttgttggctgtggtggaaagttcccagttgaggatgctaaagag4877valvalvalglycysglyglylyspheprovalgluaspalalysglu435440445450gaagtgcagctcttcttggggaatgctggaatcgcaatgcggtccttg4925gluvalglnleupheleuglyasnalaglyilealametargserleu455460465acagcagctgttactgctgctggtggaaatgcaacttacgtgcttgat4973thralaalavalthralaalaglyglyasnalathrtyrvalleuasp470475480ggagtaccaagaatgagggagagacccattggcgacttggttgtcgga5021glyvalproargmetarggluargproileglyaspleuvalvalgly485490495ttgaagcagcttggtgcagatgttgattgtttccttggcactgactgc5069leulysglnleuglyalaaspvalaspcyspheleuglythraspcys500505510ccacctgttcgtgtcaatggaatcggagggctacctggtggcaaggtc5117proprovalargvalasnglyileglyglyleuproglyglylysval515520525530aagctgtctggctccatcagcagtcagtacttgagtgccttgctgatg5165lysleuserglyserileserserglntyrleuseralaleuleumet535540545gctgctcctttggctcttggggatgtggagattgaaatcattgataaa5213alaalaproleualaleuglyaspvalgluilegluileileasplys550555560ttaatctccattccgtacgtcgaaatgacattgagattgatggagcgt5261leuileserileprotyrvalglumetthrleuargleumetgluarg565570575tttggtgtgaaagcagagcattctgatagctgggacagattctacatt5309pheglyvallysalagluhisseraspsertrpaspargphetyrile580585590aagggaggtcaaaaatacaagtcccctaaaaatgcctatgttgaaggt5357lysglyglyglnlystyrlysserprolysasnalatyrvalglugly595600605610gatgcctcaagcgcaagctatttcttggctggtgctgcaattactgga5405aspalaserseralasertyrpheleualaglyalaalailethrgly615620625gggactgtgactgtggaaggttgtggcaccaccagtttgcagggtgat5453glythrvalthrvalgluglycysglythrthrserleuglnglyasp630635640gtgaagtttgctgaggtactggagatgatgggagcgaaggttacatgg5501vallysphealagluvalleuglumetmetglyalalysvalthrtrp645650655accgagactagcgtaactgttactggcccaccgcgggagccatttggg5549thrgluthrservalthrvalthrglyproproarggluprophegly660665670aggaaacacctcaaggcgattgatgtcaacatgaacaagatgcctgat5597arglyshisleulysalaileaspvalasnmetasnlysmetproasp675680685690gtcgccatgactcttgctgtggttgccctctttgccgatggcccgaca5645valalametthrleualavalvalalaleuphealaaspglyprothr695700705gccatcagagacgtggcttcctggagagtaaaggagaccgagaggatg5693alaileargaspvalalasertrpargvallysgluthrgluargmet710715720gttgcgatccggacggagctaaccaagctgggagcatctgttgaggaa5741valalaileargthrgluleuthrlysleuglyalaservalgluglu725730735gggccggactactgcatcatcacgccgccggagaagctgaacgtgacg5789glyproasptyrcysileilethrproproglulysleuasnvalthr740745750gcgatcgacacgtacgacgaccacaggatggcgatggctttctccctt5837alaileaspthrtyraspasphisargmetalametalapheserleu755760765770gccgcctgtgccgaggtccccgtcaccatccgggaccctgggtgcacc5885alaalacysalagluvalprovalthrileargaspproglycysthr775780785cggaagaccttccccgactacttcgatgtgctgagcactttcgtcaag5933arglysthrpheproasptyrpheaspvalleuserthrphevallys790795800aattaagctctagaactagtggatcccccgatccgcgtttgtgttttctgggtttc5989asntcacttaagcgtctgcgttttacttttgtattgggtttggcgtttagtagtttgcggtag6049cgttcttgttatgtgtaattacgctttttcttcttgcttcagcagtttcggttgaaatat6109aaatcgaatcaagtttcactttatcagcgttgttttaaattttggcattaaattggtgaa6169aattgcttcaattttgtatctaaatagaagagacaacatgaaattcgacttttgacctca6229aatcttcgaacatttatttcctgatttcacgatggatgaggataacgaaagggcggttcc6289tatgtccgggaaagttcccgtagaagacaatgagcaaagctactgaaacgcggacacgac6349gtcgcattggtacggatatgagttaaaccgactcaattcctttattaagacataaaccga6409ttttggttaaagtgtaacagtgagctgatataaaaccgaaacaaaccggtacaagtttga6469ttgagcaacttgatgacaaacttcagaattttggttattgaatgaaaatcatagtctaat6529cgtaaaaaatgtacagaagaaaagctagagcagaacaaagattctatattctggttccaa6589tttatcatcgctttaacgtccctcagatttgatcgggaaacataacttcgtatagcatac6649attatacgaagttatcaaaacgtcgtgagacagtttggttaactataacggtcctaaggt6709agcgatcgaggcattacggcattacggcactcgcgagggtccgaatctatgtcgggtgcg6769gagaaagaggtaatgaaatggcaatttacaattgaatatatcctgccgccgctgccgctt6829tgcacccggtggagcttgcatgttggtttctacgcagaactgagccggttaggcagataa6889tttccattgagaactgagccatgtgcaccttccccccaacacggtgagcgacggggcaac6949ggagtgatccacatgggacttttaaacatcatccgtcggatggcgttgcgagagaagcag7009tcgatccgtgagatcagccgacgcaccgggcaggcgcgcaacacgatcgcaaagtatttg7069aacgcaggtacaatcgagccgacgttcacggtaccggaacgaccaagcaagctagcttag7129taaagccctcgctagattttaatgcggatgttgcgattacttcgccaactattgcgataa7189caagaaaaagccagcctttcatgatatatctcccaatttgtgtagggcttattatgcacg7249cttaaaaataataaaagcagacttgacctgatagtttggctgtgagcaattatgtgctta7309gtgcatctaacgcttgagttaagccgcgccgcgaagcggcgtcggcttgaacgaattgtt7369agacattatttgccgactaccttggtgatctcgcctttcacgtagtggacaaattcttcc7429aactgatctgcgcgcgaggccaagcgatcttcttcttgtccaagataagcctgtctagct7489tcaagtatgacgggctgatactgggccggcaggcgctccattgcccagtcggcagcgaca7549tccttcggcgcgattttgccggttactgcgctgtaccaaatgcgggacaacgtaagcact7609acatttcgctcatcgccagcccagtcgggcggcgagttccatagcgttaaggtttcattt7669agcgcctcaaatagatcctgttcaggaaccggatcaaagagttcctccgccgctggacct7729accaaggcaacgctatgttctcttgcttttgtcagcaagatagccagatcaatgtcgatc7789gtggctggctcgaagatacctgcaagaatgtcattgcgctgccattctccaaattgcagt7849tcgcgcttagctggataacgccacggaatgatgtcgtcgtgcacaacaatggtgacttct7909acagcgcggagaatctcgctctctccaggggaagccgaagtttccaaaaggtcgttgatc7969aaagctcgccgcgttgtttcatcaagccttacggtcaccgtaaccagcaaatcaatatca8029ctgtgtggcttcaggccgccatccactgcggagccgtacaaatgtacggccagcaacgtc8089ggttcgagatggcgctcgatgacgccaactacctctgatagttgagtcgatacttcggcg8149atcaccgcttccctcatgatgtttaactttgttttagggcgactgccctgctgcgtaaca8209tcgttgctgctccataacatcaaacatcgacccacggcgtaacgcgcttgctgcttggat8269gcccgaggcatagactgtaccccaaaaaaacagtcataacaagccatgaaaaccgccact8329gcgccgttaccaccgctgcgttcggtcaaggttctggaccagttgcgtgagcgcatacgc8389tacttgcattacagcttacgaaccgaacaggcttatgtccactgggttcgtgccttcatc8449cgtttccacggtgtgcgtcacccggcaaccttgggcagcagcgaagtcgaggcatttctg8509tcctggctggcgaacgagcgcaaggtttcggtctccacgcatcgtcaggcattggcggcc8569ttgctgttcttctacggcaagtgctgtgcacggatctgccctggcttc8617<210>21<211>358<212>prt<213>人工的<220><223>合成的構建體<400>21metalaaspleutyrgluasnprometglyleumetglyphegluphe151015ilegluphealaserprothrproglythrleugluproilepheglu202530ilemetglyphethrlysvalalathrhisargserlysasnvalhis354045leutyrargglnglygluileasnleuileleuasnasngluproasn505560serilealasertyrphealaalagluhisglyproservalcysgly65707580metalapheargvallysaspserglnlysalatyrasnargalaleu859095gluleuglyalaglnproilehisileaspthrglyprometgluleu100105110asnleuproalailelysglyileglyglyalaproleutyrleuile115120125aspargpheglygluglyserseriletyraspileaspphevaltyr130135140leugluglyvalgluargasnprovalglyalaglyleulysvalile145150155160asphisleuthrhisasnvaltyrargglyargmetvaltyrtrpala165170175asnphetyrglulysleupheasnpheargglualaargtyrpheasp180185190ilelysglyglutyrthrglyleuthrserlysalametseralapro195200205aspglymetileargileproleuasnglugluserserlysglyala210215220glyglnilegluglupheleumetglnpheasnglygluglyilegln225230235240hisvalalapheleuthraspaspleuvallysthrtrpaspalaleu245250255lyslysileglymetargphemetthralaproproaspthrtyrtyr260265270glumetleugluglyargleuproasphisglygluprovalaspgln275280285leuglnalaargglyileleuleuaspglyserservalgluglyasp290295300lysargleuleuleuglnilephesergluthrleumetglyproval305310315320phepheglupheileglnarglysglyaspaspglypheglyglutrp325330335asnphelysalaleuphegluserilegluargaspglnvalargarg340345350glyvalleuthralaasp355<210>22<211>445<212>prt<213>人工的<220><223>合成的構建體<400>22metalaglyalaglugluilevalleuglnproilelysgluileser151015glythrvallysleuproglyserlysserleuserasnargileleu202530leuleualaalaleusergluglythrthrvalvalaspasnleuleu354045asnsergluaspvalhistyrmetleuglyalaleuargthrleugly505560leuservalglualaasplysalaalalysargalavalvalvalgly65707580cysglyglylyspheprovalgluaspalalysglugluvalglnleu859095pheleuglyasnalaglyilealametargserleuthralaalaval100105110thralaalaglyglyasnalathrtyrvalleuaspglyvalproarg115120125metarggluargproileglyaspleuvalvalglyleulysglnleu130135140glyalaaspvalaspcyspheleuglythraspcysproprovalarg145150155160valasnglyileglyglyleuproglyglylysvallysleusergly165170175serileserserglntyrleuseralaleuleumetalaalaproleu180185190alaleuglyaspvalgluilegluileileasplysleuileserile195200205protyrvalglumetthrleuargleumetgluargpheglyvallys210215220alagluhisseraspsertrpaspargphetyrilelysglyglygln225230235240lystyrlysserprolysasnalatyrvalgluglyaspalaserser245250255alasertyrpheleualaglyalaalailethrglyglythrvalthr260265270valgluglycysglythrthrserleuglnglyaspvallyspheala275280285gluvalleuglumetmetglyalalysvalthrtrpthrgluthrser290295300valthrvalthrglyproproargglupropheglyarglyshisleu305310315320lysalaileaspvalasnmetasnlysmetproaspvalalametthr325330335leualavalvalalaleuphealaaspglyprothralaileargasp340345350valalasertrpargvallysgluthrgluargmetvalalailearg355360365thrgluleuthrlysleuglyalaservalglugluglyproasptyr370375380cysileilethrproproglulysleuasnvalthralaileaspthr385390395400tyraspasphisargmetalametalapheserleualaalacysala405410415gluvalprovalthrileargaspproglycysthrarglysthrphe420425430proasptyrpheaspvalleuserthrphevallysasn435440445<210>23<211>13080<212>dna<213>人工序列<220><223>載體<400>23aattacaacggtatatatcctgccagtactgggccccctcgagggcgatcgctacgtacc60tgcaggcccgggttaattaaggcccgatcaaatctgagggacgttaaagcgatgataaat120tggaaccagaatatagaatctttgttctgctctagcttttcttctgtacattttttacga180ttagactatgattttcattcaataaccaaaattctgaagtttgtcatcaagttgctcaat240caaacttgtaccggtttgtttcggttttatatcagctcactgttacactttaaccaaaat300cggtttatgtcttaataaaggaattgagtcggtttaactcatatccgtaccaatgcgacg360tcgtgtccgcgtttcagtagctttgctcattgtcttctacgggaactttcccggacatag420gaaccgccctttcgttatcctcatccatcgtgaaatcaggaaataaatgttcgaagattt480gaggtcaaaagtcgaatttcatgttgtctcttctatttagatacaaaattgaagcaattt540tcaccaatttaatgccaaaatttaaaacaacgctgataaagtgaaacttgattcgattta600tatttcaaccgaaactgctgaagcaagaagaaaaagcgtaattacacataacaagaacgc660taccgcaaactactaaacgccaaacccaatacaaaagtaaaacgcagacgcttaagtgag720aaacccagaaaacacaaacgcggatcgggcgacgcgtgctagcttttaatcagcggtcaa780aacccctctacgaacttgatccctctctatcgattcgaaaagtgctttgaagttccattc840tccgaagccatcgtcccctttacgctgaatgaactcaaagaaaacgggtcccatgagagt900ctcggaaaagatttgtaacaggagcctcttatcaccctcaaccgaagaaccatcgagaag960tatacccctggcttgcagttggtccacgggttccccatgatcaggcaggcgtccttctag1020catctcatagtaagtatcgggaggtgcagtcatgaatctcatacctatcttcttcaatgc1080atcccaggttttcacaagatcatcagtcagaaaagcgacatgctgaatgccttcaccatt1140gaactgcataagaaactcttcgatttgtcctgcacctttgcttgactcttcgttgagtgg1200aatccttatcataccatctggagcgctcatagcctttgatgtgagaccagtatactctcc1260tttgatgtcaaagtatctagcttccctaaagttgaagagtttctcatagaagttagccca1320gtataccattcgtccacgatagacgttgtgtgtcaaatgatcaataaccttcaagccagc1380accgacaggatttcgttccacgccttcaagatagacgaaatctatgtcgtagatagaact1440gccctctccgaatctgtcgataagatacaacggtgcaccaccgatgcctttgatggctgg1500aagattcaattccataggtccagtgtcaatgtgaataggctgagctccaagttctaatgc1560cctattgtaggctttctgggaatccttaactcgaaatgccatgccacaaacggatggtcc1620atgttcggcagcaaagtagcttgctatagagttaggttcgttgttcaagatcaggttaat1680ctctccttgccgatataggtgcacgttcttactgcgatgtgtagctaccttagtaaaccc1740cataatctcaaagatgggttcaagggtcccaggtgtaggagaagcaaactcaatgaactc1800gaaacccatgagtcccataggattctcgtatagatcagccatgcatctaatcctacctcc1860gttgctaacattccccagcgatctggaagatctccgagcaactggcaaactagcagtact1920cttcaacccttgaaatggtgccacagctgtggcagagctggccatcatcacagtgggagc1980catagatagcggaggtaagtaggataaggtctcaaacttcttgttgccataggctggcca2040gacttgcatatactgtactctcccaccgttgctcggaagtgttgagaagtcattagcctt2100cttagtggttggaaatgcagcatttgacttaagacctgtgaacggagccaccatattagc2160ttgggctggtgcggtacgtgaaacagtggcaactgaagatgaaatactagccatggtttt2220ggacaaacttacaaatttctctgaagttgtatcctcagtacttcaaagaaaatagcttac2280accaaattttttcttgttttcacaaatgccgaacttggttccttatataggaaaactcaa2340gggcaaaaatgacacggaaaaatataaaaggataagtagtgggggataagattcctttgt2400gataaggttactttccgcccttacattttccaccttacatgtgtcctctatgtctctttc2460acaatcaccgaccttatcttcttcttttcattgttgtcgtcagtgcttacgtcttcaaga2520ttcttttcttcgcctggttcttctttttcaatttctacgtattcttcttcgtattctggc2580agtataggatcttgtatctgtacattcttcatttttgaacataggttgcatatgtgccgc2640atattgatctgcttcttgctgagctcacataatacttccatagtttttcccgtaaacatt2700ggattcttgatgctacatcttggataattaccttcgcggccgcttggcgcgccgaattcg2760atatcattaccctgttatccctaaagcttattaatataacttcgtatagcatacattata2820cgaagttatgtttgtcgaggagaaatatgagtcgaggcatggatacactaagttcccctg2880aagtgagcatgatctttgatgctgagatgattcccagagcaagatagtttgtgctgcaag2940tgacacaattgtaatgaaaccaccactcaacgaatttacttgtggctttgacatgtcgtg3000tgctctgtttgtatttgtgagtgccggttggtaattatttttgttaatgtgattttaaaa3060cctcttatgtaaatagttactttatctattgaagtgtgttcttgtggtctatagtttctc3120aaagggaaattaaaatgttgacatcccatttacaattgataacttggtatacacaaactt3180tgtaaatttggtgatatttatggtcgaaagaaggcaatacccattgtatgttccaatatc3240aatatcaatacgataacttgataatactaacatatgattgtcattgtttttccagtatca3300atatacattaagctactacaaaattagtataaatcactatattataaatctttttcggtt3360gtaacttgtaattcgtgggtttttaaaataaaagcatgtgaaaattttcaaataatgtga3420tggcgcaattttattttccgagttccaaaatattgccgcttcattaccctaatttgtggc3480gccacatgtaaaacaaaagacgattcttagtggctatcactgccatcacgcggatcacta3540atatgaaccgtcgattaaaacagatcgacggtttatacatcattttattgtacacacgga3600tcgatatctcagccgttagatttaatatgcgatctgattgctcaaaaaatagactctccg3660tctttgcctataaaaacaatttcacatctttctcacccaaatctactcttaaccgttctt3720cttcttctacagacatcaatttctctcgactctagaggatccaagcttatcgatttcgaa3780cccctcaggcgaagaacaggtatgatttgtttgtaattagatcaggggtttaggtctttc3840cattactttttaatgttttttctgttactgtctccgcgatctgattttacgacaatagag3900tttcgggttttgtcccattccagtttgaaaataaaggtccgtcttttaagtttgctggat3960cgataaacctgtgaagattgagtctagtcgatttattggatgatccattcttcatcgttt4020ttttcttgcttcgaagttctgtataaccagatttgtctgtgtgcgattgtcattacctag4080ccgtgtatcgagaactagggttttcgagtcaattttgccccttttggttatatctggttc4140gataacgattcatctggattagggttttaagtggtgacgtttagtattccaatttcttca4200aaatttagttatggataatgaaaatccccaattgactgttcaatttcttgttaaatgcgc4260agatcacaatggcttcgatctcctcctcagtcgcgaccgttagccggaccgcccctgctc4320aggccaacatggtggctccgttcaccggccttaagtccaacgccgccttccccaccacca4380agaaggctaacgacttctccacccttcccagcaacggtggaagagttcaatgtatgcagg4440tgtggccggcctacggcaacaagaagttcgagacgctgtcgtacctgccgccgctgtcta4500tggcgcccaccgtgatgatggcctcgtcggccaccgccgtcgctccgttccaggggctca4560agtccaccgccagcctccccgtcgcccgccgctcctccagaagcctcggcaacgtcagca4620acggcggaaggatccggtgcatggccggcgccgaggagatcgtgctgcagcccatcaagg4680agatctccggcaccgtcaagctgccggggtccaagtcgctttccaaccggatcctcctac4740tcgccgccctgtccgaggggacaacagtggttgataacctgctgaacagtgaggatgtcc4800actacatgctcggggccttgaggactcttggtctctctgtcgaagcggacaaagctgcca4860aaagagctgtagttgttggctgtggtggaaagttcccagttgaggatgctaaagaggaag4920tgcagctcttcttggggaatgctggaatcgcaatgcggtccttgacagcagctgttactg4980ctgctggtggaaatgcaacttacgtgcttgatggagtaccaagaatgagggagagaccca5040ttggcgacttggttgtcggattgaagcagcttggtgcagatgttgattgtttccttggca5100ctgactgcccacctgttcgtgtcaatggaatcggagggctacctggtggcaaggtcaagc5160tgtctggctccatcagcagtcagtacttgagtgccttgctgatggctgctcctttggctc5220ttggggatgtggagattgaaatcattgataaattaatctccattccgtacgtcgaaatga5280cattgagattgatggagcgttttggtgtgaaagcagagcattctgatagctgggacagat5340tctacattaagggaggtcaaaaatacaagtcccctaaaaatgcctatgttgaaggtgatg5400cctcaagcgcaagctatttcttggctggtgctgcaattactggagggactgtgactgtgg5460aaggttgtggcaccaccagtttgcagggtgatgtgaagtttgctgaggtactggagatga5520tgggagcgaaggttacatggaccgagactagcgtaactgttactggcccaccgcgggagc5580catttgggaggaaacacctcaaggcgattgatgtcaacatgaacaagatgcctgatgtcg5640ccatgactcttgctgtggttgccctctttgccgatggcccgacagccatcagagacgtgg5700cttcctggagagtaaaggagaccgagaggatggttgcgatccggacggagctaaccaagc5760tgggagcatctgttgaggaagggccggactactgcatcatcacgccgccggagaagctga5820acgtgacggcgatcgacacgtacgacgaccacaggatggcgatggccttctcccttgccg5880cctgtgccgaggtccccgtcaccatccgggaccctgggtgcacccggaagaccttccccg5940actacttcgatgtgctgagcactttcgtcaagaattaagctctagaactagtggatcccc6000cgatccgcgtttgtgttttctgggtttctcacttaagcgtctgcgttttacttttgtatt6060gggtttggcgtttagtagtttgcggtagcgttcttgttatgtgtaattacgctttttctt6120cttgcttcagcagtttcggttgaaatataaatcgaatcaagtttcactttatcagcgttg6180ttttaaattttggcattaaattggtgaaaattgcttcaattttgtatctaaatagaagag6240acaacatgaaattcgacttttgacctcaaatcttcgaacatttatttcctgatttcacga6300tggatgaggataacgaaagggcggttcctatgtccgggaaagttcccgtagaagacaatg6360agcaaagctactgaaacgcggacacgacgtcgcattggtacggatatgagttaaaccgac6420tcaattcctttattaagacataaaccgattttggttaaagtgtaacagtgagctgatata6480aaaccgaaacaaaccggtacaagtttgattgagcaacttgatgacaaacttcagaatttt6540ggttattgaatgaaaatcatagtctaatcgtaaaaaatgtacagaagaaaagctagagca6600gaacaaagattctatattctggttccaatttatcatcgctttaacgtccctcagatttga6660tcgggaaacataacttcgtatagcatacattatacgaagttatcaaaacgtcgtgagaca6720gtttggttaactataacggtcctaaggtagcgatcgaggcattacggcattacggcactc6780gcgagggtccgaatctatgtcgggtgcggagaaagaggtaatgaaatggcaatttacaat6840tgaatatatcctgccgccgctgccgctttgcacccggtggagcttgcatgttggtttcta6900cgcagaactgagccggttaggcagataatttccattgagaactgagccatgtgcaccttc6960cccccaacacggtgagcgacggggcaacggagtgatccacatgggacttttaaacatcat7020ccgtcggatggcgttgcgagagaagcagtcgatccgtgagatcagccgacgcaccgggca7080ggcgcgcaacacgatcgcaaagtatttgaacgcaggtacaatcgagccgacgttcacggt7140accggaacgaccaagcaagctagcttagtaaagccctcgctagattttaatgcggatgtt7200gcgattacttcgccaactattgcgataacaagaaaaagccagcctttcatgatatatctc7260ccaatttgtgtagggcttattatgcacgcttaaaaataataaaagcagacttgacctgat7320agtttggctgtgagcaattatgtgcttagtgcatctaacgcttgagttaagccgcgccgc7380gaagcggcgtcggcttgaacgaattgttagacattatttgccgactaccttggtgatctc7440gcctttcacgtagtggacaaattcttccaactgatctgcgcgcgaggccaagcgatcttc7500ttcttgtccaagataagcctgtctagcttcaagtatgacgggctgatactgggccggcag7560gcgctccattgcccagtcggcagcgacatccttcggcgcgattttgccggttactgcgct7620gtaccaaatgcgggacaacgtaagcactacatttcgctcatcgccagcccagtcgggcgg7680cgagttccatagcgttaaggtttcatttagcgcctcaaatagatcctgttcaggaaccgg7740atcaaagagttcctccgccgctggacctaccaaggcaacgctatgttctcttgcttttgt7800cagcaagatagccagatcaatgtcgatcgtggctggctcgaagatacctgcaagaatgtc7860attgcgctgccattctccaaattgcagttcgcgcttagctggataacgccacggaatgat7920gtcgtcgtgcacaacaatggtgacttctacagcgcggagaatctcgctctctccagggga7980agccgaagtttccaaaaggtcgttgatcaaagctcgccgcgttgtttcatcaagccttac8040ggtcaccgtaaccagcaaatcaatatcactgtgtggcttcaggccgccatccactgcgga8100gccgtacaaatgtacggccagcaacgtcggttcgagatggcgctcgatgacgccaactac8160ctctgatagttgagtcgatacttcggcgatcaccgcttccctcatgatgtttaactttgt8220tttagggcgactgccctgctgcgtaacatcgttgctgctccataacatcaaacatcgacc8280cacggcgtaacgcgcttgctgcttggatgcccgaggcatagactgtaccccaaaaaaaca8340gtcataacaagccatgaaaaccgccactgcgccgttaccaccgctgcgttcggtcaaggt8400tctggaccagttgcgtgagcgcatacgctacttgcattacagcttacgaaccgaacaggc8460ttatgtccactgggttcgtgccttcatccgtttccacggtgtgcgtcacccggcaacctt8520gggcagcagcgaagtcgaggcatttctgtcctggctggcgaacgagcgcaaggtttcggt8580ctccacgcatcgtcaggcattggcggccttgctgttcttctacggcaagtgctgtgcacg8640gatctgccctggcttcaggagatcggaagacctcggccgtccgggcgcttgccggtggtg8700ctgaccccggatgaagtctctagagctctagagggttcgcatcctcggttttctggaagg8760cgagcatcgtttgttcgcccagcttctgtatggaacgggcatgcggatcagtgagggttt8820gcaactgcgggtcaaggatctggatttcgatcacggcacgatcatcgtgcgggagggcaa8880gggctccaaggatcgggccttgatgttacccgagagcttggcacccagcctgcgcgagca8940gggatcgataccgtgcggctgcatgaaatcctggccggtttgtctgatgccaagctggcg9000gcctggccggccagcttggccgctgaagaaaccgagcgccgccgtctaaaaaggtgatgt9060gtatttgagtaaaacagcttgcgtcatgcggtcgctgcgtatatgatgcgatgagtaaat9120aaacaaatacgcaaggggaacgcatgaaggttatcgctgtacttaaccagaaaggcgggt9180caggcaagacgaccatcgcaacccatctagcccgcgccctgcaactcgccggggccgatg9240ttctgttagtcgattccgatccccagggcagtgcccgcgattgggcggccgtgcgggaag9300atcaaccgctaaccgttgtcggcatcgaccgcccgacgattgaccgcgacgtgaaggcca9360tcggccggcgcgacttcgtagtgatcgacggagcgccccaggcggcggacttggctgtgt9420ccgcgatcaaggcagccgacttcgtgctgattccggtgcagccaagcccttacgacatat9480gggccaccgccgacctggtggagctggttaagcagcgcattgaggtcacggatggaaggc9540tacaagcggcctttgtcgtgtcgcgggcgatcaaaggcacgcgcatcggcggtgaggttg9600ccgaggcgctggccgggtacgagctgcccattcttgagtcccgtatcacgcagcgcgtga9660gctacccaggcactgccgccgccggcacaaccgttcttgaatcagaacccgagggcgacg9720ctgcccgcgaggtccaggcgctggccgctgaaattaaatcaaaactcatttgagttaatg9780aggtaaagagaaaatgagcaaaagcacaaacacgctaagtgccggccgtccgagcgcacg9840cagcagcaaggctgcaacgttggccagcctggcagacacgccagccatgaagcgggtcaa9900ctttcagttgccggcggaggatcacaccaagctgaagatgtacgcggtacgccaaggcaa9960gaccattaccgagctgctatctgaatacatcgcgcagctaccagagtaaatgagcaaatg10020aataaatgagtagatgaattttagcggctaaaggaggcggcatggaaaatcaagaacaac10080caggcaccgacgccgtggaatgccccatgtgtggaggaacgggcggttggccaggcgtaa10140gcggctgggttgtctgccggccctgcaatggcactggaacccccaagcccgaggaatcgg10200cgtgacggtcgcaaaccatccggcccggtacaaatcggcgcggcgctgggtgatgacctg10260gtggagaagttgaaggccgcgcaggccgcccagcggcaacgcatcgaggcagaagcacgc10320cccggtgaatcgtggcaagcggccgctgatcgaatccgcaaagaatcccggcaaccgccg10380gcagccggtgcgccgtcgattaggaagccgcccaagggcgacgagcaaccagattttttc10440gttccgatgctctatgacgtgggcacccgcgatagtcgcagcatcatggacgtggccgtt10500ttccgtctgtcgaagcgtgaccgacgagctggcgaggtgatccgctacgagcttccagac10560gggcacgtagaggtttccgcagggccggccggcatggccagtgtgtgggattacgacctg10620gtactgatggcggtttcccatctaaccgaatccatgaaccgataccgggaagggaaggga10680gacaagcccggccgcgtgttccgtccacacgttgcggacgtactcaagttctgccggcga10740gccgatggcggaaagcagaaagacgacctggtagaaacctgcattcggttaaacaccacg10800cacgttgccatgcagcgtacgaagaaggccaagaacggccgcctggtgacggtatccgag10860ggtgaagccttgattagccgctacaagatcgtaaagagcgaaaccgggcggccggagtac10920atcgagatcgagctagctgattggatgtaccgcgagatcacagaaggcaagaacccggac10980gtgctgacggttcaccccgattactttttgatcgatcccggcatcggccgttttctctac11040cgcctggcacgccgcgccgcaggcaaggcagaagccagatggttgttcaagacgatctac11100gaacgcagtggcagcgccggagagttcaagaagttctgtttcaccgtgcgcaagctgatc11160gggtcaaatgacctgccggagtacgatttgaaggaggaggcggggcaggctggcccgatc11220ctagtcatgcgctaccgcaacctgatcgagggcgaagcatccgccggttcctaatgtacg11280gagcagatgctagggcaaattgccctagcaggggaaaaaggtcgaaaaggtctctttcct11340gtggatagcacgtacattgggaacccaaagccgtacattgggaaccggaacccgtacatt11400gggaacccaaagccgtacattgggaaccggtcacacatgtaagtgactgatataaaagag11460aaaaaaggcgatttttccgcctaaaactctttaaaacttattaaaactcttaaaacccgc11520ctggcctgtgcataactgtctggccagcgcacagccgaagagctgcaaaaagcgcctacc11580cttcggtcgctgcgctccctacgccccgccgcttcgcgtcggcctatcgcggccgctggc11640cgctcaaaaatggctggcctacggccaggcaatctaccagggcgcggacaagccgcgccg11700tcgccactcgaccgccggcgcccacatcaaggcaccctgcctcgcgcgtttcggtgatga11760cggtgaaaacctctgacacatgcagctcccggagacggtcacagcttgtctgtaagcgga11820tgccgggagcagacaagcccgtcagggcgcgtcagcgggtgttggcgggtgtcggggcgc11880agccatgacccagtcacgtagcgatagcggagtgtatactggcttaactatgcggcatca11940gagcagattgtactgagagtgcaccatatgcggtgtgaaataccgcacagatgcgtaagg12000agaaaataccgcatcaggcgctcttccgcttcctcgctcactgactcgctgcgctcggtc12060gttcggctgcggcgagcggtatcagctcactcaaaggcggtaatacggttatccacagaa12120tcaggggataacgcaggaaagaacatgtgagcaaaaggccagcaaaaggccaggaaccgt12180aaaaaggccgcgttgctggcgtttttccataggctccgcccccctgacgagcatcacaaa12240aatcgacgctcaagtcagaggtggcgaaacccgacaggactataaagataccaggcgttt12300ccccctggaagctccctcgtgcgctctcctgttccgaccctgccgcttaccggatacctg12360tccgcctttctcccttcgggaagcgtggcgctttctcatagctcacgctgtaggtatctc12420agttcggtgtaggtcgttcgctccaagctgggctgtgtgcacgaaccccccgttcagccc12480gaccgctgcgccttatccggtaactatcgtcttgagtccaacccggtaagacacgactta12540tcgccactggcagcagccactggtaacaggattagcagagcgaggtatgtaggcggtgct12600acagagttcttgaagtggtggcctaactacggctacactagaaggacagtatttggtatc12660tgcgctctgctgaagccagttaccttcggaaaaagagttggtagctcttgatccggcaaa12720caaaccaccgctggtagcggtggtttttttgtttgcaagcagcagattacgcgcagaaaa12780aaaggatctcaagaagatccggaaaacgcaagcgcaaagagaaagcaggtagcttgcagt12840gggcttacatggcgatagctagactgggcggttttatggacagcaagcgaaccggaattg12900ccagattcgaagctcggtcccgtgggtgttctgtcgtctcgttgtacaacgaaatccatt12960cccattccgcgctcaagatggcttcccctcggcagttcatcagggctaaatcaatctagc13020cgacttgtccggtgaaatgggctgcactccaacagaaacaatcaaacaaacatacacagc13080當前第1頁12
相關技術
網友詢問留言
已有0條留言
- 還沒有人留言評論。精彩留言會獲得點贊!
1