一種單核苷酸多態性位點在輔助鑒定大豆花葉病毒抗性中的應用的制作方法
本發明涉及大豆抗病育種和分子生物學技術領域,具體涉及一種與大豆花葉病毒病相關snp位點的應用及大豆花葉病毒抗性的檢測方法。
背景技術:
大豆花葉病毒(soybeanmosaicvirus,smv)是一種世界性大豆病害,對大豆生產影響最大的病毒之一,嚴重影響大豆的產量和品質。南京農業大學將我國的smv株系劃分為sc1-sc21,其中sc3和sc7是黃淮和長江流域大豆產區最流行的株系。大豆花葉病毒分布廣、危害大,化學藥劑難以防治,培育和篩選抗病品種是解決大豆病毒性的最有效經濟途徑。
單核苷酸多態性(singlenucleotidepolymorphism,snp),是指在基因組水平上由單個核苷酸的變異所引起的dna序列多態性,snp所表現的多態性只涉及到單個堿基的變異,這種變異可由單個堿基的轉換(transition)或顛換(transversion)所引起,也可由堿基的插入或缺失所致,但通常所說的snp并不包括后兩種情況。
大豆基因組雖然早在2010年已經完成測序,但各個基因的功能并沒有完全闡明。如果能從中找出與大豆花葉病毒抗性相關的位點,并利用該位點開發分子標記,利用分子標記輔助育種,將能大大提高育種效率。近年,單核苷酸多態性(snp)越來越受到科研人員的重視,同時也發現許多snp與作物的重要農藝性狀和抗性具有一定的關聯性,程浩(2008)博士畢業論文(大豆異黃酮合酶,黃烷酮3-羥化酶基因snps與種子異黃酮含量及抗逆性的關聯分析)報道稱大豆ifs1,ifs2和f3h基因中都存在與大豆對花葉病毒抗性顯著相關的snps。如ifs1基因中與大豆對smvsc-3與smvsc-7抗性顯著相關的snps分別為14和2個;在ifs2基因中發現與大豆對smvsc-3與smvsc-7抗性顯著相關的為indel(1bp)。在f3h基因中發現與大豆對smvsc-3和sc-7抗性顯著相關的snps為7個和1個。因此,發掘和利用大豆中重要的snp位點具有重要的現實和理論意義。
技術實現要素:
本發明的目的是提供一種glyma.07g250300中與大豆花葉病毒病相關snp位點發掘及應用,本發明的目的可以通過以下技術實現:
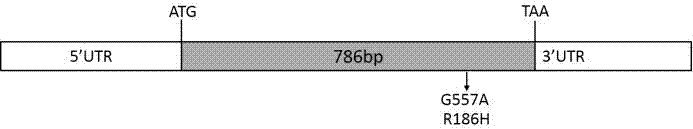
一種單核苷酸多態性位點在輔助鑒定大豆花葉病毒抗性中的應用,該點位于大豆基因組glyma.07g250300基因編碼區開放閱讀框第557位。
進一步,上述單核苷酸多態性位點的應用是指:對大豆基因組glyma.07g250300基因編碼區開放閱讀框第557位核苷酸進行基因型鑒定,若第557位核苷酸殘基為“a”,則認為大豆材料抗花葉病毒;若第557位核苷酸殘基為“g”,則認為大豆材料對花葉病毒敏感。
進一步,上述單核苷酸多態性位點的應用,其具體步驟如下:
1)根據大豆基因組glyma.07g250300基因編碼區內開放閱讀框(orf)設計上游引物和下游引物,并以大豆基因組dna為模板,進行pcr擴增;
2)對pcr擴增產物進行測序,若glyma.07g250300基因編碼區內開放閱讀框第557位核苷酸為“a”,則認為大豆材料抗花葉病毒;若第557位核苷酸為“g”,則認為該大豆材料對花葉病毒敏感。
進一步,上述單核苷酸多態性位點的應用中,所述上游引物位于開放閱讀框
(5‵→3‵)第71位核苷酸殘基開始,第90位核苷酸殘基結束;下游引物從開放讀框第635位核苷酸殘基開始,第657位核苷酸殘基結束。
進一步,所述上游引物和下游引物核苷酸序列分別如seqidno.1和seqidno.2所示;
pcr擴增體系:2×phantamaxbuffer25μl;dntpmix(10mmeach)1μl;上游引物4μl(10μm);下游引物4μl(10μm);phantamaxsuper-fidelitydnapolymerase(1u/μl);模板dna4μl(20ng/μl);ddh2o補足到50μl;
pcr擴增程序:95℃預變性3min;然后95℃變性15s,56℃退火45s,72℃延伸30s,35個循環;最后72℃延伸10min,4℃保溫;
pcr反應結束后,將所得pcr產物(786bp)進行1%瓊脂糖凝膠電泳,然后測序。
一種檢測單核苷酸多態性或基因型的物質在制備大豆花葉病毒抗性檢測試劑中的應用,所述檢測單核苷酸多態性或基因型的物質是指:檢測大豆基因組中glyma.07g250300基因編碼區開放閱讀框第557位單核苷酸多態性或基因型的物質。
進一步,所述檢測單核苷酸多態性或基因型的物質是指根據大豆基因組中glyma.07g250300基因編碼區開放閱讀框所設計的上游引物和下游引物,所述大豆花葉病毒抗性檢測試劑是指pcr檢測試劑。
進一步,該pcr檢測試劑(總體積50μl)包括:2×phantamaxbuffer25μl;dntpmix(10mmeach)1μl;核苷酸序列如seqidno.1所示的上游引物4μl(10μm);核苷酸序列如seqidno.2所示的下游引物4μl(10μm);phantamaxsuper-fidelitydnapolymerase(1u/μl)1μl;模板dna4μl(20ng/μl);ddh2o為11μl。其中模板dna為大豆基因組dna。
進一步,本申請中,所述大豆花葉病毒為sc3株系或sc7株系。
本發明對重測序的302份大豆材料,分析glyma.07g250300基因座的開放閱讀框(orf)內含有1個有義突變snp位點,即第557位核苷酸位置有g/a(r/h)兩種類型;對9個材料的glyma.07g250300序列進行測定發現bb52和sa101材料中glyma.07g250300基因編碼區第557位核苷酸為“a”,其余大豆材料為“g”;利用南方普遍流行的兩種病毒株系sc3和sc7分別對所用大豆材料分別接種鑒定,發現bb52和sa101對兩種病毒同時表現出抗性;而其余材料則均對兩種病毒表現出敏感性,結合snp位點結果,bb52和sa101在第557位a堿基,而對兩種病毒表現出抗性,其它材料均為g堿基,而表現出敏感性,為大豆抗病毒病分子標記輔助育種奠定了基礎。
在具體應用中,可以根據大豆基因組中glyma.07g250300基因開放閱讀框第557位核苷酸設計引物并制備大豆花葉病毒抗性檢測試劑(如pcr試劑),或以其他本領域常規方法對該位點的基因型進行檢測,若該位點基因型為“g”,則大豆材料表現出敏感性,當位點基因型為“a”時,則大豆材料表現出抗性;通過上述方法,在大豆苗期即可對大豆材料的花葉病毒抗性進行評價,以提高抗病育種效率,在實際育種和生產中具有廣闊的應用前景。
附圖說明
圖1為大豆基因組glyma.07g250300基因編碼區snp位點分析結果示意圖。
圖2大豆基因組glyma.07g250300基因序列在不同大豆材料中的多重比對分析結果示意圖。
具體實施方式
下述實施例中所使用的試驗方法無特殊說明,均為常規方法。實施例中所用的材料、試劑等,如無特殊說明,均可從商業途徑得到。
1、實施例所用大豆材料
以下實施例所用大豆材料包括willimas82(對應序列為gmzfp3),野生大豆bb52(gszfp3),sa001,sa002,sa025,sa062,sa101,sa162,sa189,sa242,willimas82,bb52為江蘇省農業科學院農業資源與環境研究所鹽土農業研究中心保存,其它材料均由南京農業大學大豆研究所提供。
2.以下實施例涉及到生物試劑及引物合成與測序
dna提取試劑盒和dna凝膠回收試劑盒購于axygen公司;
高保真dna聚合酶phantamaxsuper-fidelitydnapolymerase購于vazyme公司;
takaradl2000bpdnamarker購于takara公司;
實施例所使用的pcr儀為lifetechnologies的proflexpcrsystem;所用引物和測序均由南京擎科生物有限公司合成和完成。
實施例涉及的引物序列:
seqidno.1:catgccaaaatcctgaacct;
seqidno.2:tggaacactatcagccaaagat;
實施例1大豆基因glyma.07g250300中snp位點的發掘
glyma.07g250300在大豆基因組數據庫(網址
實施例2大豆材料中glyma.07g250300基因編碼序列分析
試驗大豆材料為:willimas82(對應序列為gmzfp3);野生大豆bb52(gszfp3),sa001,sa002,sa025,sa062,sa101,sa162,sa189,sa242和willimas82。
分別利用ctab法(十六烷基三甲基溴化銨)提取上述10種類大豆(glycinemax(l.)merr.)品種幼苗葉片中基因組dna;分別以該dna為模板,用引物對進行pcr擴增,所用引物序列(用于擴增glyma.07g250300的開放閱讀框)為:
正向引物(f)如seqidno.1所示,反向引物如seqidno.2所示;
pcr反應體系:2×phantamaxbuffer25μl;dntpmix(10mmeach)1μl;上下游引物(f/r)4μl(10μm);phantamaxsuper-fidelitydnapolymerase(1u/μl);模板dna4μl(20ng/μl);ddh2o補足到50μl。
pcr擴增程序為95℃預變性3min;然后95℃變性15s,56℃退火45s,72℃延伸30s,35個循環;最后72℃延伸10min,4℃保溫。
pcr反應結束后,將所得pcr產物(786bp)進行1%瓊脂糖凝膠電泳。片段回收送南京擎科生物有限公司測序,每條序列測定3次。
利用clustalx軟件對測序結果進行分析,發現野生大豆bb52和sa101材料中,glyma.07g250300基因編碼區第557位核苷酸為“a”,其余大豆材料的glyma.07g250300基因編碼區均為“g”(如圖2所示)。
實施例3大豆材料的花葉病毒病抗性鑒定
利用南方普遍流行的兩種病毒株系sc3和sc7(南京農業大學大豆研究所提供)分別對表1所列大豆材料分別接種鑒定,鑒定方法與評價指標(抗與敏)參照南京農業大學2000年王修強碩士畢業論文“黃淮和長江中下游地區大豆花葉病毒(smv)株系鑒定、抗源篩選及抗性遺傳的研究”。
表1大豆花葉病毒鑒定材料與結果
結果表明,bb52和sa101對兩種病毒同時表現出抗性,而sa062僅對sc3表現抗性,對sc7為敏感;而其余材料則均對兩種病毒表現出敏感性(表1)。結合實施例2的snp位點結果,bb52和sa101在第557位a堿基,而對兩種病毒表現出抗性,其它材料均為g堿基,而表現出敏感性;對sa062材料而言,基因序列為g堿基,但對一種病毒表現出了抗性,推斷可能大豆中還有其它抗病毒病位點。
由此可以推斷,glyma.07g250300基因中的g/asnp位點可能在大豆的抗病毒病過程中起著重要作用。抗性鑒定實驗由南京農業大學大豆研究所完成。
以上所述,僅用以說明本發明的技術方案而非限制,本領域普通技術人員對本發明的技術方案所做的其它修改或者等同替換,只要不脫離本發明技術方案的精神和范圍,均應涵蓋在本發明的權利要求范圍當中。
sequencelisting
<110>江蘇省農業科學院
<120>一種單核苷酸多態性位點在輔助鑒定大豆花葉病毒抗性中的應用
<130>2
<160>2
<170>patentinversion3.3
<210>1
<211>20
<212>dna
<213>人工合成
<400>1
catgccaaaatcctgaacct20
<210>2
<211>22
<212>dna
<213>人工合成
<400>2
tggaacactatcagccaaagat22
- 還沒有人留言評論。精彩留言會獲得點贊!
- CYP2C9*3基因多態性快速檢測的引物、分子信標、試劑盒及其檢測方法與流程
- 一種單核苷酸多態性位點在輔助鑒定大豆花葉病毒抗性中的應用的制造方法與工藝
- DHFR基因多態性檢測體系及其試劑盒的制造方法與工藝
- ERCC1基因多態性檢測體系及其試劑盒的制造方法與工藝
- 一種判定中國人群rs125055單核苷酸多態性與兒童IBD之間的相關性的方法與制造工藝
- 黃牛ADNCR基因單核苷酸多態性的四引物擴增受阻突變體系PCR檢測方法及其應用與制造工藝
- 使用PNA探針的熔解曲線分析、使用熔解曲線分析用于分析核苷酸多態性的方法和試劑盒與制造工藝
- 人KRAS中單核苷酸多態性的檢測的制造方法與工藝
- 一種InDel位點基因型分型的方法與制造工藝
- 單核苷酸多態性rs145562243在篩查麻風患者中的應用的制造方法與工藝
- 一種用于評估皮膚防御情況的引物和探針及試劑盒的制作方法
- 一種用于評估皮膚防曬情況的引物和探針及試劑盒的制作方法
- 一種用于評估皮膚抗皺情況的引物和探針及試劑盒的制作方法
- 人PLIN1基因rs2289487位點多態性檢測方法及試劑盒的制作方法
- 測定人NEFM基因rs12515位點多態性的方法及試劑盒的制作方法
- 一種人TERF1基因rs3863242位點多態性檢測技術的制作方法
- 測定人CCNH基因rs3093816位點多態性的方法及試劑盒的制作方法
- 測定人NEFL基因rs2979704位點多態性的方法及試劑盒的制作方法
- 測定人PON1基因rs854560位點多態性的方法及試劑盒的制作方法
- 測定人PON1基因rs662位點多態性的方法及試劑盒的制作方法
- CYP2C9*3基因多態性快速檢測的引物、分子信標、試劑盒及其檢測方法與流程
- 一種單核苷酸多態性位點在輔助鑒定大豆花葉病毒抗性中的應用的制造方法與工藝
- DHFR基因多態性檢測體系及其試劑盒的制造方法與工藝
- ERCC1基因多態性檢測體系及其試劑盒的制造方法與工藝
- 一種判定中國人群rs125055單核苷酸多態性與兒童IBD之間的相關性的方法與制造工藝
- 黃牛ADNCR基因單核苷酸多態性的四引物擴增受阻突變體系PCR檢測方法及其應用與制造工藝
- 使用PNA探針的熔解曲線分析、使用熔解曲線分析用于分析核苷酸多態性的方法和試劑盒與制造工藝
- 人KRAS中單核苷酸多態性的檢測的制造方法與工藝
- 一種InDel位點基因型分型的方法與制造工藝
- 單核苷酸多態性rs145562243在篩查麻風患者中的應用的制造方法與工藝
- 用于檢測人類CYP2D6基因多態性的核酸、試劑盒及方法與流程
- 單核苷酸多態性位點rs2523607在篩查皰疹樣皮炎患者中的應用的制作方法與工藝
- 核苷酸多態性檢測方法與流程
- 單核苷酸多態性rs146466242在篩查麻風患者中的應用的制作方法與工藝
- 基于信號譜差異的混合樣本單核苷酸多態性的檢測方法與流程
- 一種檢測牛PCAF基因單核苷酸多態性的RFLP方法及試劑盒與流程
- 秦川牛microRNA?320a?1基因單核苷酸多態性的檢測方法及其應用與流程
- 單核苷酸多態性rs76418789在篩查麻風患者中的應用的制作方法與工藝
- 判定rs4676410和rs2531875單核苷酸多態性與AS之間相關性的方法與流程
- 一種可視化檢測基因序列中單核苷酸多態性位點的反向探針、試劑盒及其檢測方法與流程