一株拮抗內生細菌GH011及應用的制作方法
本發明涉及微生物技術領域,具體的說是一種拮抗內生細菌gh011及應用。
背景技術:
番茄灰霉病已成為我國保護地番茄生產的主要病害,灰霉病是由半知菌亞門真菌灰葡萄孢(botrytiscinerea)侵染所致。目前番茄抗病育種還沒有突破性進展,國內防治番茄灰霉病主要依靠化學藥劑。然而,長期、連續用藥使灰霉病菌產生了抗藥性,導致化學藥劑防治效果逐年下降,并且化學藥劑殘留嚴重污染農產品和環境,危及人畜健康。生物防治已經成為控制病害的重要手段,而植物內生細菌作為植物病害生物防治的潛在資源菌具有獨特的優勢,并且已經顯示出很好的利用前景。
技術實現要素:
本發明目的是提供一株可應用于治療和預防番茄灰霉病的拮抗內生細菌gh011,分類命名為bacillusaxarquiensis,已保藏于中國典型培養物保藏中心,保藏單位簡稱為cctcc,保藏編號cctccno:m2017096。
本發明的另一目的是提供一種治療和預防番茄灰霉病的制劑。
所述拮抗內生細菌gh011可用于抑制桂花葉斑病菌(葡萄座腔菌,b.dothidea)和番茄灰霉病菌(b.cinerea)。
所述治療和預防番茄灰霉病的制劑為拮抗內生細菌gh011培養液。
所述拮抗內生細菌gh011培養液中含有菌落4×108cfu·ml–1。
所述拮抗內生細菌gh011培養液的制備方法為:接種拮抗內生細菌gh011單菌落于lb液體培養基中,28℃的恒溫振蕩培養箱中160r·min-1培養24h,采用無菌水將培養后所得菌液的菌落稀釋至4×108cfu·ml–1。
所述制劑治療和預防番茄灰霉病的方法:采用拮抗內生細菌gh011培養液對番茄植株進行噴霧,進一步說是將拮抗內生細菌gh011培養液噴在番茄葉片的正反面。
有益效果
本發明的拮抗內生細菌gh011在與宿主植物協同進化的過程中,形成了穩定的生態關系,內生細菌可以通過自身產生或誘導宿主產生一些抗菌的物質,或通過與植物病原菌競爭生態位和營養物質對植物病害起到防治作用。該抗內生細菌gh011能有效預防和治療番茄灰霉病,防治效果達到64.9%,且該菌對桂花葉斑病菌(葡萄座腔菌)抑菌率達83.69%。此外,該拮抗內生細菌gh011對桂花炭疽病菌、桂花葉斑病菌(刺球殼菌)和番茄葉霉病菌三種病原菌也有一定程度的拮抗作用,具有較好的應用前景。
生物材料的保藏
一株拮抗內生細菌gh011,分類命名為bacillusaxarquiensisgh011,已保藏于中國典型培養物保藏中心,保藏單位簡稱為cctcc,保藏地址為中國武漢武漢大學,保藏編號為cctccno:m2017096,保藏日期為2017年3月8日。
附圖說明
圖1是本發明拮抗內生細菌gh011對桂花葉斑病菌的抑菌效果圖;
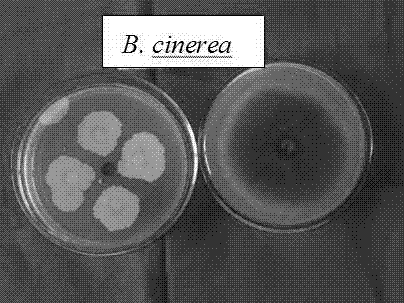
圖2是本發明拮抗內生細菌gh011對番茄灰霉病菌抑菌效果的圖;
圖中:左為對照,右為抑菌效果。
具體實施方式
實施例1
采用預防番茄灰霉病的制劑進行盆栽番茄防治:
步驟一、接種gh011菌株單菌落于200mllb液體培養基中,28℃的恒溫振蕩培養箱中160r·min-1培養24h,將所培養的菌液稀釋至4×108cfu·ml–1,制備出預防番茄灰霉病的制劑;
步驟二、取長至5-6葉,長勢一致的盆栽番茄,用治療和預防番茄灰霉病制劑噴霧至番茄葉片正反面,以清水作為對照,24h后將番茄灰霉病菌的孢子懸浮液1×107個孢子/ml(將少許無菌水注入已長滿灰霉病菌的pda平板內,將瓊脂表面的孢子刮下,倒入無菌容器內,充分振蕩后用滅菌的紗布過濾,于血球計數板中計數)噴霧接種其上。番茄植株培養于人工氣候箱(23℃,r≧90%)中。每個處理3盆,每盆3株,重復3次。接種后7d調查番茄的發病情況,病情分級采取5級標準。0級:無病斑,葉片無癥狀,植株健康;1級:病斑面積占總葉片的10%以下;2級:病斑面積占總葉片的10%-50%;3級:病斑面積占總葉片的50%以上;4級:植株死亡。
病情指數=100×[σ(各級病葉數×病害級別數)]/(調查總葉數×4)防治效果(%)=[(對照病情指數-拮抗菌株處理后病情指數)]/對照病情指數]×100
結果表明:噴霧治療和預防番茄灰霉病制劑的番茄植株比用清水處理的病情指數降低23.1,防治效果為64.9%(見表1)。
表1菌株gh011對番茄灰霉病的防治效果
實施例2
拮抗內生細菌gh011菌株的篩選、分離和純化:
步驟一、培養基的制備:
細菌平板培養用lb培養基(胰蛋白胨10g/l;酵母提取物5g/l;氯化鈉10g/l;瓊脂粉18g/l),液體培養用lb液體培養基(不加瓊脂粉),真菌培養及平板對峙試驗用pda培養基(馬鈴薯200g/l;葡萄糖20g/l;瓊脂粉18g/l)。
步驟二、采集健康桂花植株的葉片3g,在75%的酒精中浸泡30s,然后用1%的次氯酸鈉消毒3min,用無菌蒸餾水沖洗5次后,取最后一次的沖洗液200μl,涂布于lb培養基平板上;消毒后的葉片于無菌的研缽中研磨,加入5ml無菌蒸餾水混勻,取研磨液200μl,涂布于lb培養基平板上。在28℃培養,逐日觀察,如最后一次沖洗液涂布的培養基中無菌落,研磨液涂布的培養基中長出的菌落是內生菌,進行純化培養后,4℃冰箱中保存備用。
步驟三、拮抗活性測定
番茄灰霉病菌、番茄葉霉病菌、桂花葉斑病菌(刺球殼)、桂花炭疽病菌和桂花葉斑病菌(葡萄座腔菌)活化培養,制成8mm的菌餅放置于pda培養基中心,四周40mm處分別放置四片8mm大小含有內生細菌的濾紙片,置于28℃培養箱內培養4~5天,測量抑菌圈半徑并計算抑菌率。
從健康桂花的葉片中共分離出24株內生細菌,其中分離得到的gh011抑菌活性最強,對桂花葉斑病菌(葡萄座腔菌)抑菌率達83.69%,如圖1所示;對番茄灰霉病菌抑菌率達74.83%,如圖2所示;對其他三種病原菌也有一定程度的拮抗作用,結果如表2所示。
表2:菌株gh011發酵液對5種病原真菌的抑菌率
步驟四、強拮抗內生菌株的鑒定
1、形態及生理生化鑒定
方法參考《伯杰氏細菌鑒定手冊》(buchanan&gibbons,1984)和《常見細菌系統鑒定手冊》(東秀珠和蔡妙英,1999)。
1)形態特征
菌株gh011細胞為桿狀、單生或鏈生、周生鞭毛,革蘭氏染色陽性。菌落呈淡黃色、不透明,邊緣不規則。
2)生理生化特征
菌株gh011為好氧菌,不能運動,可以利用蔗糖、果糖、葡萄糖、麥芽糖、甘露醇,但不能利用乳糖,可以水解淀粉和明膠,可分解牛奶產酸,檸檬酸鹽試驗、v-p試驗、硝酸鹽還原試驗和接觸酶試驗呈陽性,苯丙氨酸脫氫酶實驗、甲基紅試驗和氧化酶試驗呈陰性,最適生長ph7.0,最適生長溫度30℃。
2、16srdnapcr擴增、測序及系統發育分析
以細菌基因組dna快速抽提試劑盒(上海生工生物工程公司refb518225-0050)提取的菌株gh011基因組dna為模板。16srdnapcr擴增采用引物:16sl:5'-acggctaccttgttacgacct-3'(其序列如seqidno:3)
和引物16sr:5'-agagtttgatcctggctcag-3'(其序列如seqidno:4)gyra基因pcr擴增采用引物gyra-f:cgatcaggaaatgcgtacgtcctt(其序列如seqidno:5)和引物gyra-r:caaggtaatgctccaggcattgct(其序列如seqidno:6)。反應體系(50μl):10×buffer5.0μl、dntps(2.5mmol·l-1)4.0μl、引物16sr(10pmol·l-1)2.0μl、引物16sl(10pmol·l-1)2.0μl、taq酶(5u·μl-1)0.5μl、模板dna(50ng·μl-1)2μl、ddh2o34.5μl。pcr反應程序:94℃5min,94℃1min,50℃1min,72℃2min,30個循環;72℃10min。將pcr產物經純化后送上海生工生物工程公司測序。根據測序結果,用blast軟件從genbank數據庫中調出相關菌株的16srdna序列和gyra基因序列,分別用clustalwv1.82軟件進行多序列比對,之后用mega4.1軟件進行序列分析,采用鄰接法(neighbor-joining,nj法)進行系統發育分析和同源性比較。
結果顯示:菌株gh011的16srdna(基因序列如seqidno:1所示)和gyra基因序列(如seqidno:2所示)與bacillusaxarquiensis同源性均高達99%。結合其形態特征、生理生化特性、16srdna(基因序列如seqidno:1所示),和gyra基因序列分析,將該菌株鑒定為b.axarquiensisgh011。
sequencelisting
<110>河南科技大學
<120>一株拮抗內生細菌gh011及應用
<130>1
<160>6
<170>patentinversion3.3
<210>1
<211>1515
<212>dna
<213>bacillusaxarquiensis
<400>1
tacggctaccttgttacgacctcaccccaatcatctgtcccaccttcggcggctggctcc60
ataaaggttacctcaccgacttcaggtgttacaaactctcgtggtgtgacgggcggtgtg120
tacaaggcccgggaacgtattcaccgcggcatgctgatccgcgattactagcgattccag180
cttcacgcagtcgagttgcagactgcgatccgaactgagaacagatttgtgggattggct240
taacctcgcggtttcgctgccctttgttctgtccattgtagcacgtgtgtagcccaggtc300
ataaggggcatgatggtttgacgtcatccccaccttcctccggtttgtcaccggcagtca360
ccttagagtgcccaactgaatgctggcaactaagatcaagggttgcgctcgttgcgggac420
ttaacccaacatctcacgacacgagctgacgacaaccatgcaccacctgtcactctgccc480
ccgaaggggacgtcctatctctaggattgtcagaggatgtcaagacctggtaaggttctt540
cgcgttgcttcgaattaaaccacatgctccaccgcttgtgcgggcccccgtcaattcctt600
tgagtttcagtcttgcgaccgtactccccaggcggagtgcttaatgcgttagctgcagca660
ctaaggggcggaaaccccctaacacttagcactcatcgtttacggtgtggactaccaggg720
tatctaatcctgttcgctccccacgctttcgctcctcagcgtcagttacagaccagagag780
tcgccttcgccactggtgttcctccacatctctacgcatttcaccgctacacgtggaatt840
ccactctcctcttctgcactcaagttccccagtttccaatgaccctccccggttgagccg900
ggggctttcacatcagacttaaggaaccgcctgcgagccctttacgcccaataattccgg960
acaacgcttgccacctacgtattaccgcggctgctggcacgtagttagccgtggctttct1020
ggttaggtaccgtcaaggtaccgccctattcgaacggtacttgttcttccctaacaacag1080
agctttacgatccgaaaaccttcatcactcacgcggcgttgctccgtcagactttcgtcc1140
attgcggaagattccctactgctgcctcccgtaggagtctgggccgtgtctcagtcccag1200
tgtggccgatcaccctctcaggtcggctacgcatcgttgccttggtgagccattacctca1260
ccaactagctaatgcgccgcgggtccatctgtaagtggtagccgaagccaccttttatgt1320
ttgaaccatgcggttcaaacaagcatccggtattagccccggtttcccggagttatccca1380
gtcttacaggcaggttacccacgtgttactcacccgtccgccgctaacatcagggagcaa1440
gctcccatctgtccgctcgacttgcatgtattaggcacgccgccagcgttcgtcctgagc1500
caggatcaaactcta1515
<210>2
<211>1032
<212>dna
<213>bacillusaxarquiensis
<400>2
tttagtcaggaaaaagcgtacgtcctttttggattatgcgatgagcgttatcgtatcccg60
ggcactgccggatgtgcgtgacggtttaaaacccggttcacagacggattttatacgcga120
tgaatgatttaggcatgacgagtgacaagccatataaaaaatcggctcgtatcgtcggag180
aagttatcggaaaataccaccctcacggtgattcagcggtatatgaatctatggtcagaa240
tggcgcaggatttcaactaccgttatatgctggtggacggtcacggaaactttggttctg300
tagacggagactcagccgctgcgatgcggtacaccgaagccagaatgtctaagatcgcca360
tggaaattctgcgagacattacaaaagacacgattgattatcaagataactatgatggtt420
cagagagagaacctgttgtcatgccgtcaagattcccgaacctgcttgtaaatggagcag480
ccggtattgcagtcggaatggctacaaatataccgccgcaccagctaggagaaattattg540
acggagtgcttgccgtaagcgagaacaaggacataacaatccaagagctgatggaattca600
ttcctggaccggatttcccgactgccggacaaattttaggcagaagcggaattcgcaagg660
catatgaatccggaagaggttctattacgattcgggcaaaagcagaaattgaagaaactt720
cgtcagggaaagaaagaattttagtaacagaactcccatatcaggtgaataaagcgcgtc780
taattgagaagattgctgatcttgtcagagataagaaaattgaaggaataactgatttgc840
gtgacgaatctgaccggaacggtatgcggattgtcattgaaatcagacgagatgccaatg900
cgcatgttatcctgaacaatctttacaaacaaacggccctgcaaacttccttcgggatta960
acatgctggcgctggttgacggtgagccgaaagtattaaatttaaagcaatgcctgagcc1020
atttaccttgaa1032
<210>3
<211>21
<212>dna
<213>人工序列
<400>3
acggctaccttgttacgacct21
<210>4
<211>20
<212>dna
<213>人工序列
<400>4
agagtttgatcctggctcag20
<210>5
<211>24
<212>dna
<213>人工序列
<400>5
cgatcaggaaatgcgtacgtcctt24
<210>6
<211>24
<212>dna
<213>人工序列
<400>6
caaggtaatgctccaggcattgct24
- 還沒有人留言評論。精彩留言會獲得點贊!