一種用于對不同銀杏進行基因分型的SNP引物及檢測方法與流程
技術領域:
本發明屬于生物技術領域,具體涉及一種用于對不同銀杏進行基因分型的snp引物及檢測方法。
背景技術:
銀杏(ginkgobilobal.)作為裸子植物銀杏綱(ginkgopsida)現存唯一種,典型的中生代孑遺物種,素有“活化石”之美譽。銀杏為落葉大喬木,樹干高大,雌雄異株。樹皮幼時淺綠色,老樹呈灰褐色,深縱裂,粗糙;分枝繁茂,分長短枝。種子核果狀,有臭味;中種皮骨質,白色,具2~3棱;內種皮膜質淡褐色,子葉2片;花期4~5月,9~10月種子成熟。銀杏集材用、果用、葉用、園林綠化、觀賞于一體,是一種全能的樹種。銀杏葉中含有大量的銀杏黃酮類和銀杏內酯類物質,對治療神經病,骨髓病和腦病有獨特的效用。銀杏葉的非凡藥用價值,已經使銀杏葉制劑形成了一個巨大的產業。
單核苷酸多態性(singlenucleotidepolymorphisms,snp)是指單個核苷酸的變異引起的基因組水平上dna序列的多態性,其形式包括單堿基的插入、缺失、轉換和顛換。在群體中,其等位基因頻率大于1%,但有時也把cdna中頻率小于1%的單堿基變異稱之為snp。snp標記是繼rflp和ssr之后發展起來的第三代分子標記,并廣泛應用于植物遺傳育種中,具有遺傳穩定性高;密度高、分布廣;二態性和等位基因性;富有代表性;易于實現高通量、自動化檢測的特點。因此,snp被認為是應用前景最好的遺傳標記之一。
隨著測序技術的發展,測序質量的提高和成本的降低,挖掘snp位點并借此進行相關研究的例子越來越多。但該技術在植物中的應用多集中于農作物,在林木類方面的研究相對較少,且在銀杏這一物種上的研究更是未見報道。隨著時代的發展,野生資源的保護至關重要,對于孑遺樹種古銀杏保護不容忽視。因此,現急需一種快速、方便、可靠的銀杏古樹名木資源分子鑒定技術。利用dna分子標記方法繪制dna指紋圖譜,能從本質上反映生物個體間的差異。常規的dna指紋圖譜是采用第二代分子標記簡單序列重復(ssr)進行的,但是第三代分子標記snp,無須基于凝膠電泳進行指紋圖譜的檢測,減少誤差,省時,且高通量,準確性高。
技術實現要素:
發明目的:針對現有技術中存在的不足,本發明的目的是提供一種用于對不同銀杏進行基因分型的snp引物,為84株古樹名木建立指紋圖譜。本發明的另一目的是提供上述snp引物的應用。
技術方案:為了實現上述發明目的,本發明采用的技術方案為:
一種用于對不同古銀杏進行基因分型的snp引物,由22對引物組成,各引物的核苷酸序列如下表所示:
應用所述的snp引物構建銀杏古樹名木的指紋圖譜,獲得84個銀杏古樹名木對應的指紋圖譜,如下表所示:
所述的應用snp引物構建銀杏古樹名木的指紋圖譜,所使用的引物為11,名稱分別:snp1,snp4,snp6,snp8,snp10,snp14,snp16,snp17,snp19,snp20,snp22。
所述snp引物在不同古銀杏基因組dna的snp位點基因分型檢測中的應用。
所述的應用,包括以下步驟:
1)待測銀杏樣品dna的提取;
2)使用相同反應體系及程序對銀杏樣本基因組dna進行pcr擴增,得到待測銀杏的pcr產物;
3)對pcr擴增產物進行高分辨率溶解曲線分析確定snp位點的基因型;
4)sanger測序再次驗證snp基因型。
步驟2)中,pcr反應體系10μl:25ng模板dna;1xforget-me-nottmqpcrmastermix;0.1μmol/lsnp引物;剩余ddh2o補齊。
步驟2)中,pcr反應程序:預變性95℃2min;95℃變性5s,45個循環;60℃復性30s。
步驟3)中,hrm的反應程序:95℃10s,60℃1min,95℃15s,60℃15s。
本發明通過實驗證明:本發明的引物共22對,其中11對snp引物組合可以用于構建銀杏古樹名木的指紋圖譜。本發明利用銀杏轉錄組數據開發的snp引物能夠對銀杏進行基因分型,并且本發明操作方法簡單,通量高,成本低。此外,本發明開發的11對snp引物構建了不同銀杏古樹名木的指紋圖譜,驗證的22個snp位點還可以用來分子標記輔助育種,遺傳多樣性研究,基于snp的關聯分析,以及用于種質資源鑒定與分類等。
有益效果:與現有技術相比,本發明具有以下的技術優勢:
1)結果高度準確、可靠:利用本發明進行檢測與鑒定,檢測結果100%準確,檢測結果提供了高度的可靠性,為銀杏古樹名木提供指紋圖譜,對野生資源的保護意義重大。
2)操作快速簡便:進行不同個體間的樣本檢測,一般整個試驗過程只需幾小時即可完成,與傳統的測序技術相比,省時省力,可以實現高通量。
3)無需基于凝膠電泳進行,減少試劑和耗材的使用,更佳環保,同時節省人力和減少人工誤差。
4)檢測結果靈敏度高:對待檢測樣品僅需提供模板dna25ng即可準確進行鑒定。
附圖說明
圖1是從銀杏轉錄組測序數據中鑒定出的snp及其變異類型和數目結果圖;
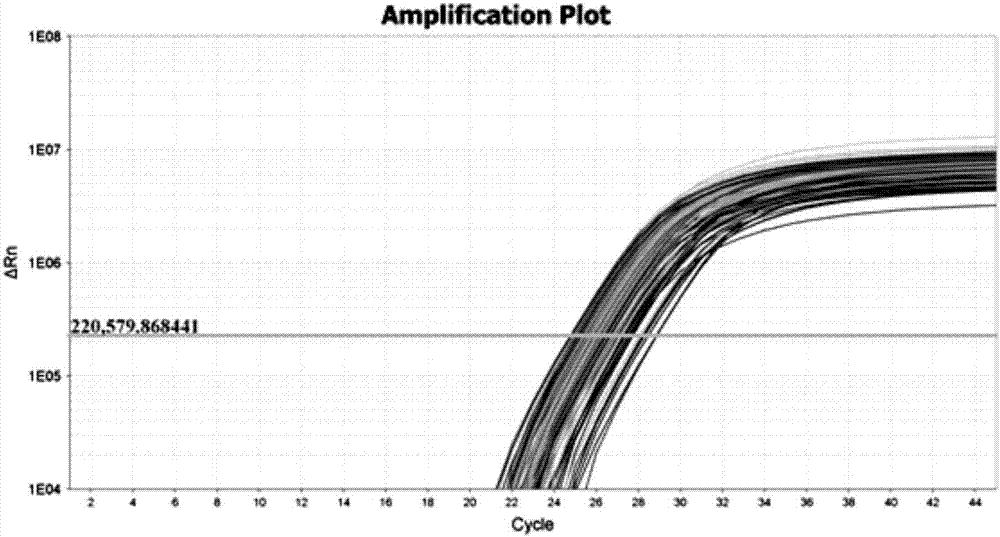
圖2是以引物snp18為例,在不同古銀杏個體表現出的擴增曲線圖;
圖3是以引物snp18為例,利用高分辨率溶解曲線分析后,不同古銀杏個體表現出不同的溶解曲線峰圖;
圖4是以引物snp18為例,根據不同古銀杏個體表現出的差異溶解曲線峰,對其進行基因分型結果的溶解曲線,紅色為cc,藍色為ct,綠色為tt。
具體實施方式
下面結合具體實施例對本發明做進一步的說明。
實施例1
1、初步篩選對銀杏進行基因分型的snp引物
1)轉錄組數據的獲得
根據制造商的說明書使用rneasyplantminikit(qiagen,hilde,germany)進行銀杏總rna提取。提取后,在1.0%的瓊脂糖凝膠上檢測rna的降解和污染。使用
每個樣品的總量為3μgrna用作rna樣品制備的輸入材料。按照制造商的說明書,使用
fastq格式的原始數據(rawreads)首先通過內部perl腳本進行處理。在此步驟中,通過刪除包含適配器的讀取,包含poly-n的讀取以及從原始數據讀取的低質量來獲取干凈的數據(cleanreads)。同時,計算了cleandata的q20,q30,gc含量和序列重復水平。所有的下游分析都是以高質量的cleandata為基礎。然后,將所有庫/樣本中的左文件(read1files)合并到一個大的left.fq文件中,將正確的文件(read2files)合并到一個大的right.fq文件中。基于left.fq和right.fq使用trinity(grabherretal.,2011)完成轉錄組組裝,默認情況下min_kmer_cov設置為2,其他所有參數設置為默認值。選擇具有最長長度的每個基因的轉錄本作為unigenes。
2)snp的識別
本實施例通過使用picard-toolsv1.41和samtoolsv0.1.18來排序,刪除重復讀取,對齊合并獲得每個樣本的bam結果。采用gatk2v3.2軟件用于執行snpcalling(mckennaetal,2010)。原始的vcf文件使用gatk標準方法過濾(參數為clusterwindowsize:10;mq0>=4and(mq0/(1.0*dp))>0.1;qual<10;qual<30.0orqd<5.0orhrun>5),最后僅保留距離大于5的snp。通過該方法鑒定出65535個snp,snp變異的形式有多種,但類型主要有轉換和顛換,轉換是顛換的2倍左右,如圖1所示。
3)snp引物的設計
根據轉錄組unigenes序列,挑選60個snp位點,利用oligo6.0軟件設計60對snp引物。設計的方法如下:首先確定snp位點所在序列的位置,然后在snp位點的上下游分別設計正向引物和反向引物,引物設計的參數為引物長度18-23bp;tm60℃左右,盡可能保證上下游引物的tm值一致,一般不超過2℃;gc含量在40-60%(45-55%最佳);pcr產物大小為100-300bp;其它還應注意引物3'端不應出現超過3個的連續g或c;引物不能形成發夾結構,即不能有4bp以上的回文序列;引物自身、引物之間盡量不能有連續4個以上的堿基互補,尤其避免3'端的互補。
4)snp引物的有效性驗證
提取銀杏基因組dna,以銀杏基因組dna為模板,采用待檢測snp引物進行pcr擴增,獲得pcr擴增產物;反應結束后,取3μl的pcr產物加入4μlloadingbuffer,再加3μlddh2o,利用8%聚丙烯酰胺凝膠進行電泳,然后采用銀染方法對pcr擴增產物進行檢測。能檢測到目的條帶且無其它雜帶和引物二聚體的,該待檢測引物即為有效引物。
10μl的pcr反應體系中包括:10×buffer;0.2mmol/ldntps;2.5mmol/lmg2+;0.2μmol/lsnp引物;1utaqdna聚合酶和40ngdna模版;剩余ddh2o補齊。
pcr的反應程序為:預變性94℃5min;30循環的94℃30s,最佳tm55℃30s,72℃30s;延伸72℃3min,最終4℃保存。
結果表明:挑選的60對snp引物中,有45對引物能夠擴增出條帶,說明引物設計的方法較好,但其中的18對引物擴增出的條帶不單一,可能存在多個snp位點,暫不考慮進行hrm分析,另外27對引物擴增條帶單一且產物大小與預期一致,暫為有效snp引物,用于hrm分析。1對引物對應1個目標snp位點,1對引物的擴增產物為含有對應snp位點的dna片段。
2、再次篩選對銀杏進行基因分型的snp引物
1)pcr擴增及高分辨率溶解曲線(hrm)分析進行基因分型
以90個銀杏個體(其中6個銀杏個體為轉錄組測序的銀杏;84棵銀杏古樹名木,分別來自3個群體,貴州盤縣28株,浙江天目山28株和湖北安陸28株)的基因組dna為模板,分別采用上述27對snp有效性引物進行pcr擴增,得到每個個體的27個引物對應的pcr擴增產物,每個引物對的pcr擴增產物為含有某個snp位點的dna片段。
pcr反應體系10μl:25ng模板dna;1×forget-me-nottmqpcrmastermix;0.1μmol/lsnp引物;剩余ddh2o補齊。采用viia7中的highresolutionmelt進行pcr及基因分型。第一步,10μl的pcr反應程序:預變性95℃2min;45個循環的95℃變性5s,60℃復性30s;第二步,hrm的反應程序:95℃10s,60℃1min,95℃15s,60℃15s。
結果表明:有22對引物能夠進行基因分型,其余5對引物(snp23,snp24,snp25,snp26,snp27)因有雜峰或基因分型時可信度不高,所以被淘汰。采用applied
圖2為以snp18為例,在不同銀杏個體表現出的擴增曲線;圖3為以snp18為例,利用高分辨率溶解曲線分析后,不同銀杏個體表現出不同的溶解曲線峰;圖4為以snp18為例,根據不同銀杏個體表現出的差異溶解曲線峰,對其進行基因分型結果的溶解曲線,紅色為cc,藍色為ct,綠色為tt。22對snp引物對90個銀杏個體的基因分型結果如下表所示:
其中,11對snp引物組合(snp1,snp4,snp6,snp8,snp10,snp14,snp16,snp17,snp19,snp20,snp22)構建銀杏古樹名木指紋代碼,獲得84個銀杏古樹名木對應的指紋圖譜,序號1-28為貴州盤縣這個群體的28株古銀杏,序號29-56為浙江天目山這個群體的28株古銀杏,序號57-84為湖北安陸這個群體的28株古銀杏,如下表所示:
2)高分辨率溶解曲線分析結果的驗證
通過對pcr產物測序對高分辨率溶解曲線分析得到的基因分型的結果進行驗證。具體步驟如下:挑選snp的不同分型的pcr產物10μl,進行sanger測序,根據測序峰圖和等位基因出現的頻率確定基因型,并與hrm技術的snp基因分型結果進行比較,以驗證引物基因分型的準確性。
經過sanger測序驗證本發明的引物通過hmr分析對銀杏進行基因分型的結果100%正確,結果可靠。
- 還沒有人留言評論。精彩留言會獲得點贊!