源于稻瘟病菌的致病性基因MoSNT2及其用途的制作方法
本發明屬于微生物基因工程領域,具體涉及一個源于稻瘟病菌的致病性基因mosnt2;還涉及該基因的用途。
背景技術:
稻瘟病(又名稻熱病)是水稻重要病害之一,可引起大幅度減產,嚴重時減產40%~50%,甚至顆粒無收。稻瘟病在世界各稻區均有發生,其中以葉部、節部發生為多,尤其穗頸瘟或節瘟發生早而重,可造成白穗以致絕產。引起稻瘟病的病原體為稻瘟病菌,屬于半活體寄生性真菌(magnaportheoryzae),它具有生理小種多、繁殖變異快、流行性強等特點,對水稻生產威脅巨大。為了保障水稻生產安全,迫切需要尋找防治稻瘟病的有效途徑。
稻瘟病菌對寄主水稻的侵染過程主要分為以下幾個階段:分生孢子粘附寄主、孢子萌發出芽管并分化產生侵染器官附著胞、附著胞產生侵染釘穿透植物表皮、侵染菌絲在植物組織中增殖并分泌效應蛋白抑制寄主免疫力、菌絲殺傷植物組織形成壞死斑、形成分生孢子梗并釋放分生孢子(wilson等,naturereviewsmicrobiology,2009,7(3):185-195)。許多基因在稻瘟病侵染寄主水稻過程中發揮重要功能,調控稻瘟病菌侵染器官發育及侵染菌絲增殖,從而決定稻瘟病的致病力與侵染性。闡明這些致病性相關基因的分子功能,將有助于開發及利用新的稻瘟病防治靶點。
dean等(dean等,2005,434(7036):980-986)完成了稻瘟病菌70-15菌株的基因組序列測定。韓國首爾大學構建了稻瘟病菌突變體庫,并報道了202個致病性基因(jeon等,naturegenetics,2007(39):561-565)。我國科研工作者對稻瘟病菌流行性菌株進行了基因組測序及解讀,鑒定出許多與稻瘟病菌致病性相關的基因,這些基因所編碼的轉錄因子、分泌性效應蛋白、胞泌調控因子等,在稻瘟病菌侵染寄主水稻過程中起著至關重要的作用(dong等,2015,plospathogens11(4):e1004801)。國內外學者已鑒定許多致病相關基因(yan等,currentopinioninmicrobiology,2016,34:147–153)。這些致病性基因所參與的代謝與信號通路主要包括:黑色素合成、脂肪酸β-氧化、海藻糖代謝、乙醛酸循環、細胞自噬、細胞分泌、camp/pka、g蛋白、mapk信號途徑等。但是,針對這些代謝與信號途徑,目前可利用的真菌特異防治靶點還十分有限。
目前,防治植物病原真菌的方法主要是化學防治。鑒定并解析植物病原真菌的致病性基因,尤其是與入侵寄主過程相關的功能基因,可以為設計、篩選抗真菌藥物提供重要的靶標位點。傳統的抗真菌藥物的靶點主要包括細胞壁合成相關的重要酶類、細胞膜中甾醇與鞘磷脂等關鍵成分的合成酶類、真菌蛋白質與核酸的合成機器等(余新旭,2013,福建農林大學碩士學位論文;劉曉環,藥物分析雜志,2015,35(2):193-202)。三環唑廣泛應用于防治稻瘟病菌,它可破壞侵染器官附著胞的穿透功能,其作用靶標是細胞壁黑色素合成相關的三羥萘還原酶。稻瘟病菌易變異、繁殖快和適應性強,長期使用單一抗真菌藥物,易使病原菌累積抗藥性,導致防效下降。因此,采用分子生物學技術,克隆及鑒定新的在稻瘟病菌侵染過程中發揮重要功能的致病基因,可為設計與篩選新型抗真菌藥劑提供重要的藥物靶點,這對稻瘟病的綜合防治具有十分重要的理論意義和應用價值。
技術實現要素:
針對目前存在的稻瘟病危害嚴重,缺少有效防治方法的問題,本發明目的在于提供一種源于稻瘟病菌的致病性蛋白。
本發明另一目的在于提供編碼上述致病性蛋白的基因。該基因對稻瘟病菌的菌絲營養生長、分生孢子產生和萌發、附著胞形成及致病性等都有重要作用。
本發明第三目的在于提供含有上述基因的表達載體。
本發明第四目的在于提供上述致病性蛋白的用途。
本發明第五目的在于提供上述基因的用途。
本發明第六目的在于提供上述表達載體的用途。
為實現上述目的,本發明的技術方案如下:
本發明提供了一種源于稻瘟病菌的致病性蛋白,命名為mosnt2,所述蛋白的氨基酸序列如下:
(1)、具有seqidno:1所示的氨基酸序列;
(2)、將seqidno:1所示的氨基酸序列經過一個或幾個氨基酸殘基的添加、替換或缺失所形成的、并具有稻瘟病致病性功能的氨基酸序列。
本發明還提供了編碼上述致病性蛋白的基因,命名為mosnt2,該基因具有如下(a)、(b)或(c)的核苷酸序列:
(a)具有seqidno:2所示的核苷酸序列;
(b)在嚴謹條件下與seqidno:2所示的核苷酸序列互補的核苷酸序列;
(c)將seqidno:2所示的核苷酸序列經過一個或幾個堿基的添加、缺失或替換所形成的、且編碼具有稻瘟病致病性蛋白功能的核苷酸序列。
本發明還提供了含有上述基因的表達載體。
本發明還提供了上述致病性蛋白作為設計或/和篩選抗真菌藥物靶標的應用。
本發明還提供了上述基因作為設計或/和篩選抗真菌藥物靶標的應用。
上述應用中所述的真菌是指稻瘟病菌(magnaportheoryzae)等。
本發明還提供了上述表達載體在篩選抗真菌藥物上的應用。
上述應用中所述的真菌是指稻瘟病菌(magnaportheoryzae)等。
本發明具有的優點和有益效果:本發明為防治稻瘟病菌提供了一個重要的靶標基因或靶標蛋白。本發明稻瘟病致病性蛋白對稻瘟病菌的菌絲營養生長、分生孢子產生和萌發、附著胞形成等各個生長發育階段,以及致病性有重要影響,以該基因或蛋白作為靶標研制的藥物,可破壞稻溫病菌的基因表達或蛋白功能,可以有效地控制稻瘟病菌的致病性和流行性。
附圖說明
圖1.不同發育階段mosnt2基因表達水平柱形圖;其中1為液生營養菌絲;2為分生孢子梗;3為分生孢子;4為發育4小時的附著孢;5為發育8小時附著孢;6為發育12小時附著孢;7為發育16小時附著孢;8為發育20小時附著孢;9為發育24小時附著孢;10為生長于水稻葉鞘的侵染菌絲;11為侵染水稻葉片的侵染菌絲。
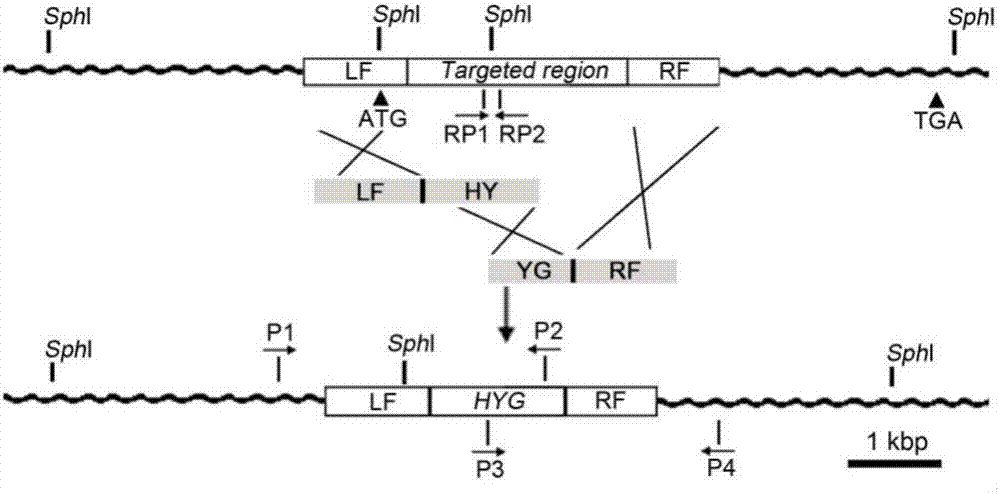
圖2.mosnt2基因敲除突變體的構建過程示意圖。
圖3.以p1和p2為引物的mosnt2敲除突變體的pcr檢測電泳圖譜;其中1為dna分子量標準;2為△mosnt2.1;3為△mosnt2.2;4為△mosnt2.3;5為△mosnt2.4;6為△mosnt2.5;7為野生型guy11。
圖4.以p3和p4為引物的mosnt2敲除突變體的pcr檢測電泳圖譜;其中1為dna分子量標準;2為△mosnt2.1;3為△mosnt2.2;4為△mosnt2.3;5為△mosnt2.4;6為△mosnt2.5;7為野生型guy11。
圖5.驗證mosnt2敲除突變體的southern雜交圖譜;其中1為guy11;2為△mosnt2.1;3為△mosnt2.2;4為△mosnt2.3;5為△mosnt2.4;6為△mosnt2.5。
圖6.mosnt2敲除突變體等在cm平板培養基上的菌落及氣生菌絲形態照片;其中1為guy11;2為△mosnt2.1;3為△mosnt2.2;4為compl.。
圖7.mosnt2敲除突變體等氣生菌絲生長及產孢顯微照片;其中1為guy11;2為△mosnt2.1;3為△mosnt2.2;4為compl.。
圖8.mosnt2敲除突變體等產孢量柱形圖;其中1為guy11;2為△mosnt2.1;3為△mosnt2.2;4為compl.。
圖9.mosnt2敲除突變體等孢子形態顯微照片;其中1為guy11;2為△mosnt2.1;3為△mosnt2.2;4為compl.。
圖10.mosnt2敲除突變體等在細胞壁抑制劑cfw脅迫下的菌落形態照片;其中1為guy11;2為△mosnt2.1;3為△mosnt2.2;4為compl.。
圖11.mosnt2敲除突變體等在h2o2氧化脅迫下的菌落形態照片;其中1為guy11;2為△mosnt2.1;3為△mosnt2.2;4為compl.。
圖12.mosnt2敲除突變體等侵染器官附著孢的發育的顯微照片;其中1為guy11;2為△mosnt2.1;3為△mosnt2.2;4為compl.。
圖13.mosnt2敲除突變體等對寄主水稻葉片的致病力照片;其中1為guy11;2為△mosnt2.1;3為△mosnt2.2;4為compl.;5為接種無菌瓊脂塊的空白對照。
圖14.mosnt2敲除突變體等對寄主水稻根部的致病力照片;其中1為guy11;2為△mosnt2.1;3為△mosnt2.2;4為compl.;5為接種無菌瓊脂塊的空白對照。
具體實施方式
下面通過實施例對本發明作進一步解釋和說明,但是對本發明的保護范圍不構成任何限制。下述實施例中所用方法如無特別說明,均為常規方法。
實施例1:mosnt2基因的分離和克隆
(1)snt2蛋白在酵母的氧化脅迫應答過程中發揮重要作用(baker等,molecularandcellularbiology,2013,(33)19:3735–3748),但該蛋白在稻瘟病菌中的功能未知。酵母和稻瘟病菌的蛋白序列之間存在一定相似性,因此首先從酵母的基因組數據庫(http://www.yeastgenome.org/)網站搜索獲得酵母snt2蛋白序列,然后利用blastp在稻瘟病菌基因組數據庫(http://www.broadinstitute.org/annotation/genome/magnaporthe_grisea/multihome.html)中進行同源序列搜索,從中獲得一個稻瘟病菌預測蛋白mgg_04421,命名為mosnt2,該蛋白的氨基酸序列見seqidno:1。
(2)mosnt2基因的克隆:為克隆mosnt2的cdna序列,首先使用rneasyplantminikit試劑盒(qiagen公司),從稻瘟病菌野生型菌株guy11中提取總rna,然后使用superscriptiiireversetranscriptase試劑盒(invitrogen公司),將總rna逆轉錄生成第一鏈cdna,再以snt2-cdna-for和snt2-cdna-rev為引物,使用phusion高保真酶(thermofisher公司)從第一鏈cdna中克隆mosnt2基因編碼序列,共5382bp,最后將其連接到克隆載體peasy-blunt(全式金公司)上進行測序分析,獲得mosnt2基因的克隆;mosnt2基因序列見seqidno:2。
所述pcr的反應體系(50μl):guy11菌株cdna0.5μl(50ng),phusiondna聚合酶0.5μl(2u/μl),5×phusionhfbuffer10μl,dntp(25mmol/l,each)1μl,上游引物(50μmol/l)0.5μl,下游引物(50μmol/l)0.5μl,ddh2o37μl。pcr反應程序為:98℃30s;98℃10s,60℃30s,72℃3.5min,35個循環;72℃10min,4℃保溫。所述的引物snt2-cdna-for(上游引物)和snt2-cdna-rev(下游引物)為:
snt2-cdna-for:5’-atggctcaaaagggttcaggtg-3’(seqidno:3);
snt2-cdna-rev:5’-agacagtagatttcgcaacgaag-3’(seqidno:4)。
實施例2:mosnt2基因在不同發育階段表達水平的定量rt-pcr檢測
(一)有關培養基和溶液的配制:
(1)cm液體培養基:其組成成分及其比例為:d-glucose10g,peptone2g,yeastextract1g,casaminoacids1g,20×nitratesalts*50ml,vitaminsolution#1ml,traceelements&1ml,加ddh2o至1000ml,并用1mol/l的naoh溶液調節ph值至6.5。
(2)cm瓊脂平板:其組成成分及其比例為:d-glucose10g,peptone2g,yeastextract1g,casaminoacids1g,20×nitratesalts*50ml,vitaminsolution#1ml,traceelements&1ml,瓊脂粉15g,加ddh2o至1000ml,并用1mol/l的naoh溶液調節ph值至6.5;121℃濕熱滅菌20min。
(3)*20×nitratesalts(1000ml)組成成分及其比例為:nano3120g,kcl10.4g,mgso4·7h2o10.4g,kh2po430.4g,加ddh2o至1000ml,121℃濕熱滅菌20min。
(4)#vitaminsolution(1000ml)組成成分及其比例為:biotin0.1g,pyridoxin0.1g,thiamine0.1g,riboflavin0.1g,paba(p-aminobenzoicacid)0.1g,nicotinicacid0.1g,加ddh2o至1000ml,過濾除菌,4℃黑暗儲存。
(5)&traceelements(100ml)組成成分及其比例為:znso4·7h2o2.2g,h3bo31.1g,mncl2·4h2o0.5g,feso4·7h2o0.5g,cocl2·6h2o0.17g,cuso4·5h2o0.16g,na2moo4·2h2o0.15g,加ddh2o至100ml,過濾除菌,4℃黑暗儲存。
(二)試驗方法:
以稻瘟病菌野生型菌株guy11為材料,收集cm液體培養基中營養菌絲、cm瓊脂平板上產孢階段的分生孢子梗和分生孢子、附著胞發育階段不同時間點的菌體、以及定殖水稻葉鞘或葉片的侵染菌絲;使用rneasyplantminikit試劑盒(qiagen公司)從收集的菌體中分別提取總rna,然后用primescriptrtreagentkitwithgdnaeraser(takara公司)反轉錄產生cdna,再以rp1和rp2為引物,用sybrpremixextaq(takara公司)進行定量rt-pcr,并利用稻瘟病菌基因β-tublin的表達水平進行均一化校正。所述的引物rp1和rp2為:
rp1:5’-caggattatggagttcttg-3’(seqidno:5);
rp2:5’-ctagtatcgttcaccttac-3’(seqidno:6)。
結果(見圖1)mosnt2基因在產孢階段分生孢子梗與分生孢子中的表達量最高;附著胞發育過程中,mosnt2基因表達量在孢子接種12小時后達到最高值,侵染菌絲與營養菌絲中的表達量比較接近。上述結果說明mosnt2基因在稻瘟病菌的各個生長發育階段均有表達。
實施例3:稻瘟病菌mosnt2基因敲除突變體的構建
按照如下方法進行:
采用split-marker基因敲除方法(參考文獻:kershaw等,pnatlacadsciusa,2009,106(37):15967-15972)構建基因mosnt2的敲除突變體(見圖2)。基因mosnt2約2.5kbp的編碼序列選為打靶位點,基因敲除過程中,同源重組事件將該打靶位點序列置換為潮霉素篩選標記基因hyg,從而形成基因敲除突變體。具體構建步驟如下:
(1)以稻瘟病菌野生型guy11基因組dna為模板,分別以snt2-lf-for和snt2-lf-rev、或snt2-rf-for/snt2-rf-rev為引物,用dna聚合酶kod-plus(康為世紀生物科技有限公司)進行pcr擴增,分別擴增得到mosnt2基因的左翼lf(seqidno:7)或右翼rf片段(seqidno:8)。其中pcr反應體系(50μl)為:guy11基因組dna0.5μl(100ng),taq聚合酶0.5μl(5u/μl),10×buffer(+mgcl2,25mmol/l)5μl,dntp(25mmol/l,each)0.5μl,上游引物(50μmol/l)0.5μl,下游引物(50μmol/l)0.5μl,ddh2o42.5μl。pcr反應程序為:94℃5min;94℃30s,58℃30s,72℃1min,35個循環;72℃10min,4℃保溫。所述的引物snt2-lf-for和snt2-lf-rev為:
snt2-lf-for:5’-aatccctgcgggctactt-3’(seqidno:9);
snt2-lf-rev:5’-ccttcaatatcatcttctgtcgacctcacaaaggcaccgctg-3(seqidno:10)。
所述的引物snt2-rf-for和snt2-rf-rev為:
snt2-rf-for:5’-cccagcactcgtccgagggcaaaggaataggcatctccaagttcggctca-3’(seqidno:11);
snt2-rf-rev:5’-acaaccgcaaccctggga-3’(seqidno:12)。
(2)分別以hyg-for和hy-split、或yg-split和hyg-rev為引物從質粒模板pcb1004中分別擴增潮霉素篩選標記基因hyg的片段hy(見seqidno:17)或片段yg(見seqidno:18)。所述的引物hyg-for和hy-split為:
hyg-for:5’-gtcgacagaagatgat-3’(seqidno:13);
hy-split:5’-tacttcgagcggaggcatcc-3’(seqidno:14)。
所述的引物yg-split和hyg-rev為:
yg-split:5’-cgttgcaagacctgcctgaa-3’(seqidno:15);
hyg-rev:5’-ctattcctttgccctcggacg-3’(seqidno:16)。
上述pcr的反應體系(50μl)為:pcb1004質粒模板0.5μl(10ng),taq聚合酶0.5μl(5u/μl),10×buffer(+mgcl2,25mmol/l)5μl,dntp(25mmol/l,each)0.5μl,上游引物(50μmol/l)0.5μl,下游引物(50μmol/l)0.5μl,ddh2o42.5μl。pcr的反應程序為:94℃5min;94℃30s,58℃30s,72℃1min30s,35個循環;72℃10min,4℃保溫。
(3)利用瓊脂糖凝膠電泳回收上面擴增得到的四個片段lf、rf、hy和yg;通過融合pcr,使用引物對snt2-lf-for和hy-split將片段lf和hy連接形成融合片段lf-hy(見seqidno:19);同時使用引物對yg-split和snt2-rf-rev將片段yg和rf連接形成融合片段yg-rf(見seqidno:20)。
上述獲得融合片段lf-hy或yg-rf的pcr的反應體系(50μl)為:上游片段1μl(約50ng),下游片段1μl(約50ng),taq聚合酶0.5μl(5u/μl),10×buffer(+mgcl2,25mmol/l)5μl,dntp(25mmol/l,each)0.5μl,上游引物(50μmol/l)0.5μl,下游引物(50μmol/l)0.5μl,ddh2o41μl。pcr反應程序為:94℃5min;94℃30s,58℃30s,72℃1min30s,35個循環;72℃10min,4℃保溫。
(4)mosnt2敲除突變體的獲得:瓊脂糖凝膠電泳回收dna片段lf-hy和yg-rf,同時參照tablot等(tablot等,theplantcell,1993,5:1575-1590)所述方法制備稻瘟病菌野生型菌株guy11的原生質體,將lf-hy和yg-rf片段共轉至原生質體中,采用含潮霉素的cm瓊脂平板篩選真菌轉化子。結果獲得mosnt2基因敲除的轉化子(或稱為mosnt2敲除突變體)。在基因置換過程(如圖2所示)中,大部分外源dna插入到稻瘟病菌的基因組中,由于融合片段的lf和rf序列與基因組上mosnt2的位點序列存在同源性,因此會發生同源置換,獲得mosnt2敲除突變體。
實施例4稻瘟病菌mosnt2基因敲除突變體的鑒定
按照如下方法進行:
(1)提取實施例3中所得轉化子的基因組dna,分別以p1和p2、或p3和p4為引物進行pcr擴增,其中隨機插入的片段不能產生擴增條帶,只有在mosnt2基因位點發生了同源置換的轉化子才能產生擴增條帶。其中轉化子檢測的pcr反應體系(25μl)為:轉化子基因組dna0.5μl(50ng),taq聚合酶0.3μl(5u/μl),10×buffer(+mgcl2,25mmol/l)2.5μl,dntp(25mmol/l,each)0.2μl,上游引物0.2μl,下游引物0.2μl,ddh2o21.1μl。pcr反應程序為:94℃5min;94℃30s,58℃30s,72℃2.5min,35個循環;72℃10min,4℃保溫。其中所述的引物p1和p2為:
p1:5’-ggtaggagaaaggctgatgg-3’(seqidno:21);
p2:5’-gcttctgcgggcgatttgtgta-3’(seqidno:22)。
所述的引物p3和p4為:
p3:5’-cagcgagagcctgacctattgc-3’(seqidno:23);
p4:5’-cttcctgacgcttccgataa-3’(seqidno:24)。
結果以p1和p2為引物所得的擴增產物大小為2.8kbp(見圖3),以p3和p4為引物所得的擴增產物大小為2.2kbp(見圖4)。5個轉化子是經同源置換而產生的敲除突變體,分別命名為△mosnt2.1、△mosnt2.2、△mosnt2.3、△mosnt2.4和△mosnt2.5。
(2)southern雜交鑒定:提取野生型菌株guy11與5個敲除突變體的基因組dna,然后使用限制性內切酶sphi進行酶切,酶切產物經瓊脂糖凝膠電泳之后進行轉膜。southern雜交試驗使用地高辛dna標記與檢測試劑盒(roche公司)。雜交探針為lf片段,用于合成探針的pcr反應體系與前述操作方法相同。
結果(見圖5)野生型guy11菌株在1.2kbp和4.1kbp處有雜交信號,而敲除突變體δmosnt2由于同源置換,使得編碼框內的一個sphi酶切位點消失(如圖2所示),從而導致在4.1kbp和5.5kbp處有雜交信號,該southern鑒定結果證實△mosnt2.1、△mosnt2.2、△mosnt2.3、△mosnt2.4和△mosnt2.5均是正確的mosnt2敲除突變體。
實施例5mosnt2基因互補載體及回復菌株的構建
為了驗證突變體的表型是因為基因mosnt2的缺失而引起的,按照oldenburg等(oldenburg等,1997,nucleicacidsresearch,25:451-452)所述的酵母細胞內重組的方法,構建互補載體pneb-mosnt2-gfp-bar,該載體中mosnt2蛋白的c末端標記綠色熒光蛋白gfp,以利于分析mosnt2的蛋白亞細胞定位,該載體構建方法如下:
(1)以guy11的基因組dna為模板,分別以snt2-for1和snt2-rev1、snt2-for2和snt2-rev2、snt2-for3和snt2-rev3、或snt2-for4和snt2-rev4為引物進行pcr擴增,擴增產物包含mosnt2全長基因的啟動子和編碼框的四個重疊片段;同時以gfp-snt2-for和gfp-snt2-rev為引物擴增獲得綠色熒光蛋白的編碼框及終止子;以bar-for和bar-rev為引物擴增草銨膦的抗性篩選標記基因bar;將上述擴增得到的六個dna片段,與經saci和hindiii雙酶切線性化處理的載體pneb-nat(he等,plosone,2012,7(3):e33270)一起,共轉化酵母感受態細胞,在酵母細胞內的重組修復系統的作用下,dna片段和載體即可連接形成重組載體,命名為pneb-mosnt2-gfp-bar。其中所述的引物snt2-for1和snt2-rev1為:
snt2-for1:
5’-aactgttgggaagggcgatcggtgcgggccactagttcattgttaccgaaagaagcc-3’(seqidno:25);
snt2-rev1:5’-aacccttttgagccatgaattcgacgaccaggcaggcaaatc-3’(seqidno:26)。
所述的引物snt2-for2和snt2-rev2為:
snt2-for2:5’-cctgcctggtcgtcgaattcatggctcaaaagggttcaggtg-3’(seqidno:27);
snt2-rev2:5’-agtggattcccaggtagcg-3’(seqidno:28)。
所述的引物snt2-for3和snt2-rev3為:
snt2-for3:5’-tgtaccatgccagcctctg-3’(seqidno:29);
snt2-rev3:5’-tgacgcttccgataatgttct-3’(seqidno:30)。
所述的引物snt2-for4/snt2-rev4為:
snt2-for4:5’-acccaaccagcattcttccc-3’(seqidno:31);
snt2-rev4:5’-agacagtagatttcgcaacg-3’(seqidno:32)。
所述的引物gfp-snt2-for和gfp-snt2-rev為:
gfp-snt2-for:5’-gccagcccttcgttgcgaaatctactgtctcccgggatggtgagcaagggcgagga-3’(seqidno:33);
gfp-snt2-rev:
5’-agtgctccttcaatatcatcttctgtcgacggccggccgtggagatgtggagtgggc-3’(seqidno:34)。
所述的引物bar-for和bar-rev為:
bar-for:5’-gtcgacagaagatgatattg-3’(seqidno:35);
bar-rev:
5’-ttcacacaggaaacagctatgaccatgattatttaaatacgcgtggccggccgtcgacctaaatctcggtgacgg-3’(seqidno:36)。
(2)將步驟(1)中所得的重組載體pneb-mosnt2-gfp-bar轉化至突變體△mosnt2.1原生質體,獲得回復突變體,將回復突變體命名為:compl.(compl.為英文單詞complement縮寫形式,表示補充、恢復的意思)。
實施例6:mosnt2敲除突變體△mosnt2和野生型及回復菌株的對比分析
(1)敲除突變體△mosnt2菌落生長試驗
將野生型guy11、敲除突變體△mosnt2.1、△mosnt2.2和回復菌株compl.分別接種于cm瓊脂培養基上,在25℃條件下培養培養10天,然后觀察菌落。
結果(見圖6)敲除突變體△mosnt2.1和△mosnt2.2的菌落明顯小于野生型菌株guy11和回復菌株compl.的菌落;此外,敲除突變體△mosnt2.1和△mosnt2.2菌落的氣生菌絲很少,而野生型guy11和回復菌株compl.的氣生菌絲濃密。說明mosnt2基因的缺失影響了稻瘟病菌的菌落形態和生長速度。
(2)敲除突變體△mosnt2的產孢量檢測
(a)將野生型菌株guy11、敲除突變體△mosnt2.1和△mosnt2.2、回復菌株compl.分別接種在cm瓊脂培養基上,16小時光照/8小時黑暗,在25℃條件下生長10天,然后在顯微鏡下觀察分生孢子產生情況。
結果(見圖7)野生型guy11和回復菌株compl.的氣生菌絲上分化形成大量分生孢子,而敲除突變體△mosnt2.1和△mosnt2.2的氣生菌絲上則極少形成分生孢子;說明mosnt2基因的缺失使產孢量大大減少。
(b)用5ml無菌水刮取菌落表面的分生孢子,并用擦鏡紙過濾孢懸液,然后于顯微鏡下統計孢子數量。
結果(見圖8)敲除突變體△mosnt2.1和△mosnt2.2產生的孢子數量明顯少于野生型菌株guy11和回復菌株compl.。說明mosnt2在產孢過程中發揮關鍵作用。
(c)在顯微鏡下對敲除突變體△mosnt2的孢子形態進行觀測。
結果(見圖9)野生型guy11和回復菌株compl.的孢子呈現正常的梨形,而敲除突變體△mosnt2.1和△mosnt2.2的分生孢子的形態異常,呈長條形、圓形或棒狀。說明mosnt2在孢子形態建成過程中發揮重要作用。
以上結果說明mosnt2基因在稻瘟病菌的產孢和孢子形態建成過程中發揮重要作用。
(3)敲除突變體△mosnt2對細胞壁脅迫的敏感性試驗
熒光增白劑(calcofluorwhite,cfw)能結合真菌胞壁成分幾丁質,因此會影響真菌胞壁的組裝與結構,對真菌造成細胞壁脅迫。將野生型guy11、敲除突變體△mosnt2.1、△mosnt2.2菌株和回復菌株compl.接種于包含200μg/ml熒光增白劑的cm瓊脂平板上生長5天,然后觀察菌落形態。
結果(見圖10):與野生型guy11與回復菌株compl.相比,敲除突變體△mosnt2.1、△mosnt2.2在cfw脅迫平板上的菌落生長顯著減弱,對cfw更加敏感。這說明mosnt2影響稻瘟病菌細胞壁完整性。
(4)敲除突變體△mosnt2對氧化脅迫的敏感性試驗
向cm瓊脂培養基添加過氧化氫(h2o2),使其終濃度分別為2mm、3mm或5mm,從而形成氧化脅迫生長環境,將野生型guy11、敲除突變體△mosnt2.1、△mosnt2.2菌株和回復菌株compl.接種于添加過氧化氫(h2o2)的cm瓊脂培養基上,在25℃生長10天,然后觀察菌落形態。
結果(見圖11)過氧化氫可明顯抑制各菌株的菌落生長,但它對敲除突變體△mosnt2.1和△mosnt2.2的抑制程度要明顯高于野生型guy11和回復菌株compl.;當過氧化氫濃度為5mm時,guy11與回復菌株compl.均可生長,但敲除突變體△mosnt2.1和△mosnt2.2則完全不能生長。說明敲除突變體對氧化脅迫高度敏感,因此mosnt2是調控稻瘟病菌氧化脅迫應答的重要因子。
(5)敲除突變體△mosnt2侵染器官附著孢觀測試驗
從生長于cm瓊脂培養基的菌落收集分生孢子,制備為孢懸液,然后將孢懸液接種至疏水性玻片(fisherscientific產品),在25℃放置0、10和24小時,然后在顯微鏡下進行觀察。
結果(見圖12)野生型guy11與回復菌株compl.在接種10小時后,均能在疏水性玻片上萌發并分化形成黑色素化的附著胞,但敲除突變體△mosnt2.1和△mosnt2.2雖然可萌發,但卻不能分化形成附著孢;接種24小時后,野生型guy11與回復菌株compl.的附著孢成熟,并且分生孢子由于自噬而坍塌,但敲除突變體△mosnt2.1和△mosnt2.2仍不能形成附著孢,分生孢子保持完整形態而未坍塌。上述結果說明mosnt2在附著胞形成過程中發揮關鍵作用。
實施例7:mosnt2基因敲除突變體的稻瘟病致病性對比試驗
按照如下方法進行:
(1)將稻瘟病菌野生型菌株guy11、敲除突變體△mosnt2.1、△mosnt2.2和回復菌株compl.分別接種于cm瓊脂培養基上,在25℃生長10天,然后對生長有菌絲的瓊脂塊打孔。以實驗室常用高感稻瘟病的水稻品種co39(該水稻材料保存于四川農業大學水稻研究所)為寄主材料。
(2)將菌塊接種在育苗21天的co39的葉片上,未接菌瓊脂塊接種葉片作為對照。接種后5天觀察水稻葉片發病情況。
結果(見圖13)接種野生型guy11和回復菌株compl.的水稻葉片上形成了了典型的稻瘟病病斑,而接種敲除突變體△mosnt2.1和△mosnt2.2的水稻葉片上沒有產生典型的稻瘟病病斑,只形成由超敏反應所引起的急性褐色斑點。
(3)將菌塊接種在育苗21天的co39的根上,未接菌瓊脂塊接種的根作為對照,接種7天后觀察發病情況。
結果(見圖14)野生型guy11和回復菌株compl.可使根部壞死而變為黑色,而敲除突變體△mosnt2.1和△mosnt2.2不能使水稻根部產生黑色壞死斑,因而喪失了對水稻根部的致病性。
以上結果說明了mosnt2基因是稻瘟病菌致病性的關鍵基因。
以上列舉的僅是本發明的若干個具體實施例。但本發明不受以上實施例的限制,本領域的普通技術人員可以從本發明公開的內容直接導出或聯想到許多變形,其他任何未背離本發明的精神實質和原理下所作的改變、修飾、替代、組合、簡化,均應視為等效的置換方式,都應包含在本發明的保護范圍之內。
sequencelisting
<110>四川農業大學
<120>源于稻瘟病菌的致病性基因mosnt2及其用途
<160>36
<170>patentinversion3.5
<210>1
<211>1794
<212>prt
<213>magnaportheoryzae
<400>1
metalaglnlysglyserglyglyseraspalaalathrproasnthr
151015
thrthrthrseralahislysglythralagluserlysglnserser
202530
asnalaproserserglyproserthrthrserserserseralaser
354045
valleuprolysmetseraspproglyproproglnalaleuglyser
505560
proalasergluserthrthrlysserserserthralalysaspala
65707580
thralaglythrthralaserprotyrglythrargserargasnarg
859095
glyglyasnserargproasntyralagluasplysaspileaspmet
100105110
aspilephegluglnleuhisproglnlysargaspaspaspserlys
115120125
lysthrserargglnasnalaserserseralathrasnthrglyasp
130135140
thrhisasnthrproproproproproargthrthrasnglyleuser
145150155160
serarglysproleuprometaspasnlysglnserglnalaalaala
165170175
alalysgluserserserasnalaasnglnalathrglyalaglyser
180185190
thrglyserthrglnthrthrserlyslysarglysalaalasergln
195200205
thrserserasnglnproproalathrgluserthrhisvalproala
210215220
asnalalyslysproserasnasnasnhisasnserasnalaalaser
225230235240
serhisglyalaasplysglytyralaalathrasnleuleuthrphe
245250255
gluasncysglyalaargprolysaspglylysleuilealaaspasp
260265270
glythrtyrleugluvalasnasphisvaltyrleuvalcysglupro
275280285
proglygluprotyrtyrleualaargilemetglupheleuhisala
290295300
lysasnaspproserglnprovalaspalaleuargvalasntrptyr
305310315320
tyrargprolysaspilealaarglysvalasnaspthrargalaval
325330335
phealathrmethisseraspileserproleuthrserleuarggly
340345350
lyscysthrilelyshislysalagluilelysglylysleugluglu
355360365
tyrarglysasnproaspcysphetrppheglulysleutyrasparg
370375380
tyrileglnlysasntyrgluvalileprothrpheglnileileasn
385390395400
valproglulysvallyslysvalleuaspgluargtrplystyrile
405410415
leuvalgluglnglyargglylysgluleuthrseralavallysthr
420425430
cysargargcysserglytyrcysalaserasnaspservalaspcys
435440445
alavalcysgluhisthrtyrhismetasncysvalargproproleu
450455460
leulyslysproserargglyphealatrpsercysalaalacysser
465470475480
argalaglngluarglysleuglualaargasnthrproasnvalser
485490495
leuaspproasnalaglualaglugluglugluphepheaspgluglu
500505510
glugluaspalaglyleuaspthrglyargthrserproalaaspgly
515520525
alaasnaspmethisileproalathrglugluglnmettyrhisala
530535540
serleutrpprotyrargtyrleuglyilehiscyslysvalgluasp
545550555560
alaleuasptyraspaspargiletyrproargalaserthrargval
565570575
glyproarghisglnalathrvalleuasptrpproglyargproval
580585590
glntyrvallysalaprogluilegluilelyslysthrglyarglys
595600605
aspglylysleuasnlysglualaglnalaalaleuglualaglulys
610615620
valalalysalalysargprolystrpileglnaspgluproprogly
625630635640
tyrvalproargglygluasptyrproasnaspaspproargasnthr
645650655
alaglnleuhistrpargproprogluleuaspleuproglugluser
660665670
glyprogluglualahisileserglusergluileasplystyrmet
675680685
gluglnalalysglymetalaleuaspleuaspleuprogluhisser
690695700
thrasnleuleuaspglnalaleuargleuleutyrgluhisglytyr
705710715720
aspalagluargalaleuglugluleuprolysleuserlysgluala
725730735
pheaspgluproglnleuthralaalagluleulyslysphegluglu
740745750
glyileserlyspheglysergluleutyrservallyslyshisile
755760765
lysthrvallysproglythrleuvalargphetyrtyrthrtrplys
770775780
lysthrgluargglylysglnvaltrpglyasntyrserglyarglys
785790795800
serlyslysglualalysglualalyslysalagluthralasergln
805810815
asnlysmetglnaspaspvalalaaspasphisaspaspseralaphe
820825830
aspalaalalysalaalaglulyslysargserpheilecyslysphe
835840845
cysasnthrlysserserargglntrpargargalaproasnalaser
850855860
glyalaleuvalthrgluserglyglylysglyalaasnlysasplys
865870875880
glyvalglntyrvalvalalaleucysargargcysalagluleutrp
885890895
argargtyralaileglntrpgluaspvalaspglnleutyrserlys
900905910
valalaglnalaglyglyargalatrplyslyslysileaspgluglu
915920925
leuleulysgluilevalalaalagluglnargserlysasnthrpro
930935940
serserserglyalaalathrproproserasnthrthrproalapro
945950955960
alaserthrglnproalaalaserglyglngluproalaarglyslys
965970975
glnlysthrthrglnproproglnasplysaspvalglumetthrgly
980985990
thrgluprovalglythrthrthrthralaproalaserlyslyslys
99510001005
glulysalaserleuglulysglulysglulysglulysglulys
101010151020
glulysglulysglulysglulysgluprovallysglulyslys
102510301035
glualaproalaproproprovalprogluileprolysproarg
104010451050
thrmetprocysaspilecysargglnleugluproleuglyasp
105510601065
glnhisilethrcyslysglucysargmetthrvalhisargasn
107010751080
cystyrglyvalvalaspasnargasnproglylystrpvalcys
108510901095
aspmetcysileasnaspargserprohisvalserilehistyr
110011051110
lyscysvalleucysprovalglutyrthrgluhisasppheval
111511201125
gluproprolysvalserhislyslyslysthrglulysasparg
113011351140
gluarggluargglngluargglualaalavalasnalaalaglu
114511501155
histyrarglysargglnglugluleuasnargprovalasnpro
116011651170
arggluproleulysargthralaaspasnasntrpvalhisval
117511801185
thrcysservaltrpthrprogluvallyspheglyasnalalys
119011951200
alaleugluprosergluglyileproserileproargserarg
120512101215
tyrsergluvalcysgluvalcyslysserthrglyglyalacys
122012251230
thrasncysproglncyslysalaservalhisvalglucysala
123512401245
hisglnseraspasptyrvalleuglyphegluilethrproval
125012551260
lysglyserargargaspglnhisasnilevalthrileglygly
126512701275
gluserglysermetseralaservaltrpcyslysleuhisala
128012851290
prolyslysthrvalvalhisglnmettyraspvalvalaspglu
129513001305
alaglythrasnalaleuglnleutyrvalglnasnphelyslys
131013151320
alaaspleuthrleuthrglycysalaarglysalaasnleuile
132513301335
serthralaalaargmetserasnproilevalthrthrthrthr
134013451350
alavalasnargargalaserthrthrthrvalserthrthrthr
135513601365
proseralamethisilehisserleuleuasnglyaspserpro
137013751380
glyaspprohisaspleualavalproglyglylysilecysile
138513901395
thrcysglyvalaspvalserproargtrptyrproileserasp
140014051410
serhisgluarggluleualaasnglyhistyrglyalaleugly
141514201425
thrglualaglnlysphealagluglnarghispheglncyshis
143014351440
lyscyslyslysleuasnlysglnprolysserhisvalpropro
144514501455
proproproproglnaspproproprovalalaalaasnthrile
146014651470
asnproserglyproglualathralahisseralaalavalmet
147514801485
thrasnglyileasphisglyproasnglyvalaspalaalaval
149014951500
argargglythrproproleuthrserproargglnprogluhis
150515101515
asphisileproglyargproserprotyrleutrpglnsergly
152015251530
leuglnalaprohisprothrglyleuvalhisprothrvalpro
153515401545
ileglnglyproproproprometglnalaproproleuglnpro
155015551560
proproilealaproproprometalaargsermetserglyarg
156515701575
glyglnthrglyvalglnproproglyprovalproprosergln
158015851590
alatyrglnproleuproproproproprothrhisseralapro
159516001605
serglyprotyrglyasptrphisargthrthrhishisglypro
161016151620
prometasnglyargproproserargalaserargileserpro
162516301635
ileileproproleualaproproalaleuargproproserleu
164016451650
hishisserprohisalaprohisalahisleuthrasnglyhis
165516601665
metvalasnglyalaglyalaproglyargargleuserglypro
167016751680
proproproproserargaspglyglnglyprotyrmetglyser
168516901695
tyrhisserproalaprotyrhisserproalaprohisglnser
170017051710
asnglythrmetvalproproargileasphisalaphealaser
171517201725
valleuasnproproargalatyrglyasnserglyservalgln
173017351740
proproalahismetserproalavalalaargaspalaproile
174517501755
serargaspglyproleuleuserglnproproproproalaarg
176017651770
alaprogluserargproalathrglyalaseralaserproser
177517801785
leuargasnleuleuser
1790
<210>2
<211>5382
<212>dna
<213>magnaportheoryzae
<400>2
atggctcaaaagggttcaggtggctcagatgccgcaacacctaataccacgacgacgtcg60
gctcacaaaggcaccgctgagagcaagcaatcttccaatgcgccctcttctgggccatcg120
acgacatcgtcgtcatctgcatcggttctgcccaaaatgtcagatcccggtccaccacag180
gccctaggaagtccagcgtcagaatcgacaaccaagtcgagttcgactgccaaggacgcg240
acggccggcaccacggcatctccttatggcacgcgatctcgcaacaggggcggcaactct300
cgtcccaattatgcagaggacaaggacattgatatggacatttttgagcagttgcatcct360
cagaagcgagatgacgacagcaagaagacgtcgcgccaaaacgcatcgtcgtcagccacc420
aacactggcgatacacacaacacacctccgccaccacctcgcaccactaacgggttgtcg480
tccagaaagccgctgcccatggataacaagcagtcccaggctgcagccgccaaagagtca540
tcctcaaatgctaaccaggcaaccggcgctggctcaactggatccacacagaccacatcg600
aaaaagcgaaaagctgcatcacaaactagcagtaatcagccaccagcgacggaatcgacg660
cacgtgccagccaacgccaagaagcccagtaataataatcacaacagcaacgctgcatcg720
agccacggcgccgacaagggatatgctgctactaatttgctcactttcgaaaactgtggc780
gcgcggcccaaagatggcaaattgattgctgacgatggcacatatcttgaagtcaatgac840
catgtctacctggtttgcgagcccccaggcgaaccctattatctcgccaggattatggag900
ttcttgcatgcaaagaacgacccttcgcagccggtcgatgctttgcgtgtgaattggtac960
taccgcccaaaagatattgcccgtaaggtgaacgatactagggcggtttttgcaaccatg1020
cactcggacattagccctttgacatcgctccgcggcaaatgcacgatcaagcacaaggca1080
gagatcaagggcaagctcgaagagtaccgcaaaaatcccgattgtttctggttcgagaaa1140
ctctacgacaggtacatccagaaaaattacgaggtgatcccgacgtttcagatcatcaat1200
gtacccgagaaggtcaaaaaggtccttgacgagaggtggaaatacatcctggtcgagcag1260
ggccgcggcaaggagctcaccagcgccgtcaagacgtgcaggaggtgctctggctattgc1320
gcaagtaacgattctgtcgactgcgctgtgtgtgagcacacgtaccacatgaactgtgtg1380
cgaccacccctgctcaagaagccatcacgtggctttgcctggtcctgtgccgcctgcagt1440
agagcccaggagcgcaagctggaggctcgcaacacgccaaatgtcagcctggatccgaat1500
gcagaggccgaggaggaggagtttttcgatgaggaggaggaggacgctggtctcgacacc1560
ggccgcaccagtcctgccgatggcgccaacgatatgcacatccctgcaaccgaggagcag1620
atgtaccatgccagcctctggccatatcgctacctgggaatccactgcaaggtggaggac1680
gccctcgattacgacgaccgaatatatccccgtgcatcgactcgtgttggcccaagacac1740
caggcgacggtgcttgactggcctggtcgaccggtccaatacgtcaaggctcccgaaatt1800
gaaataaaaaagacgggtcgtaaagacgggaagctgaacaaggaagcgcaagctgccctc1860
gaggccgaaaaggtcgccaaggccaaacgtcccaagtggatccaggatgaaccccctggc1920
tatgtgccccgtggtgaggactaccccaatgacgatccccggaataccgcccagcttcac1980
tggagacctcctgagcttgacctcccggaggaatcgggtccggaggaggcgcatatttcc2040
gaatcggagatcgacaagtacatggagcaagcgaaaggaatggcgctagacctcgacctc2100
ccggagcactctacgaatctgctggaccaggcgctccgacttctctacgaacacggttac2160
gacgcggagcgtgcactggaagagctccccaagctgagtaaggaagcttttgacgaacca2220
cagctcacagctgccgagctcaaaaagttcgaggaaggcatctccaagttcggctcagag2280
ctttacagcgtgaagaaacatatcaagaccgtcaaaccagggacgcttgttcgtttctac2340
tacacatggaagaagacagagcgcggaaagcaggtgtggggcaactactctggccgcaag2400
agcaagaaggaggccaaggaagccaagaaagcggagacagcctcacagaataagatgcag2460
gacgacgtcgcagacgaccacgatgactcggctttcgacgcagctaaagcggcggaaaag2520
aaacggtctttcatttgcaagttttgcaacaccaagagctcgcgtcagtggagacgcgcg2580
ccaaatgcatcaggcgctctggtcacggagagcgggggcaagggtgccaacaaggacaag2640
ggcgttcagtatgtcgtggcattgtgtcgaagatgtgctgagctctggcgccgctatgcc2700
attcagtgggaggatgtcgaccagctctacagcaaggtggcacaggccggtggccgtgcg2760
tggaagaagaagatcgacgaggagctgctcaaggagatcgttgctgctgagcaaagaagc2820
aagaacacgccaagcagtagcggtgcggctactccgccatcgaacaccacgcctgcgccc2880
gcttccactcaaccagcagcttcgggtcaagaaccggcgcggaagaaacagaagacaacg2940
cagccgcctcaagacaaggatgtggaaatgaccggcacggagcctgttggcacgacaaca3000
acggcgcccgcatccaagaaaaaggagaaggccagcttggaaaaggagaaagagaaggaa3060
aaggagaaagagaaggaaaaggagaaggagaaagaaccagtcaaagaaaagaaggaagcg3120
ccggctcctccccctgttccggagatacccaagccgcgaacaatgccttgcgatatctgt3180
cggcagctggaacctctgggcgaccagcatatcacgtgcaaagaatgtcgcatgacagtg3240
catcggaactgctatggcgtcgtcgacaaccgcaaccctgggaagtgggtgtgtgacatg3300
tgcatcaacgacagaagtccgcatgtctccattcattacaaatgtgtcttgtgtcctgtt3360
gaatacaccgagcacgattttgtggaaccgccaaaggtatctcataaaaagaagacagaa3420
aaggatcgcgagcgcgagcgccaagaacgtgaggctgcggtcaacgcagcagaacattat3480
cggaagcgtcaggaagagctgaaccgcccggtcaatcctcgtgagcctctcaaacgaaca3540
gccgacaacaattgggtacatgtcacttgctcggtgtggactcctgaggtcaagtttggc3600
aatgccaaagctctcgagcccagcgaagggatcccttctatcccgagatcgaggtactcg3660
gaggtttgcgaggtctgcaaatctacgggcggtgcctgcacaaactgccctcagtgcaag3720
gcatcagtgcatgtcgagtgtgcccaccagtcagacgactacgtcctaggttttgagatc3780
actcccgtcaagggatcacgccgcgatcagcacaacatcgtaactattggtggtgagagc3840
ggctccatgagcgcgtctgtctggtgcaagcttcatgcaccgaagaagactgtcgtgcat3900
caaatgtacgatgttgtcgacgaagcgggaacgaacgctttacaactatacgtgcagaac3960
ttcaagaaggcagatcttactctcactggctgcgcacgaaaggccaacttgataagcact4020
gctgcgcgcatgtccaacccgatagtgacgacgactacggcggtgaatcgcagagcatcg4080
accacgacggtttcaacaaccaccccttcggcaatgcatattcacagtctcctcaacggg4140
gacagccctggagaccctcacgacctagcagtcccaggtggcaagatatgtataacatgc4200
ggagtcgacgtcagccctcgatggtatcccatcagcgacagtcacgagcgggagcttgca4260
aacggtcactatggtgctctcggcactgaggcgcagaagtttgctgagcagcgacatttc4320
cagtgtcacaaatgcaagaagctcaacaagcaacccaagtctcacgtgccaccgccgccg4380
ccgccgcaagatcctccgccggttgctgcaaatacgataaatccctccgggcctgaagca4440
acagcgcattcagcagctgtaatgacgaacggcatagatcacggtcccaatggcgtcgat4500
gcagcggttcgtcgaggcacaccacctctaacaagccctcgccaaccagagcacgatcat4560
ataccgggtaggcctagcccgtacctatggcaatccggacttcaggctccacatccgacc4620
ggcttggtgcatcctacggtccctatacaggggccgccacccccgatgcaggcaccgcct4680
ctccaaccaccgccgatagctccgccgccgatggcacgtagtatgtcaggccgcggtcaa4740
accggtgtacagcctccggggccagtaccgccctcccaagcatatcaacccttgccgccg4800
cctcctcccactcattcggcaccatctggcccctacggcgattggcatcggacgactcat4860
cacggacccccaatgaatggtcgtcctccttcacgcgcatcacggatttccccgatcata4920
ccacctctggcaccaccagcacttcgtccaccgtctctccaccactctcctcatgctcct4980
cacgcgcatctgaccaacggccacatggtcaacggtgcgggtgcaccaggaagacgatta5040
agcgggccgccaccgccaccttcgcgagatggtcagggcccatacatgggaagctatcat5100
tctcctgcaccataccatagcccagcgccgcaccaaagcaacgggacaatggtgccacct5160
cgcatagatcatgcttttgcgtcagtcctcaaccctccgagagcatatggaaacagcggg5220
agcgtgcagccgcctgcgcacatgagcccagctgtggcgagggacgctcctatttctagg5280
gatggaccacttctttcccagccaccgccaccggccagggcacctgagtcgagacctgca5340
acaggtgccagcgccagcccttcgttgcgaaatctactgtct5382
<210>3
<211>22
<212>dna
<213>artificialsequence
<220>
<223>snt2-cdna-for
<400>3
atggctcaaaagggttcaggtg22
<210>4
<211>23
<212>dna
<213>artificialsequence
<220>
<223>snt2-cdna-rev
<400>4
agacagtagatttcgcaacgaag23
<210>5
<211>19
<212>dna
<213>artificialsequence
<220>
<223>rp1
<400>5
caggattatggagttcttg19
<210>6
<211>19
<212>dna
<213>artificialsequence
<220>
<223>rp2
<400>6
ctagtatcgttcaccttac19
<210>7
<211>1167
<212>dna
<213>artificialsequence
<220>
<223>lf
<400>7
aatccctgcgggctactttgacgatactacgcaccgcagagcctgcaaaaagctgtagct60
ggcggtcccatacaccggacccatatattcgaagctagctcactaaatcgattggacaag120
ggtgtggggtcccacccagggactgatgtaggaagtgcaaaaaaatggacgagccttgcg180
ggcgccgccagcccttgttgctttctgcgtgccgggcaagccagccagccagtacgggac240
gtccccagttgggtttggtcttccatgtctctttcttgtcttgggggcgtgtggtccgtc300
catcatatccgcctttctgggacatgcatccattgaccatctttctctaaatcctttctt360
cgcggcactccatctctttcctcactttccacttttccgccttccaagaggtcagtcgtt420
gtgcactcccacgcgcccccgacgaaggacatttttgcttccctctcggcaccaggcaag480
caagccagccagtccctctctacatgatcggattaccagcccggtttgggttcccagcgc540
cacaacgcttgtaactcgcgcgaagggctgctggccttcaaactcgattcctcataacat600
ttgctcgtgcagatctaactcaccgggcatctcgggggacacgaacacccgcccacttcc660
ttaacttctttgtcaatcgcccatatatctctccttttcctattccattcccccccacac720
aattcaaaccttgcacctttctcacccatttcgacataaactgggtccgactttctttcc780
tttcctccttgtcacccaaccttgggtttcgaaaccttcgatttccatctgtacccatca840
atctcttctggcatgcattatccgccacacgctcacctgaatctgggagtctcatcactg900
tttacgtcgcgcattgcggcctgcccagagcagttgtattctgcttcttgcggctgcgcc960
gcctacctttccttcccatagttgtttctttgcagcggaaggcgaaatttgatcgacccg1020
gcctctgggtctcacttgggaccttataaacagcgcccacgtccagcagatttgcctgcc1080
tggtcgtcatggctcaaaagggttcaggtggctcagatgccgcaacacctaataccacga1140
cgacgtcggctcacaaaggcaccgctg1167
<210>8
<211>1026
<212>dna
<213>artificialsequence
<220>
<223>rf
<400>8
gcatctccaagttcggctcagagctttacagcgtgaagaaacatatcaagaccgtcaaac60
cagggacgcttgttcgtttctactacacatggaagaagacagagcgcggaaagcaggtgt120
ggggcaactactctggccgcaagagcaagaaggaggccaaggaagccaagaaagcggaga180
cagcctcacagaataagatgcaggacgacgtcgcagacgaccacgatgactcggctttcg240
acgcagctaaagcggcggaaaagaaacggtctttcatttgcaagttttgcaacaccaaga300
gctcgcgtcagtggagacgcgcgccaaatgcatcaggcgctctggtcacggagagcgggg360
gcaagggtgccaacaaggacaagggcgttcagtatgtcgtggcattgtgtcgaagatgtg420
ctgagctctggcgccgctatgccattcagtgggaggatgtcgaccagctctacagcaagg480
tggcacaggccggtggccgtgcgtggaagaagaagatcgacgaggagctgctcaaggaga540
tcgttgctgctgagcaaagaagcaagaacacgccaagcagtagcggtgcggctactccgc600
catcgaacaccacgcctgcgcccgcttccactcaaccagcagcttcgggtcaagaaccgg660
cgcggaagaaacagaagacaacgcagccgcctcaagacaaggatgtggaaatgaccggca720
cggagcctgttggcacgacaacaacggcgcccgcatccaagaaaaaggagaaggccagct780
tggaaaaggagaaagagaaggaaaaggagaaagagaaggaaaaggagaaggagaaagaac840
cagtcaaagaaaagaaggaagcgccggctcctccccctgttccggagatacccaagccgc900
gaacaatgccttgcgatatctgtcggcagctggaacctctgggcgaccagcatatcacgt960
gcaaagaatgtcgcatgacagtgcatcggaactgctatggcgtcgtcgacaaccgcaacc1020
ctggga1026
<210>9
<211>18
<212>dna
<213>artificialsequence
<220>
<223>snt2-lf-for
<400>9
aatccctgcgggctactt18
<210>10
<211>42
<212>dna
<213>artificialsequence
<220>
<223>snt2-lf-rev
<400>10
ccttcaatatcatcttctgtcgacctcacaaaggcaccgctg42
<210>11
<211>50
<212>dna
<213>artificialsequence
<220>
<223>snt2-rf-for
<400>11
cccagcactcgtccgagggcaaaggaataggcatctccaagttcggctca50
<210>12
<211>18
<212>dna
<213>artificialsequence
<220>
<223>snt2-rf-rev
<400>12
acaaccgcaaccctggga18
<210>13
<211>16
<212>dna
<213>artificialsequence
<220>
<223>hyg-for
<400>13
gtcgacagaagatgat16
<210>14
<211>20
<212>dna
<213>artificialsequence
<220>
<223>hy-split
<400>14
tacttcgagcggaggcatcc20
<210>15
<211>20
<212>dna
<213>artificialsequence
<220>
<223>yg-split
<400>15
cgttgcaagacctgcctgaa20
<210>16
<211>21
<212>dna
<213>artificialsequence
<220>
<223>hyg-rev
<400>16
ctattcctttgccctcggacg21
<210>17
<211>1148
<212>dna
<213>artificialsequence
<220>
<223>hy
<400>17
gtcgacagaagatgatattgaaggagcactttttgggcttggctggagctagtggaggtc60
aacaatgaatgcctattttggtttagtcgtccaggcggtgagcacaaaatttgtgtcgtt120
tgacaagatggttcatttaggcaactggtcagatcagccccacttgtagcagtagcggcg180
gcgctcgaagtgtgactcttattagcagacaggaacgaggacattattatcatctgctgc240
ttggtgcacgataacttggtgcgtttgtcaagcaaggtaagtgaacgacccggtcatacc300
ttcttaagttcgcccttcctccctttatttcagattcaatctgacttacctattctaccc360
aagcaacgcttcgattaggaagtaaccatgaaaaagcctgaactcaccgcgacgtctgtc420
gagaagtttctgatcgaaaagttcgacagcgtctccgacctgatgcagctctcggagggc480
gaagaatctcgtgctttcagcttcgatgtaggagggcgtggatatgtcctgcgggtaaat540
agctgcgccgatggtttctacaaagatcgttatgtttatcggcactttgcatcggccgcg600
ctcccgattccggaagtgcttgacattggggaattcagcgagagcctgacctattgcatc660
tcccgccgtgcacagggtgtcacgttgcaagacctgcctgaaaccgaactgcccgctgtt720
ctgcagccggtcgcggaggccatggatgcgatcgctgcggccgatcttagccagacgagc780
gggttcggcccattcggaccgcaaggaatcggtcaatacactacatggcgtgatttcata840
tgcgcgattgctgatccccatgtgtatcactggcaaactgtgatggacgacaccgtcagt900
gcgtccgtcgcgcaggctctcgatgagctgatgctttgggccgaggactgccccgaagtc960
cggcacctcgtgcacgcggatttcggctccaacaatgtcctgacggacaatggccgcata1020
acagcggtcattgactggagcgaggcgatgttcggggattcccaatacgaggtcgccaac1080
atcttcttctggaggccgtggttggcttgtatggagcagcagacgcgctacttcgagcgg1140
aggcatcc1148
<210>18
<211>731
<212>dna
<213>artificialsequence
<220>
<223>yg
<400>18
cgttgcaagacctgcctgaaaccgaactgcccgctgttctgcagccggtcgcggaggcca60
tggatgcgatcgctgcggccgatcttagccagacgagcgggttcggcccattcggaccgc120
aaggaatcggtcaatacactacatggcgtgatttcatatgcgcgattgctgatccccatg180
tgtatcactggcaaactgtgatggacgacaccgtcagtgcgtccgtcgcgcaggctctcg240
atgagctgatgctttgggccgaggactgccccgaagtccggcacctcgtgcacgcggatt300
tcggctccaacaatgtcctgacggacaatggccgcataacagcggtcattgactggagcg360
aggcgatgttcggggattcccaatacgaggtcgccaacatcttcttctggaggccgtggt420
tggcttgtatggagcagcagacgcgctacttcgagcggaggcatccggagcttgcaggat480
cgccgcggctccgggcgtatatgctccgcattggtcttgaccaactctatcagagcttgg540
ttgacggcaatttcgatgatgcagcttgggcgcagggtcgatgcgacgcaatcgtccgat600
ccggagccgggactgtcgggcgtacacaaatcgcccgcagaagcgcggccgtctggaccg660
atggctgtgtagaagtactcgccgatagtggaaaccgacgccccagcactcgtccgaggg720
caaaggaatag731
<210>19
<211>2315
<212>dna
<213>artificialsequence
<220>
<223>lf-hy
<400>19
aatccctgcgggctactttgacgatactacgcaccgcagagcctgcaaaaagctgtagct60
ggcggtcccatacaccggacccatatattcgaagctagctcactaaatcgattggacaag120
ggtgtggggtcccacccagggactgatgtaggaagtgcaaaaaaatggacgagccttgcg180
ggcgccgccagcccttgttgctttctgcgtgccgggcaagccagccagccagtacgggac240
gtccccagttgggtttggtcttccatgtctctttcttgtcttgggggcgtgtggtccgtc300
catcatatccgcctttctgggacatgcatccattgaccatctttctctaaatcctttctt360
cgcggcactccatctctttcctcactttccacttttccgccttccaagaggtcagtcgtt420
gtgcactcccacgcgcccccgacgaaggacatttttgcttccctctcggcaccaggcaag480
caagccagccagtccctctctacatgatcggattaccagcccggtttgggttcccagcgc540
cacaacgcttgtaactcgcgcgaagggctgctggccttcaaactcgattcctcataacat600
ttgctcgtgcagatctaactcaccgggcatctcgggggacacgaacacccgcccacttcc660
ttaacttctttgtcaatcgcccatatatctctccttttcctattccattcccccccacac720
aattcaaaccttgcacctttctcacccatttcgacataaactgggtccgactttctttcc780
tttcctccttgtcacccaaccttgggtttcgaaaccttcgatttccatctgtacccatca840
atctcttctggcatgcattatccgccacacgctcacctgaatctgggagtctcatcactg900
tttacgtcgcgcattgcggcctgcccagagcagttgtattctgcttcttgcggctgcgcc960
gcctacctttccttcccatagttgtttctttgcagcggaaggcgaaatttgatcgacccg1020
gcctctgggtctcacttgggaccttataaacagcgcccacgtccagcagatttgcctgcc1080
tggtcgtcatggctcaaaagggttcaggtggctcagatgccgcaacacctaataccacga1140
cgacgtcggctcacaaaggcaccgctggtcgacagaagatgatattgaaggagcactttt1200
tgggcttggctggagctagtggaggtcaacaatgaatgcctattttggtttagtcgtcca1260
ggcggtgagcacaaaatttgtgtcgtttgacaagatggttcatttaggcaactggtcaga1320
tcagccccacttgtagcagtagcggcggcgctcgaagtgtgactcttattagcagacagg1380
aacgaggacattattatcatctgctgcttggtgcacgataacttggtgcgtttgtcaagc1440
aaggtaagtgaacgacccggtcataccttcttaagttcgcccttcctccctttatttcag1500
attcaatctgacttacctattctacccaagcaacgcttcgattaggaagtaaccatgaaa1560
aagcctgaactcaccgcgacgtctgtcgagaagtttctgatcgaaaagttcgacagcgtc1620
tccgacctgatgcagctctcggagggcgaagaatctcgtgctttcagcttcgatgtagga1680
gggcgtggatatgtcctgcgggtaaatagctgcgccgatggtttctacaaagatcgttat1740
gtttatcggcactttgcatcggccgcgctcccgattccggaagtgcttgacattggggaa1800
ttcagcgagagcctgacctattgcatctcccgccgtgcacagggtgtcacgttgcaagac1860
ctgcctgaaaccgaactgcccgctgttctgcagccggtcgcggaggccatggatgcgatc1920
gctgcggccgatcttagccagacgagcgggttcggcccattcggaccgcaaggaatcggt1980
caatacactacatggcgtgatttcatatgcgcgattgctgatccccatgtgtatcactgg2040
caaactgtgatggacgacaccgtcagtgcgtccgtcgcgcaggctctcgatgagctgatg2100
ctttgggccgaggactgccccgaagtccggcacctcgtgcacgcggatttcggctccaac2160
aatgtcctgacggacaatggccgcataacagcggtcattgactggagcgaggcgatgttc2220
ggggattcccaatacgaggtcgccaacatcttcttctggaggccgtggttggcttgtatg2280
gagcagcagacgcgctacttcgagcggaggcatcc2315
<210>20
<211>1757
<212>dna
<213>artificialsequence
<220>
<223>yg-rf
<400>20
cgttgcaagacctgcctgaaaccgaactgcccgctgttctgcagccggtcgcggaggcca60
tggatgcgatcgctgcggccgatcttagccagacgagcgggttcggcccattcggaccgc120
aaggaatcggtcaatacactacatggcgtgatttcatatgcgcgattgctgatccccatg180
tgtatcactggcaaactgtgatggacgacaccgtcagtgcgtccgtcgcgcaggctctcg240
atgagctgatgctttgggccgaggactgccccgaagtccggcacctcgtgcacgcggatt300
tcggctccaacaatgtcctgacggacaatggccgcataacagcggtcattgactggagcg360
aggcgatgttcggggattcccaatacgaggtcgccaacatcttcttctggaggccgtggt420
tggcttgtatggagcagcagacgcgctacttcgagcggaggcatccggagcttgcaggat480
cgccgcggctccgggcgtatatgctccgcattggtcttgaccaactctatcagagcttgg540
ttgacggcaatttcgatgatgcagcttgggcgcagggtcgatgcgacgcaatcgtccgat600
ccggagccgggactgtcgggcgtacacaaatcgcccgcagaagcgcggccgtctggaccg660
atggctgtgtagaagtactcgccgatagtggaaaccgacgccccagcactcgtccgaggg720
caaaggaataggcatctccaagttcggctcagagctttacagcgtgaagaaacatatcaa780
gaccgtcaaaccagggacgcttgttcgtttctactacacatggaagaagacagagcgcgg840
aaagcaggtgtggggcaactactctggccgcaagagcaagaaggaggccaaggaagccaa900
gaaagcggagacagcctcacagaataagatgcaggacgacgtcgcagacgaccacgatga960
ctcggctttcgacgcagctaaagcggcggaaaagaaacggtctttcatttgcaagttttg1020
caacaccaagagctcgcgtcagtggagacgcgcgccaaatgcatcaggcgctctggtcac1080
ggagagcgggggcaagggtgccaacaaggacaagggcgttcagtatgtcgtggcattgtg1140
tcgaagatgtgctgagctctggcgccgctatgccattcagtgggaggatgtcgaccagct1200
ctacagcaaggtggcacaggccggtggccgtgcgtggaagaagaagatcgacgaggagct1260
gctcaaggagatcgttgctgctgagcaaagaagcaagaacacgccaagcagtagcggtgc1320
ggctactccgccatcgaacaccacgcctgcgcccgcttccactcaaccagcagcttcggg1380
tcaagaaccggcgcggaagaaacagaagacaacgcagccgcctcaagacaaggatgtgga1440
aatgaccggcacggagcctgttggcacgacaacaacggcgcccgcatccaagaaaaagga1500
gaaggccagcttggaaaaggagaaagagaaggaaaaggagaaagagaaggaaaaggagaa1560
ggagaaagaaccagtcaaagaaaagaaggaagcgccggctcctccccctgttccggagat1620
acccaagccgcgaacaatgccttgcgatatctgtcggcagctggaacctctgggcgacca1680
gcatatcacgtgcaaagaatgtcgcatgacagtgcatcggaactgctatggcgtcgtcga1740
caaccgcaaccctggga1757
<210>21
<211>20
<212>dna
<213>artificialsequence
<220>
<223>p1
<400>21
ggtaggagaaaggctgatgg20
<210>22
<211>22
<212>dna
<213>artificialsequence
<220>
<223>p2
<400>22
gcttctgcgggcgatttgtgta22
<210>23
<211>22
<212>dna
<213>artificialsequence
<220>
<223>p3
<400>23
cagcgagagcctgacctattgc22
<210>24
<211>20
<212>dna
<213>artificialsequence
<220>
<223>p4
<400>24
cttcctgacgcttccgataa20
<210>25
<211>57
<212>dna
<213>artificialsequence
<220>
<223>snt2-for1
<400>25
aactgttgggaagggcgatcggtgcgggccactagttcattgttaccgaaagaagcc57
<210>26
<211>42
<212>dna
<213>artificialsequence
<220>
<223>snt2-rev1
<400>26
aacccttttgagccatgaattcgacgaccaggcaggcaaatc42
<210>27
<211>42
<212>dna
<213>artificialsequence
<220>
<223>snt2-for2
<400>27
cctgcctggtcgtcgaattcatggctcaaaagggttcaggtg42
<210>28
<211>19
<212>dna
<213>artificialsequence
<220>
<223>snt2-rev2
<400>28
agtggattcccaggtagcg19
<210>29
<211>19
<212>dna
<213>artificialsequence
<220>
<223>snt2-for3
<400>29
tgtaccatgccagcctctg19
<210>30
<211>21
<212>dna
<213>artificialsequence
<220>
<223>snt2-rev3
<400>30
tgacgcttccgataatgttct21
<210>31
<211>20
<212>dna
<213>artificialsequence
<220>
<223>snt2-for4
<400>31
acccaaccagcattcttccc20
<210>32
<211>20
<212>dna
<213>artificialsequence
<220>
<223>snt2-rev4
<400>32
agacagtagatttcgcaacg20
<210>33
<211>56
<212>dna
<213>artificialsequence
<220>
<223>gfp-snt2-for
<400>33
gccagcccttcgttgcgaaatctactgtctcccgggatggtgagcaagggcgagga56
<210>34
<211>57
<212>dna
<213>artificialsequence
<220>
<223>gfp-snt2-rev
<400>34
agtgctccttcaatatcatcttctgtcgacggccggccgtggagatgtggagtgggc57
<210>35
<211>20
<212>dna
<213>artificialsequence
<220>
<223>bar-for
<400>35
gtcgacagaagatgatattg20
<210>36
<211>75
<212>dna
<213>artificialsequence
<220>
<223>bar-rev
<400>36
ttcacacaggaaacagctatgaccatgattatttaaatacgcgtggccggccgtcgacct60
aaatctcggtgacgg75
- 還沒有人留言評論。精彩留言會獲得點贊!
- 水稻稻瘟病抗性基因Pi2?1的分子標記方法及應用與流程
- 一種稻瘟病抗性基因Piz?t功能特異性分子標記及其檢測方法和應用與流程
- 一種接種架及稻瘟病菌接種系統的制作方法與工藝
- 谷梅4號抗稻瘟病基因Pigm的分子標記及其應用的制作方法與工藝
- 水稻稻瘟病抗性基因Pita、Pib分子標記方法與流程
- 一種基于豬非典型瘟病毒E2基因的熒光定量RT?PCR檢測試劑盒的制作方法與工藝
- 稻瘟病菌LAMP檢測引物組及應用的制造方法與工藝
- 一種制備稻瘟病菌促分裂原活化蛋白激酶MoMps1晶體的方法與流程
- 水稻抗稻瘟病Pi9基因的共顯性功能分子標記Pi9InDel1及其應用的制造方法與工藝
- 水稻抗稻瘟病Pi9基因的共顯性功能分子標記Pi9InDel2及其應用的制造方法與工藝