采用鋅指核酸酶技術破壞人bcr?abl融合基因以抑制CML細胞增殖和促使其凋亡的制作方法
本發明屬于分子生物學技術領域,具體涉及一種采用鋅指核酸酶技術破壞人bcr-abl融合基因以抑制cml細胞增殖和促使其凋亡。
背景技術:
慢性粒細胞白血病(chronicmyeloidleukemia,cml)的致病根源是由于t(9;22)(q34;q11)平衡易位導致c-abl基因與bcr基因形成bcr-abl融合基因。該融合基因編碼的bcr-abl融合蛋白具有強烈的酪氨酸激酶活性,持續激活下游的ras/mapk,pi3k/akt,stat5等促增殖抑凋亡信號,導致細胞的惡性轉化。臨床上的一線治療藥物伊馬替尼是一種酪氨酸激酶抑制劑(tyrosinekinaseinhibitors,tkis),可以使近70%的cml慢性期患者達到血液學甚至遺傳學的完全緩解,但是,另外30%的患者由于bcr-abl激酶區突變引發耐藥,尤其是t315i突變,即使是新開發的第二代酪氨酸激酶抑制劑,也束手無策;并且,酪氨酸激酶抑制劑只能抑制abl激酶活性,不能使bcr-abl基因轉為陰性,特別是白血病干細胞(leukemiastemcell,lsc)對tkis不敏感,殘留的lsc成為復發的根源。因此,探索新的根治cml的方法迫在眉睫。
細胞本身存在一種dna損傷-修復機制,當dna雙鏈的一條鏈斷裂后,以另外一條鏈為模板進行同源修復;當dna雙鏈的兩條鏈均斷裂后,以同源染色體的dna雙鏈為模板進行同源修復,從而確保基因組的穩定。當兩條染色體的dna雙鏈均斷裂后,以非同源末端連接(nonhomologousendjoining,nhej)的方式進行修復,這種修復方式極易發生插入、缺失致移碼突變。利用細胞的這種損傷-修復原理,誕生了一種可以定點操作基因組的核酸酶修飾技術。它由特異性的dna識別結合結構域和非特異性剪切dna的核酸酶組成。該技術可以靶向特定位點的染色體dna雙鏈斷裂(double-strandedbreaks,dsb),以供體dna為模板,促發同源定向修復(homologydirectedrepair,hdr),或者以極易出錯的nhej方式進行修復。其中hdr可對靶基因進行定點插入、缺失、修正等操作,而nhej由于極易出錯,很容易導致靶基因發生插入、缺失和移碼突變,從而實現對靶基因的破壞(genedisruption)。
目前,主要的核酸酶修飾技術包括鋅指核酸酶(zincfingernucleases,zfns),類轉錄激活因子效應物核酸酶(transcriptionactivator-likeeffectornuclease,tal-ens)和規律成簇間隔短回文重復/cas核酸酶(clusteredregularlyinterspacedshortpalindromicrepeats/crispr-associatedsystems,crispr/cas)核酸酶。crispr/ca-s核酸酶技術起步較晚,該技術操作起來最簡單,但是,其潛在的完全性問題尚未得到證實。talens的主要優勢在于不受靶序列特征的限制,但是特異識別靶序列的talens片段太長,不利于后續載體的裝載和表達,并且在靶細胞內引入大量的重復序列也是未知的安全隱患。相比而言,雖然zfns構建過程較復雜,但其是人類基因編輯最確立的技術,技術成熟,免疫原性低,特異性較高,較適用于體內療法,且已在基因治療領域顯示出良好的前景,是本研究較為理想的工具。
技術實現要素:
針對以上問題,本發明一方面提供一種dna分子,其序列如seqidno:2所示。
本發明的另一方面在于提供如seqidno:2所示的dna分子作為靶點在抑制人bcr-abl融合基因表達或導致人bcr-abl基因功能喪失中的應用;所述抑制人bcr-abl基因表達或導致人bcr-abl基因功能喪失是由鋅指核酸酶介導的bcr-abl融合基因同源重組技術帶來的。
在上述技術方案中,所述人bcr-abl基因的核苷酸序列如seqidno:1所示。
所述的鋅指核酸酶的鋅指蛋白的核苷酸編碼序列如seqidno:3和4所示。
所述的同源重組為同源定向修復,使用到的供體dna的左臂擴增引物如seqidno:7和8所示,右臂擴增引物如seqidno:9和10所示。
本發明的有益效果是:本發明定位了bcr-abl基因中一段特異的靶序列位點dna片段,并針對此特異位點構建zfn質粒,將此質粒核轉染k562細胞可以使bcr-abl基因發生定點斷裂,并以供體dna為模板進行同源修復,使其插入8個堿基最終導致bcr-abl基因的破壞。本發明的顯著優勢表現在針對bcr-abl序列中特異的靶序列位點構建的zfn質粒可高效靶向bcr-abl基因發生dna雙鏈斷裂,以hdr方式進行修復使bcr-abl發生移碼等突變而被破壞,從而喪失促增殖、抑凋亡等惡性轉化潛能,證實zfns靶向破壞bcr-abl基因殺傷和抑制cml細胞的可行性。
附圖說明
圖1是鑒定轉染zfn質粒的k562細胞基因組dna測序結果峰圖。
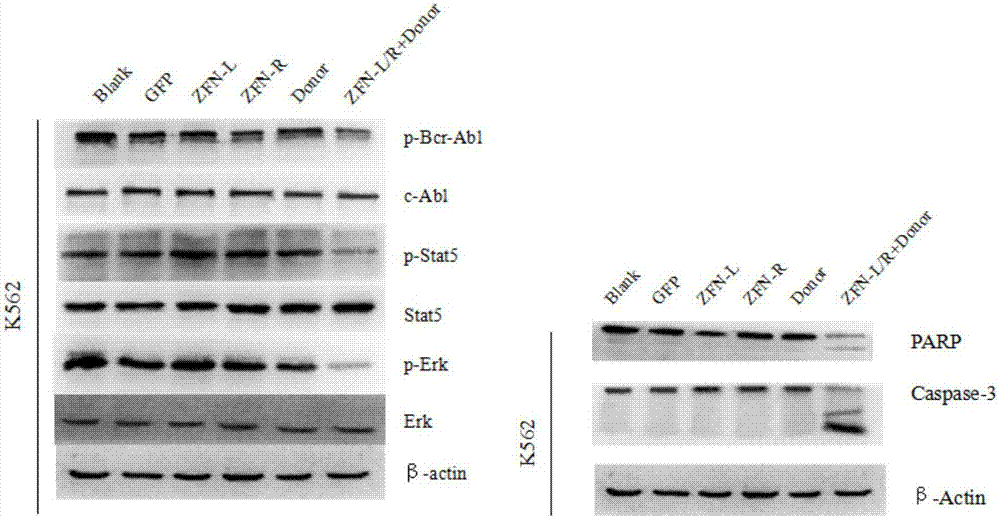
圖2是k562細胞相關蛋白westernblot檢測結果圖,blank:空白對照組;gfp:空載組。
圖3是k562細胞、gfp和zfn-l/r+donor質粒核轉染k562細胞克隆形成鏡下圖,k562細胞:未處理細胞,gfp:空載質粒,zfn-l/r+donor:zfns和donor同時作用于k562細胞。
圖4是gfp和zfn-l/r+donor質粒核轉染k562細胞后細胞克隆形成數柱狀圖。
具體實施方式
以下實施例用于說明本發明,但不用來限制本發明的范圍。
實施例1、針對人bcr-abl基因特異靶位點序列的zfn質粒和donor質粒的設計與構建
一、針對人bcr-abl基因特異靶位點序列的鋅指核酸酶的設計合成
根據ncbi的查詢和拼接確定了人bcr-abl基因的基因序列(如seqidno:1所示),通過生物信息學的方法,完成zfns設計,確定了該人bcr-abl基因的鋅指核酸酶作用的特異靶位點序列為:
ggcgtcgacggcgactacgaggacgccgag(如seqidno:2所示);中間部分序列(gactac)為foki內切核酸酶切割位點,也即zfn特異敲除的靶位點序列,且本發明中所用的foki內切核酸酶為專性異二聚化foki質粒,購自addgene公司,可避免同源二聚化導致的非特異切割。
本發明中設計的針對人bcr-abl基因的鋅指核酸酶(zfns)由鋅指蛋白(zfp)和foki酶構成
(1)鋅指蛋白(zfp)的左臂(zfp-l)、右臂(zfp-r)序列
zfp-l的dna序列為(如seqidno:3所示):
5’-gtcgacctggagcccggcgagaagccctacaagtgccccgagtgcggcaagagcttcagcgactgccgcgacctggcccgccaccagcgcacccacaccggcgagaagccctacaagtgccccgagtgcggcaagagcttcagcgaccccggcaacctggtgcgccaccagcgcacccacaccggcgagaagccctacaagtgccccgagtgcggcaagagcttcagcgaccccggcgccctggtgcgccaccagcgcacccacaccggcgagaagccctacaagtgccccgagtgcggcaagagcttcagcgactgccgcgacctggcccgccaccagcgcacccacaccggcaagaagaccagctgcggccgc-3’。
zfp-r的dna序列為(如seqidno:4所示):
5’-gtcgacctggagcccggcgagaagccctacaagtgccccgagtgcggcaagagcttcagccgcagcgacaacctggtgcgccaccagcgcacccacaccggcgagaagccctacaagtgccccgagtgcggcaagagcttcagcgactgccgcgacctggcccgccaccagcgcacccacaccggcgagaagccctacaagtgccccgagtgcggcaagagcttcagcgaccccggcaacctggtgcgccaccagcgcacccacaccggcgagaagccctacaagtgccccgagtgcggcaagagcttcagccgcagcgacaacctggtgcgccaccagcgcacccacaccggcaagaagaccagctgcggccgc-3’。
在上述zfp-l和zfp-r的dna序列中,5’端的下劃線部分為sali酶切位點。3’端的下劃線部分為noti酶切位點,下劃線前面的堿基t為防止移碼添加的堿基。
(2)foki酶
foki-l質粒(質粒編號37198)和foki-r質粒(質粒編號37199)均購自addgene。
二、質粒pad-track-cmv-zfn-l的構建
按照如下步驟操作:
(1)將上述zfp-l的編碼序列交由北京華大基因公司合成;用sali酶和noti酶(takara公司)對zfp-l和pad-track-cmv(重慶醫科大學臨床檢驗診斷學教育部重點實驗室保存)進行雙酶切,37℃,1h;
(2)隨后將酶切產物分別進行純化回收,具體步驟按照天根生化科技有限公司普通dna產物純化試劑盒(貨號dp204);
(3)將酶切回收產物用t4連接酶(takara公司)進行連接,16℃,16h;
(4)連接產物轉化入dh5α感受態后在kana抗性的平板上進行分區劃線,隨后在37℃孵箱中培養12h;
(5)在上述kana平板上挑取8-10個單克隆菌落進行擴大培養;
(6)在擴增得到的菌液中提取質粒,具體步驟按照天根生化科技有限公司質粒小提試劑盒(貨號dp103);
(7)提取得到的質粒進行測序驗證,其中得到的測序結果正確的質粒就為pad-track-cmv-zfp-l;
(8)以foki-l質粒為模板,pcr擴增foki-l質粒,以表1所列的引物進行目的片段pcr擴增反應。反應體系為25μl,12.5μlpremixtaq、1μl10μm引物foki-senceprimer、1μl25μm引物foki-antisenceprimer、100ng質粒dna,加超純水至25μl。pcr擴增程序:94℃5min;94℃30s,退火溫度58℃,72℃30s,循環29次,最后72℃延伸5min;pcr擴增產物用2%瓊脂糖凝膠電泳檢測;
表1擴增foki質粒dna目的片段引物表
注:foki-senceprimer中gcggccgc為noti酶切位點,下劃線為linker序列,5’端的aaggaaaaaa為保護堿基。foki-antisenceprimer中ctcgag為xhoi酶切位點,5’端的ccg為保護堿基。
(9)隨后將pcr產物進行純化回收,具體步驟按照天根生化科技有限公司普通dna產物純化試劑盒(貨號dp204);
(10)用xhoi酶和noti酶(takara公司)對foki-l和pad-track-cmv-zfp-l質粒進行雙酶切,37℃,1h;
(11)隨后將酶切產物分別進行純化回收,具體步驟按照天根生化科技有限公司普通dna產物純化試劑盒(貨號dp204);
(12)將它們的酶切回收產物用t4連接酶(takara公司)進行連接,16℃,16h;
(13)連接產物轉化入dh5α感受態后在kana抗性的平板上進行分區劃線,隨后在37℃孵箱中培養12h;
(15)在上述kana平板上挑取8-10個單克隆菌落進行擴大培養;
(16)在擴增得到的菌液中提取質粒,具體步驟按照天根生化科技有限公司質粒小提試劑盒(貨號dp103);
(17)提取得到的質粒進行測序驗證,其中得到的測序結果正確的質粒就為pad-track-cmv-zfn-l。
三、質粒pad-track-cmv-zfn-r質粒的構建
過程與上述“質粒pad-track-cmv-zfn-l的構建”過程類似,不同處是將zfp-r合成后構建pad-track-cmv-zfp-r質粒,再與foki-r質粒相連接構建pad-track-cmv-zfn-r質粒。(擴增foki-r質粒所用的pcr引物\程序和產物長度同表1)。
四、同源模板donor質粒的構建
以k562細胞cdna為模板,pcr擴增donor(左臂bcr217~758,長542bp;右臂:bcr759~1523bp,長765bp),左臂克隆入kpni和noti酶切位點,右臂克隆入noti和xbai酶切位點,將左、右臂同時克隆入質粒pad-track-cmv中。
按照如下步驟操作:
(1)以k562細胞(上海細胞所提供)cdna為模板,pcr擴增donor左臂(donor-l),以表2所列的引物進行目的片段pcr擴增反應。反應體系為25μl,12.5μlpremixtaq、1μl10μm引物donor-l-f、1μl25μm引物donor-l-r、100ngcdna,加超純水至25μl。pcr擴增程序:94℃5min;94℃30s,退火溫度56℃,72℃30s,循環29次,最后72℃延伸5min,pcr擴增產物用2%瓊脂糖凝膠電泳檢測;
表2擴增donor左臂的引物
注:以上2條引物的下劃線處堿基分別為kpni和noti酶切位點
(2)用kpni酶和noti酶(takara公司)對donor-l的pcr產物和pad-track-cmv進行雙酶切,37℃,1h;
(3)隨后將酶切產物分別進行純化回收,具體步驟按照天根生化科技有限公司普通dna產物純化試劑盒(貨號dp204);
(4)將它們的酶切回收產物用t4連接酶(takara公司)進行連接,16℃,16h;
(5)連接產物轉化入dh5α感受態后在kana抗性的平板上進行分區劃線,隨后在37℃孵箱中培養12h;
(6)在上述kana平板上挑取8-10個單克隆菌落進行擴大培養;
(7)在擴增得到的菌液中提取質粒,具體步驟按照天根生化科技有限公司質粒小提試劑盒(貨號dp103);
(8)提取得到的質粒進行測序驗證,其中得到的測序結果正確的質粒就為pad-track-cmv-donor-l;
(9)再以k562細胞cdna為模板,pcr擴增donor右臂(donor-r),以表3所列的引物進行目的片段pcr擴增反應。反應體系為25μl,12.5μlpremixtaq、1μl10μm引物donor-r-f、1μl25μm引物donor-r-r、100ngcdna,加超純水至25μl。pcr擴增程序:94℃5min;94℃30s,退火溫度56℃(見表3),72℃30s,循環29次,最后72℃延伸5min,pcr擴增產物用2%瓊脂糖凝膠電泳檢測;
表3擴增donor右臂的引物
注:以上2條引物的下劃線部分堿基分別為noti和xbai酶切位點
(10)隨后將pcr產物進行純化回收,具體步驟按照天根生化科技有限公司普通dna產物純化試劑盒(貨號dp204);
(11)用noti酶和xbai酶(takara公司)對donor-r的pcr產物和pad-track-cmv-donor-l質粒進行雙酶切,37℃,1h;
(12)隨后將酶切產物分別進行純化回收,具體步驟按照天根生化科技有限公司普通dna產物純化試劑盒(貨號dp204);
(13)將它們的酶切回收產物用t4連接酶(takara公司)進行連接,16℃,16h;
(14)連接產物轉化入dh5α感受態后在kana抗性的平板上進行分區劃線,隨后在37℃孵箱中培養12h;
(15)在上述kana平板上挑取8-10個單克隆菌落進行擴大培養;
(16)在擴增得到的菌液中提取質粒,具體步驟按照天根生化科技有限公司質粒小提試劑盒(貨號dp103);
(17)提取得到的質粒進行測序驗證,其中得到的測序結果正確的質粒就為pad-track-cmv-donor。
實施例2、鋅指核酸酶定點切割bcr-abl基因并插入noti酶切位點促發同源定向修復
1、凍存細胞的復蘇與培養
從液氮中取出裝有k562細胞的凍存小管,立即投入37℃的溫水中快速晃動,直至凍存液完全融解;在2min內完成復溫;將細胞懸液移入無菌的離心管,加入5ml培養液,輕輕吹勻;將細胞懸液1000r/min離心5min,棄上清;向含有細胞沉淀的離心管加入1ml完全培養基。
2、細胞培養條件及傳代培養:
細胞培養條件
培養基組成成分:1640(gibcolot:1737734)
10%fbs(bilot:1616756)
傳代培養:
(1)k562細胞密度達約80%左右;
(2)用移液器反復吹打數次,至細胞全部均勻重懸在培養基中,將其轉移到離心管中;500rpm離心5min;
(3)棄上清,加入2ml培養基重懸細胞,分兩份加到有培養基的培養瓶中;
(4)晃動培養瓶使細胞分布均勻后,至37℃、5%co2培養箱中培養。
3、核轉染方法
使用lonza公司提供的電轉試劑celllinenucleofectorkitv及核轉染儀進行轉染實驗,具體操作按照試劑說明書進行。
4、細胞基因組dna提取:按照天根生化科技有限血液/組織/細胞基因組提取試劑盒(貨號:dp304)操作步驟說明,提取步驟3得到的培養48小時的細胞基因組dna。
5、目的片段pcr擴增反應和pcr產物測序驗證
以上述步驟4得到的細胞基因組dna為模板,以下表4所列的引物進行目的片段pcr擴增反應。反應體系為25μl,12.5μlpremixtaq、1μl10μm引物senceprimer、1μl25μm引物anti-senceprimer、100ng基因組dna,加超純水至25μl。pcr擴增程序:94℃5min;94℃30s,退火溫度56℃,72℃30s,循環29次,最后72℃延伸5min。pcr擴增產物用2%瓊脂糖凝膠電泳檢測。
表4擴增bcr-abl基因組dna目的片段引物表
將pcr產物直接進行測序,將測序所得的序列結果與bcr-abl基因中的靶位點序列進行同源性比較分析,以鑒定轉染zfn質粒k562細胞基因組dna中靶位點是否被定點插入noti酶切位點。測序結果峰圖如圖1所示,k562細胞基因組dna的靶位點被定點插入noti酶切位點。
6、westernblot檢測相關蛋白變化
提取細胞總蛋白與濃度測定:
(1)收集前述步驟3得到的培養48小時的細胞到10ml離心管里,1000rpm離心10min,棄盡上清液;
(2)再加入2mlpbs于離心管中將細胞懸液,吹打混勻,1000rpm離心10min,棄盡上清液;
(3)加入1mlpbs重新混勻,將細胞懸液轉至新的1.5mlep管中;
(4)配制裂解液:pmsf:naf:navo4:ripa=1:1:1:100,每個dish加60μl裂解液。冰上裂解30分鐘,每10分鐘振蕩一次(5-10s);
(5)將ep管放入4℃離心機,12000rpm離心40分鐘,上清液轉移至另一個新的ep管中,記錄體積,加上清液1/4體積的5×蛋白上樣buffer,沸水煮5分鐘,-80℃超低溫冰箱保存;
(6)bca試劑盒測蛋白濃度:配制bca標準品工作液,終濃度0.5mg/ml。將標準品和待測品加入96孔板,按照說明書向每孔加工作液,振蕩混勻后37℃水浴箱孵育35分鐘,測定560nm處吸光度值,繪制標準曲線并計算蛋白濃度及上樣量(見表5)。
表5蛋白濃度測定
westernblot:
(1)配制10%分離膠加入安裝好的電泳儀中,1ml無水乙醇壓線,37℃烤箱靜置30min取出,棄去無水乙醇,配制5%濃縮膠,根據實驗需求加入適量孔梳,再將配好的濃縮膠加入,37℃烤箱靜置20min。待濃縮膠完全凝固后去掉膠條放入電泳槽中,加入內槽液(1×sds)后拔掉梳子,加入外槽液后排氣泡。每孔加入30μg蛋白樣品,兩步法電泳:80v電泳30分鐘,120v電泳90分鐘;
(2)提前預冷濕轉液并將濾紙浸泡在濕轉液中,按目的條帶分子量切膠浸潤濕轉液待用。剪與切膠大小相等的膜,甲醇活化1min,雙蒸水2min轉入濕轉液中。轉膜:按照三層濾紙、膜、膠和三層濾紙的順序放入轉膜儀,210ma恒流,小分子蛋白按照分子量大小相同時間轉膜,大分子量蛋白按照分子量×0.85時間轉膜;
(3)配制5%的封閉奶粉待轉膜結束時將pvdf膜放入進行封閉,4℃冰箱4h;
(4)tbst去除膜上封閉液放置于蠟板上,加入一抗(1:1000稀釋)后密封,4℃冰箱充分反應14-18h;
(5)取出pvdf膜,tbst洗3次/5min,加入二抗(1:2000稀釋),4℃90分鐘后再用tbst洗2次/5min,tbs洗5min;
(6)配制發光液:a液:b液=1:1,加在膜上避光于發光儀內顯像。
westernblot檢測相關蛋白結果如圖2所示,與空白組,空載組以及各種質粒單獨作用組對比zfn-l/r和donor共同作用于cml細胞后能使p-bcr-abl蛋白明顯下調,且bcr-abl蛋白下游的效應分子p-stat5和p-erk都明顯下調;與空白組,空載組以及各種質粒單獨作用組對比zfn-l/r和donor共同作用于cml細胞后能激活parp和caspase-3蛋白,說明其能促進cml細胞凋亡。
7、克隆形成實驗
(1)分別用空載質粒gfp和zfn-l/r+donor質粒核轉染k562細胞,并且設對照組(無質粒轉染),每組處理設置5個復孔,每孔鋪300個細胞于24孔板,調整每孔終培養基至750μl;
(2)在每孔中加入750μl2.7%甲基纖維素,混勻;
(3)37℃、5%co2恒溫細胞孵箱內培養7-10天觀察結果。結果如圖3、4所示:zfn-l/r和donor共同作用于k562細胞后能明顯抑制k562細胞的克隆大小和克隆數量,即抑制k562細胞的克隆形成能力。
序列表
<110>重慶醫科大學
<120>采用鋅指核酸酶技術破壞人bcr-abl融合基因以抑制cml細胞增殖和促使其凋亡
<130>1
<160>12
<210>1
<211>6021
<212>dna
<213>人工序列
<223>人bcr-abl基因
<400>1
atggtggacccggtgggcttcgcggaggcgtggaaggcgcagttcccggactcagagccc60
ccgcgcatggagctgcgctcagtgggcgacatcgagcaggagctggagcgctgcaaggcc120
tccattcggcgcctggagcaggaggtgaaccaggagcgcttccgcatgatctacctgcag180
acgttgctggccaaggaaaagaagagctatgaccggcagcgatggggcttccggcgcgcg240
gcgcaggcccccgacggcgcctccgagccccgagcgtccgcgtcgcgcccgcagccagcg300
cccgccgacggagccgacccgccgcccgccgaggagcccgaggcccggcccgacggcgag360
ggttctccgggtaaggccaggcccgggaccgcccgcaggcccggggcagccgcgtcgggg420
gaacgggacgaccggggaccccccgccagcgtggcggcgctcaggtccaacttcgagcgg480
atccgcaagggccatggccagcccggggcggacgccgagaagcccttctacgtgaacgtc540
gagtttcaccacgagcgcggcctggtgaaggtcaacgacaaagaggtgtcggaccgcatc600
agctccctgggcagccaggccatgcagatggagcgcaaaaagtcccagcacggcgcgggc660
tcgagcgtgggggatgcatccaggcccccttaccggggacgctcctcggagagcagctgc720
ggcgtcgacggcgactacgaggacgccgagttgaacccccgcttcctgaaggacaacctg780
atcgacgccaatggcggtagcaggcccccttggccgcccctggagtaccagccctaccag840
agcatctacgtcgggggcatgatggaaggggagggcaagggcccgctcctgcgcagccag900
agcacctctgagcaggagaagcgccttacctggccccgcaggtcctactccccccggagt960
tttgaggattgcggaggcggctataccccggactgcagctccaatgagaacctcacctcc1020
agcgaggaggacttctcctctggccagtccagccgcgtgtccccaagccccaccacctac1080
cgcatgttccgggacaaaagccgctctccctcgcagaactcgcaacagtccttcgacagc1140
agcagtccccccacgccgcagtgccataagcggcaccggcactgcccggttgtcgtgtcc1200
gaggccaccatcgtgggcgtccgcaagaccgggcagatctggcccaacgatggcgagggc1260
gccttccatggagacgcagatggctcgttcggaacaccacctggatacggctgcgctgca1320
gaccgggcagaggagcagcgccggcaccaagatgggctgccctacattgatgactcgccc1380
tcctcatcgccccacctcagcagcaagggcaggggcagccgggatgcgctggtctcggga1440
gccctggagtccactaaagcgagtgagctggacttggaaaagggcttggagatgagaaaa1500
tgggtcctgtcgggaatcctggctagcgaggagacttacctgagccacctggaggcactg1560
ctgctgcccatgaagcctttgaaagccgctgccaccacctctcagccggtgctgacgagt1620
cagcagatcgagaccatcttcttcaaagtgcctgagctctacgagatccacaaggagttc1680
tatgatgggctcttcccccgcgtgcagcagtggagccaccagcagcgggtgggcgacctc1740
ttccagaagctggccagccagctgggtgtgtaccgggccttcgtggacaactacggagtt1800
gccatggaaatggctgagaagtgctgtcaggccaatgctcagtttgcagaaatctccgag1860
aacctgagagccagaagcaacaaagatgccaaggatccaacgaccaagaactctctggaa1920
actctgctctacaagcctgtggaccgtgtgacgaggagcacgctggtcctccatgacttg1980
ctgaagcacactcctgccagccaccctgaccaccccttgctgcaggacgccctccgcatc2040
tcacagaacttcctgtccagcatcaatgaggagatcacaccccgacggcagtccatgacg2100
gtgaagaagggagagcaccggcagctgctgaaggacagcttcatggtggagctggtggag2160
ggggcccgcaagctgcgccacgtcttcctgttcaccgagctgcttctctgcaccaagctc2220
aagaagcagagcggaggcaaaacgcagcagtatgactgcaaatggtacattccgctcacg2280
gatctcagcttccagatggtggatgaactggaggcagtgcccaacatccccctggtgccc2340
gatgaggagctggacgctttgaagatcaagatctcccagatcaagagtgacatccagaga2400
gagaagagggcgaacaagggcagcaaggctacggagaggctgaagaagaagctgtcggag2460
caggagtcactgctgctgcttatgtctcccagcatggccttcagggtgcacagccgcaac2520
ggcaagagttacacgttcctgatctcctctgactatgagcgtgcagagtggagggagaac2580
atccgggagcagcagaagaagtgtttcagaagcttctccctgacatccgtggagctgcag2640
atgctgaccaactcgtgtgtgaaactccagactgtccacagcattccgctgaccatcaat2700
aaggaagaagcccttcagcggccagtagcatctgactttgagcctcagggtctgagtgaa2760
gccgctcgttggaactccaaggaaaaccttctcgctggacccagtgaaaatgaccccaac2820
cttttcgttgcactgtatgattttgtggccagtggagataacactctaagcataactaaa2880
ggtgaaaagctccgggtcttaggctataatcacaatggggaatggtgtgaagcccaaacc2940
aaaaatggccaaggctgggtcccaagcaactacatcacgccagtcaacagtctggagaaa3000
cactcctggtaccatgggcctgtgtcccgcaatgccgctgagtatctgctgagcagcggg3060
atcaatggcagcttcttggtgcgtgagagtgagagcagtcctggccagaggtccatctcg3120
ctgagatacgaagggagggtgtaccattacaggatcaacactgcttctgatggcaagctc3180
tacgtctcctccgagagccgcttcaacaccctggccgagttggttcatcatcattcaacg3240
gtggccgacgggctcatcaccacgctccattatccagccccaaagcgcaacaagcccact3300
gtctatggtgtgtcccccaactacgacaagtgggagatggaacgcacggacatcaccatg3360
aagcacaagctgggcgggggccagtacggggaggtgtacgagggcgtgtggaagaaatac3420
agcctgacggtggccgtgaagaccttgaaggaggacaccatggaggtggaagagttcttg3480
aaagaagctgcagtcatgaaagagatcaaacaccctaacctggtgcagctccttggggtc3540
tgcacccgggagcccccgttctatatcatcactgagttcatgacctacgggaacctcctg3600
gactacctgagggagtgcaaccggcaggaggtgaacgccgtggtgctgctgtacatggcc3660
actcagatctcgtcagccatggagtacctggagaagaaaaacttcatccacagagatctt3720
gctgcccgaaactgcctggtaggggagaaccacttggtgaaggtagctgattttggcctg3780
agcaggttgatgacaggggacacctacacagcccatgctggagccaagttccccatcaaa3840
tggactgcacccgagagcctggcctacaacaagttctccatcaagtccgacgtctgggca3900
tttggagtattgctttgggaaattgctacctatggcatgtccccttacccgggaattgac3960
ctgtcccaggtgtatgagctgctagagaaggactaccgcatggagcgcccagaaggctgc4020
ccagagaaggtctatgaactcatgcgagcatgttggcagtggaatccctctgaccggccc4080
tcctttgctgaaatccaccaagcctttgaaacaatgttccaggaatccagtatctcagac4140
gaagtggaaaaggagctggggaaacaaggcgtccgtggggctgtgagtaccttgctgcag4200
gccccagagctgcccaccaagacgaggacctccaggagagctgcagagcacagagacacc4260
actgacgtgcctgagatgcctcactccaagggccagggagagagcgatcctctggaccat4320
gagcctgccgtgtctccattgctccctcgaaaagagcgaggtcccccggagggcggcctg4380
aatgaagatgagcgccttctccccaaagacaaaaagaccaacttgttcagcgccttgatc4440
aagaagaagaagaagacagccccaacccctcccaaacgcagcagctccttccgggagatg4500
gacggccagccggagcgcagaggggccggcgaggaagagggccgagacatcagcaacggg4560
gcactggctttcacccccttggacacagctgacccagccaagtccccaaagcccagcaat4620
ggggctggggtccccaatggagccctccgggagtccgggggctcaggcttccggtctccc4680
cacctgtggaagaagtccagcacgctgaccagcagccgcctagccaccggcgaggaggag4740
ggcggtggcagctccagcaagcgcttcctgcgctcttgctccgcctcctgcgttccccat4800
ggggccaaggacacggagtggaggtcagtcacgctgcctcgggacttgcagtccacggga4860
agacagtttgactcgtccacatttggagggcacaaaagtgagaagccggctctgcctcgg4920
aagagggcaggggagaacaggtctgaccaggtgacccgaggcacagtaacgcctcccccc4980
aggctggtgaaaaagaatgaggaagctgctgatgaggtcttcaaagacatcatggagtcc5040
agcccgggctccagcccgcccaacctgactccaaaacccctccggcggcaggtcaccgtg5100
gcccctgcctcgggcctcccccacaaggaagaagctggaaagggcagtgccttagggacc5160
cctgctgcagctgagccagtgacccccaccagcaaagcaggctcaggtgcaccagggggc5220
accagcaagggccccgccgaggagtccagagtgaggaggcacaagcactcctctgagtcg5280
ccagggagggacaaggggaaattgtccaggctcaaacctgccccgccgcccccaccagca5340
gcctctgcagggaaggctggaggaaagccctcgcagagcccgagccaggaggcggccggg5400
gaggcagtcctgggcgcaaagacaaaagccacgagtctggttgatgctgtgaacagtgac5460
gctgccaagcccagccagccgggagagggcctcaaaaagcccgtgctcccggccactcca5520
aagccacagtccgccaagccgtcggggacccccatcagcccagcccccgttccctccacg5580
ttgccatcagcatcctcggccctggcaggggaccagccgtcttccaccgccttcatccct5640
ctcatatcaacccgagtgtctcttcggaaaacccgccagcctccagagcggatcgccagc5700
ggcgccatcaccaagggcgtggtcctggacagcaccgaggcgctgtgcctcgccatctct5760
aggaactccgagcagatggccagccacagcgcagtgctggaggccggcaaaaacctctac5820
acgttctgcgtgagctatgtggattccatccagcaaatgaggaacaagtttgccttccga5880
gaggccatcaacaaactggagaataatctccgggagcttcagatctgcccggcgacagca5940
ggcagtggtccggcggccactcaggacttcagcaagctcctcagttcggtgaaggaaatc6000
agtgacatagtgcagaggtag6021
<210>2
<211>30
<212>dna
<213>人工序列
<223>人bcr-abl基因的鋅指核酸酶作用的特異靶位點
<400>2
ggcgtcgacggcgactacgaggacgccgag30
<210>3
<211>375
<212>dna
<213>人工序列
<223>zfp-l
<400>3
gtcgacctggagcccggcgagaagccctacaagtgccccgagtgcggcaagagcttcagc60
gactgccgcgacctggcccgccaccagcgcacccacaccggcgagaagccctacaagtgc120
cccgagtgcggcaagagcttcagcgaccccggcaacctggtgcgccaccagcgcacccac180
accggcgagaagccctacaagtgccccgagtgcggcaagagcttcagcgaccccggcgcc240
ctggtgcgccaccagcgcacccacaccggcgagaagccctacaagtgccccgagtgcggc300
aagagcttcagcgactgccgcgacctggcccgccaccagcgcacccacaccggcaagaag360
accagctgcggccgc375
<210>4
<211>375
<212>dna
<213>人工序列
<223>zfp-r
<400>4
gtcgacctggagcccggcgagaagccctacaagtgccccgagtgcggcaagagcttcagc60
cgcagcgacaacctggtgcgccaccagcgcacccacaccggcgagaagccctacaagtgc120
cccgagtgcggcaagagcttcagcgactgccgcgacctggcccgccaccagcgcacccac180
accggcgagaagccctacaagtgccccgagtgcggcaagagcttcagcgaccccggcaac240
ctggtgcgccaccagcgcacccacaccggcgagaagccctacaagtgccccgagtgcggc300
aagagcttcagccgcagcgacaacctggtgcgccaccagcgcacccacaccggcaagaag360
accagctgcggccgc375
<210>5
<211>78
<212>dna
<213>人工序列
<223>foki-senceprimer
<400>5
aaggaaaaaagcggccgcggcggcggcggcagcggcggcggcggcagcgccctggtgaag60
agcgagctggaggagaag78
<210>6
<211>46
<212>dna
<213>人工序列
<223>foki-antisenceprimer
<400>6
ccgctcgagtcagaagttgatctcgccgttgttgaacttgcgccgc46
<210>7
<211>29
<212>dna
<213>人工序列
<223>donor-l-f
<400>7
cggggtacccagcgatggggcttccggcg29
<210>8
<211>45
<212>dna
<213>人工序列
<223>donor-l-r
<400>8
aaggaaaaaagcggccgcgggttcaactcggcgtcctcgtagtcg45
<210>9
<211>44
<212>dna
<213>人工序列
<223>donor-r-f
<400>9
aaggaaaaaagcggccgcccgcttcctgaaggacaacctgatcg44
<210>10
<211>36
<212>dna
<213>人工序列
<223>donor-r-r
<400>10
gctctagagccaggattcccgacaggacccattttc36
<210>11
<211>18
<212>dna
<213>人工序列
<223>senceprimer
<400>11
gacgccgagaagcccttc18
<210>12
<211>20
<212>dna
<213>人工序列
<223>anti-senceprimer
<400>12
aatcctcaaaactccggggg20
- 還沒有人留言評論。精彩留言會獲得點贊!