一種培育水稻普通核不育系的方法與流程
本發明涉及水稻生物技術育種領域,具體涉及一種培育水稻普通核不育系的方法。
背景技術:
普通核不育受一對隱性核基因控制,一般不受外部環境因子的影響,不管環境條件怎樣變化,總是表現為不育。在生產應用中,普通核不育具有以下優點:1)育性正常的品種都是它的恢復系,恢復譜及其廣泛,配組自由使得選育優良組合的幾率大增;2)能緊跟常規稻的選育步伐,開辟秈粳亞種間雜種優勢新領域,使水稻產量在現有雜交水稻基礎上實現更高產目標。因此,袁隆平院士將普通核不育系應用于雜交水稻制種的技術稱為第三代的雜交水稻。目前普通核不育系的繁殖難題已經攻克,湖南雜交水稻研究中心申請的ZL201210426678.7號和ZL201210426939.5號中國專利文獻中記載了利用基因工程手段構建工程保持系來大規模繁殖普通核不育系的方法,為普通核不育系應用于實際生產奠定了基礎。但利用普通核不育系配制高產、高抗和質優的超級稻組合還存在難題,主要的難題就是優良普通核不育系的選育方面還只能通過傳統的物理化學方法誘變優良的保持系和恢復系等可育材料獲得或者將這些優良的可育材料與不育系通過多代回交選育獲得。物理化學誘變方法具有隨機性,效率低下,勞動強度大;回交選育需要經過多代回交進行背景篩選,時間跨度大,即使經過多代回交也很難恢復原有的遺傳背景,導致原有的一些優良性狀丟失和利用工程保持系所繁育的普通核不育系株型不一致。所以,這兩種傳統方法都有很大的局限性,延緩了第三代雜交水稻技術在生產制種中的應用。要突破傳統方法的局限,必須利用基因工程手段。近年來,基因組編輯技術取得了突破性進展,尤其是以TALEN(Transcription activator-like effector nucleases)和CRISPR/Cas9(Clustered regularly interspaced short palindromic repeats and CRISPR associated)這兩種高效的基因編輯技術的成熟和完善,已廣泛應用于各種生物的基因組的定點突變。比如CN201410496037.8號和CN201510009526.0號專利報道了利用CRISPR/Cas9基因編輯系統定點突變控制水稻光溫敏不育性狀的tms5基因和P-TMS12-1基因實現快速培育兩系不育系目標的方法。目前尚沒有采用CRISPR/Cas9基因工程手段快速培育普通核不育系的方法。
技術實現要素:
本發明要解決的技術問題是克服現有技術的不足,提供一種利用基因組編輯技術定點突變普通核不育基因獲得普通核不育系的方法,通過徹底失活普通核不育基因,實現快速培育普通核不育系的目標,育種周期短、成本低且實用性強,將為推進第三代雜交水稻的廣泛應用具有重要意義。
為此,本發明提供了一種培育水稻普通核不育系的方法,包括以下步驟:
S1、利用CRISPR/Cas9系統,根據水稻普通核不育基因設計靶位點序列;
S2、構建含所述靶位點序列片段的pCRISPR/Cas9重組載體;
S3、將所獲得的pCRISPR/Cas9重組載體導入保持系的胚性愈傷組織中得到轉基因苗;
S4、篩選所述轉基因苗中的轉基因陽性植株;
S5、篩選所述轉基因陽性植株中的突變植株;
S6、將所述突變植株進行繁種,于后代植株中分離不含轉基因成分的普通核不育系。
上述的方法,優選的,所述步驟S1中設計的靶位點序列的一條鏈具有以下結構中的一種:5’-(N)n-NGG-3’、5’-(N)n-NAG-3’、5’-(N)n-NGA-3’,所述N表示A,T,C和G中的任意一個,14≤n≤30。
上述的方法,優選的,所述普通核不育基因為msp1、pair1、pair2、zep1、mel1、pss1、tdr、udt1、gamyb4、ptc1、api5、wda1、cyp704B2、dpw、mads3、osc6、rip1、csa和aid1中的一種。
上述的方法,優選的,所述普通核不育基因為ptc1,所述靶位點序列包括PTC1-Target1和PTC1-Target2,所述PTC1-Target1的DNA序列為SEQ ID NO.1所示的序列,所述PTC1-Target1的DNA序列為SEQ ID NO.2所示的序列。
上述的方法,優選的,所述步驟S2具體包括以下步驟:
S2-1、根據靶位點序列以及酶切位點信息,設計帶粘性末端的接頭引物;
S2-2、酶切原始載體;
S2-3、將所述帶粘性末端的接頭引物退火后連接到酶切過的原始載體上,得到重組的gRNA表達盒;
S2-4、將所述重組gRNA表達盒進行PCR擴增得到擴增產物;
S2-5、酶切擴增產物,將酶切后的擴增產物連接到酶切過的pCRISPR/Cas9載體上得到重組載體。
上述的方法,優選的,所述步驟S2-1中所述接頭引物包括PTC1-Target1-F、PTC1-Target1-R、PTC1-Target2-F和PTC1-Target2-R;所述PTC1-Target1-F的DNA序列為SEQ ID NO.3所示的序列,所述PTC1-Target1-R的DNA序列為SEQ ID NO.4所示的序列,所述PTC1-Target2-F的DNA序列為SEQ ID NO.5所示的序列;所述PTC1-Target2-R的DNA序列為SEQ ID NO.6所示的序列。
上述的方法,優選的,所述原始載體為pU3-gRNA或pU6a-gRNA。
上述的方法,優選的,所述步驟S2-2中采用BsaI酶切所述原始載體;所述步驟S2-3中,所述帶粘性末端的接頭引物位于所述表達載體的兩個Bsa I酶切位點之間,形成重組的gRNA表達盒;所述步驟S2-5中,采用BsaI酶切所述擴增產物。
上述的方法,優選的,所述步驟S5步驟具體為:提取所述轉基因陽性植株的DNA,進行PCR擴增得到擴增產物;對所述擴增產物進行測序,選擇任一靶位點發生功能缺失突變的T0代純合突變體作為突變植株。
上述的方法,優選的,所述步驟S6具體為:將突變植株與保持系回交,于后代植株中分離不含轉基因成分的普通核不育系。
與現有技術相比,本發明的優點在于:
(1)本發明提供了一種基于CRISPR/Cas9系統靶向突變普通核不育基因獲得普通核不育系的方法,培育出的普通核不育系不含轉基因成分,與物理化學誘變方法和回交選育方法獲得的普通核不育系沒有本質區別,規避了轉基因帶來的潛在風險。
(2)本發明提供了一種水稻普通核不育系的方法,實現了快速培育普通核不育系,比物理化學誘變方法效率高,基因組損傷小;比傳統的回交選育方法節省時間(傳統的回交選育方法一般需要4~6年,而本發明提供的水稻普通核不育系的方法僅需兩年),并且能克服傳統的回交選育導致原有的一些優良性狀丟失和利用工程保持系所繁育的普通核不育系株型不一致的缺點。
附圖說明
為使本發明實施例的目的、技術方案和優點更加清楚,下面將結合本發明實施例中的附圖,對本發明實施例中的技術方案進行清楚、完整的描述。
圖1為本發明培育普通核不育系的流程圖。
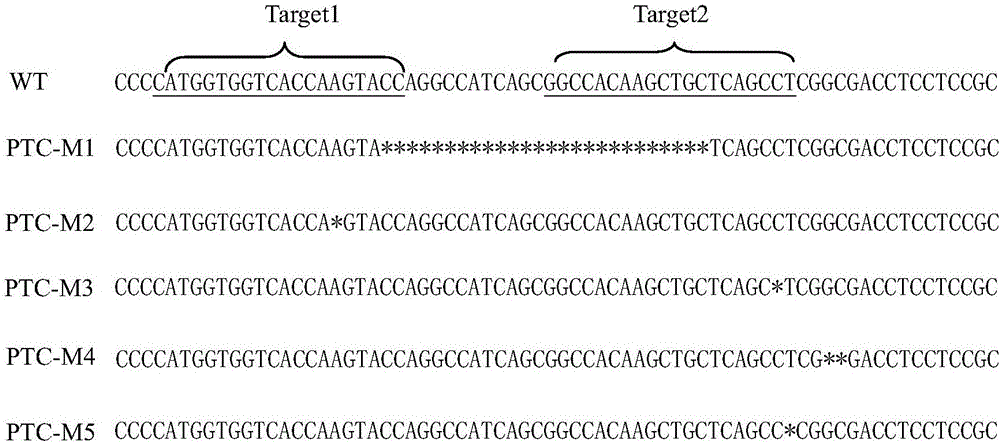
圖2為五個陽性植株的突變類型,其中WT為野生型832B;PTC-M1至PTC-M5為5個陽性植株。
圖3為PT6-F/PT6-R引物對BC1F2群體中的單株進行PTC1基因分型的部分結果。
圖4為雜合單株與普通核不育系單株穎花形態對比。其中雜合單株花藥為淺黃色,內含正常的花粉,表現出可育性狀;普通核不育系花藥呈白色,內無正常的花粉,表現出不育性狀。
圖5為PCR檢測潮霉素基因結果,雜合株含有潮霉素基因,因此含有轉基因成份;普通核不育系不含潮霉素基因,因此不含轉基因成份。
具體實施方式
以下結合說明書附圖和具體優選的實施例對本發明作進一步描述,但并不因此而限制本發明的保護范圍。
實施例
以下實施例中所采用的材料和儀器均為市售。
實施例1:
一種本發明的快速培育普通核不育系的方法,其應用的具體對象即為本實施例所述的由CRISPR/Cas9系統定點突變保持系832B中的PTC1基因(SEQ ID NO.7為PTC1基因的cDNA序列、SEQ ID NO.8為PTC1基因的qDNA序列)獲得普通核不育系,具體包括以下步驟(工藝流程可參見圖1):
(1)根據PTC1基因的外顯子序列設計雙靶位點:
PTC1-Target1(SEQ ID NO.1):CATGGTGGTCACCAAGTACC;
PTC1-Target2(SEQ ID NO.2):GGCCACAAGCTGCTCAGCCT。
兩個位點分別位于PTC1基因編碼區的886bp-905bp和917bp-936bp。
PTC1-Target1具有5’-(N)n-NGG-3’的結構;PTC1-Target2具有5’-(N)n-NGA-3’結構。
(2)含有PTC1-Target1、PTC1-Target2雙靶點的CRISPR/Cas9重組載體(pCRISPR/Cas9-PTC1)的構建:
2.1根據靶位點序列合成兩個靶位點互補配對的核苷酸單鏈:
PTC1-Target1-F(SEQ ID NO.3):ggcaCATGGTGGTCACCAAGTACC;
PTC1-Target1-R(SEQ ID NO.4):aaacGGTACTTGGTGACCACCATG;
PTC1-Target2-F(SEQ ID NO.5):gccGGCCACAAGCTGCTCAGCCT;
PTC1-Target2-R(SEQ ID NO.6):aaacAGGCTGAGCAGCTTGTGGC。
2.2構建CRISPR/Cas9-PTC1重組載體:
2.2.1將步驟2.1中互補配對的兩條核苷酸單鏈等量混合后,經過90℃變性3min和20℃退火5min后形成具有粘性末端的雙鏈接頭。
2.2.2酶切環狀pU3-gRNA、pU6a-gRNA載體,獲得線性化pU3-gRNA、pU6a-gRNA載體:在20μL反應體系中用10U BsaI酶切pU3-gRNA、pU6a-gRNA載體20min,在70℃下放置5min使酶失活,得到線性化的pU3-gRNA、pU6agRNA載體。
2.2.3將步驟2.2.1中得到的具有粘性末端的雙鏈接頭與線性化的pU3-gRNA、pU6a-gRNA載體連接。連接步驟為:取1μl 10x T4 DNA ligase buffer,0.5μl pU3-gRNA/pU6a-gRNA載體(12ng),1μl PTC1-Target1/PTC1-Target1,1μl T4 DNA ligase,最后加ddH2O至總體積10μl,在20℃~25℃下連接30min,得到重組的guide-RNA表達盒中間載體:pU3-PTC1-Target1-gRNA、pU6a-PTC1-Target2-gRNA。
2.2.4 PCR擴增表達盒:用引物U3-F和U3-R擴增步驟2.2.3中得到的pU3-PTC1-Target1-gRNA;用引物U6a-F和U6a-R擴增步驟2.2.3中得到的pU6a-PTC1-Target2-gRNA。其中引物的序列分別為:
U3-F:TTCAGAGGTCTCTCTCGCACTGGAATCGGCAGCAAAGG;
U3-R:AGCGTGGGTCTCGTCAGGGTCCATCCACTCCAAGCTC;
U6a-F:TTCAGAGGTCTCTCTGACACTGGAATCGGCAGCAAAGG;
U6a-R:AGCGTGGGTCTCGACCGGGTCCATCCACTCCAAGCTC;
反應體系為:1μl pU3-PTC1-Target1-gRNA/pU6a-PTC1-Target2-gRNA載體,引物各1.5μl,10μl dNTP,25μl 2xbuffer,1μlKOD酶,加ddH2O至總體積50μL。
反應程序為:95℃預變性3min,35個循環:95℃變性10s,60℃退火15s,68℃延伸20s。
擴增獲得的PTC1-Target1-gRNA和PTC1-Target2-gRNA用PCR純化試劑盒純化。
2.2.5將PTC1-Target1-gRNA和PTC1-Target2-gRNA連接到pCRISPR/Cas9載體上構建成pCRISPR/Cas9-PTC1重組載體。具體構建方法:1μlPTC1-Target1-gRNA和1μl PTC1-Target2-gRNA,1μLpCRISPR/Cas9載體,1μL BsaI,1.5μL 10xSmart Buffer,1.5μL ATP(1mM),1μL T4 DNA ligase,最后加ddH2O至總體積15μL。
將上述反應體系在PCR上進行反應,反應程序為37℃5min、10℃5min、20℃5min。
(3)采用農桿菌介導的技術將pCRISPR/Cas9-PTC1重組載體整合到保持系832B的胚性愈傷組織并通過愈傷的分化獲得16株T0代轉基因植株。具體步驟如下:
3.1構建好的pCRISPR/Cas9-PTC1重組載體導入農桿菌種EHA105得到含有pCRISPR/Cas9-PTC1重組載體的EHA105;用832B的成熟種子在誘導培養基上誘導愈傷組織得到水稻愈傷組織。
3.2將含有pCRISPR/Cas9-PTC1重組載體的EHA105接種于YM瓊脂培養基28℃培養2天得到培養液。
3.3將收集到的培養液加入到含有100mol/L的乙酰丁香酮的NB液體培養基中,得到OD600為0.5的菌液。
3.4將步驟3.1獲得水稻愈傷組織浸泡在步驟3.3的菌液中30min,用無菌水沖洗3次,干燥后,將水稻愈傷組織轉移到NB培養基上28℃暗培養3天。然后轉移水稻愈傷組織至含有50mg/L潮霉素的培養基上,放置于28℃暗培養,每15天繼代一次,繼代2次。抗性篩選后,轉入到再生培養基,共分化出16株轉基因苗。經潮霉素PCR檢測,獲得5株轉基因陽性植株。
(4)突變位點的鑒定:
4.1提取5株陽性植株的DNA。
4.2根據兩個靶位點的位置設計檢測引物:
PT6-F:CCTCCGACATGATGCCCCGGTAGTCCAT和
PT6-R:GAGCTCCCCATGGTGGTCACCAAGTACCAG。
4.3使PT6-F在靶位點PTC1-Target1的上游,PT6-R在靶位點PTC1-Target2的下游。以5株陽性株DNA為模板,以PT6-F和PT6-R為引物進行PCR擴增得到PCR擴增產物。PCR擴增的反應體系為:5μl PCR Mix,PT6-F和PT6-R各0.2μl,1μl模板,ddH2O補足至10μl。反應程序為:95℃預變性3min,35個循環:94℃變性10s,60℃退火30s,72℃延伸30s。
4.4將PCR擴增產物送測序公司測序。測序結果參見圖2,*表示堿基缺失。從圖2中顯示5個陽性植株中有兩個為純合突變株系,T0代表現出不育性狀,分別命名為PTC-M1和PTC-M2。PTC-M1在兩個靶位點之間缺失26堿基,PTC-M2在PTC1-Target1靶位點處缺失1個堿基A;其他三個為雜合可育突變植株,分別命名為PTC-M3、PTC-M4、PTC-M5;PTC-M3在PTC1-Target2靶位點附近缺失了一個堿基C;PTC-M4在PTC1-Target2靶位點附近缺失了兩個堿基GC;PTC-M5在PTC1-Target2靶位點附近缺失了一個堿基T。
(5)在兩個為純合突變株系中選擇PTC-M1株系與832B回交1代獲得BC1F1后代;將BC1F1后代自交1代后獲得378株BC1F2后代。由于PTC-M1株系的PTC1基因缺失了26個堿基,因此用PT6-F和PT6-R引物擴增832B和PTC-M1株系的DNA時,兩者PCR產物條帶大小會相差26個堿基,而雜合單株的PCR產物則會出現大小相差26個堿基的兩條帶。故可以根據PT6-F和PT6-R引物擴增條帶的大小和數目對回交后代的PTC1基因進行基因分型,通過基因分型鑒定回交后代中的不育株、雜合株和可育株。
具體步驟為:
5.1以832B(PCR產物電泳條帶較大)和不育突變株PTC-M1(PCR產物電泳條帶較小)為對照,用PT6-F和PT6-R引物分別擴增378株BC1F2后代的DNA,擴增產物進行聚丙烯酰胺凝膠電泳,根據電泳條帶的大小和數目,鑒定出不育單株、雜合可育單株、純合可育單株。結果參見圖3:M為50bp marker;P1為832B帶型,為純合可育的野生型;P2為PTC-M1帶型,為純合不育的突變帶型,雙帶為可育雜合型。因此,1-24號單株中,1、2、6、7、15、16為可育單株,8、9、10、11、14、18、21、22、23、24為突變的不育單株,其他雙帶的為雜合可育株。
利用上述方法,從378株BC1F2后代中鑒定出不育單株89株,雜合可育株183株,純合可育株106。
抽穗揚花期進行田間育性性狀調查,圖4中雜合單株與普通核不育系單株穎花形態,其中雜合單株花藥為淺黃色,內含正常的花粉,表現出可育性狀;普通核不育系花藥呈白色,內無正常的花粉,表現出不育性狀。育性調查結果與上述基因分型結果完全一致。
對89株不育單株進行潮霉素基因PCR檢測,圖5中PCR檢測潮霉素基因,雜合株含有潮霉素基因,因此含有轉基因成份;普通核不育系不含潮霉素基因,因此不含轉基因成份。結果從89株不育單株中篩選出2株不含轉基因成分的普通核不育系。
潮霉素基因PCR檢測引物為HPT-F:GACGTCTGTCGAGAAGTTTC和HPT-R:GCTGTTATGCGGCCATTGTC,反應程序為:95℃預變性3min,35個循環:95℃變性10s,58℃退火15s,72℃延伸30s。
以上所述,僅是本發明的較佳實施例而已,并非對本發明作任何形式上的限制。雖然本發明已以較佳實施例揭示如上,然而并非用以限定本發明。任何熟悉本領域的技術人員,在不脫離本發明的精神實質和技術方案的情況下,都可利用上述揭示的方法和技術內容對本發明技術方案做出許多可能的變動和修飾,或修改為等同變化的等效實施例。因此,凡是未脫離本發明技術方案的內容,依據本發明的技術實質對以上實施例所做的任何簡單修改、等同替換、等效變化及修飾,均仍屬于本發明技術方案保護的范圍內。
SEQUENCE LISTING
<110> 湖南雜交水稻研究中心
<120> 一種培育水稻普通核不育系的方法
<130> 無
<160> 8
<170> PatentIn version 3.5
<210> 1
<211> 20
<212> DNA
<213> 人工序列
<400> 1
catggtggtc accaagtacc 20
<210> 2
<211> 20
<212> DNA
<213> 人工序列
<400> 2
ggccacaagc tgctcagcct 20
<210> 3
<211> 24
<212> DNA
<213> 人工序列
<400> 3
ggcacatggt ggtcaccaag tacc 24
<210> 4
<211> 24
<212> DNA
<213> 人工序列
<400> 4
aaacggtact tggtgaccac catg 24
<210> 5
<211> 23
<212> DNA
<213> 人工序列
<400> 5
gccggccaca agctgctcag cct 23
<210> 6
<211> 23
<212> DNA
<213> 人工序列
<400> 6
aaacaggctg agcagcttgt ggc 23
<210> 7
<211> 2232
<212> DNA
<213> 水稻
<400> 7
cgttgattgg cagcaactag ctagctcgcc gtccggccgg ccggccatgg cgcctaagat 60
ggtgatcagc ctggggagct cgcggcggcg gaagcgcggc gagatgctgt tccggttcga 120
ggccttctgc cagcccggct acccggcgaa cttcgccggc gccggcggct tcagggacaa 180
cgtgaggacg ctgctcggct tcgcgcacct ggaggccggc gtccacggcg agaccaagtg 240
ctggtcgttc cagctcgagc tgcaccgcca cccccccacc gtcgtgaggc tcttcgtcgt 300
cgaggaggag gtcgccgcct cgccgcaccg ccagtgccac ctctgccgcc atattgggtg 360
ggggaggcat ctgatatgca gcaagaggta tcacttcttg ctgccgagga gggaatcggc 420
ggcggaagcc gacggcctgt gcttcgcgat caaccacggc ggcggcggtg gcgcggagaa 480
agcgtcgtcg aaagggacga cgacgacggc ctccagcaga ggccacctgc tacacggcgt 540
cgtgcacctc aacggctacg gccacctcgt cgccctccac ggcctcgagg gcggctccga 600
cttcgtctcc ggccaccaga tcatggacct ctgggaccgc atttgctcag ccttgcacgt 660
aaggacggtg agcctggtgg acacggcgag gaagggccac atggagctga ggctgctgca 720
cggcgtcgcg tacggcgaga cgtggttcgg gcggtggggg tacaggtacg gccggccgag 780
ctacggcgtc gcgctgccgt cgtaccggca gtcgctgcac gtgctcggct ccatgccgct 840
ctgcgtgctg gtgccgcacc tgtcgtgctt cagccaggag ctccccatgg tggtcaccaa 900
gtaccaggcc atcagcggcc acaagctgct cagcctcggc gacctcctcc gcttcatgct 960
cgagctgcgc gcccgcctgc cggccacctc cgtcacggcc atggactacc ggggcatcat 1020
gtcggaggcc tcgtgccggt ggtcggcgaa gcgcgtcgac atggcggcgc gcgccgtcgt 1080
ggacgcgctc cgccgcgccg agccggcggc gcggtgggtc acgcggcagg aggtgcgcga 1140
cgcggcgcgc gcctacatcg gcgacacggg cctcctcgac ttcgtgctca agtccctcgg 1200
caaccacatc gtcggcaact acgtcgtgcg ccgcaccatg aacccggtga ccaaggtgct 1260
cgagtactgc ctcgaggacg tctccagcgt gctcccggcg gtcgccgccg gcggcggcgt 1320
gccggcgcag ggcaagatga gggtgaggtt ccagctcacg cgtgcgcagc tcatgaggga 1380
cctggtgcac ctgtaccggc acgtgctcaa ggagcccagc caggcgctca ccggcggcgc 1440
gttcggcgcg atcccggtgg cggtgcggat ggtcctggac atcaagcact tcgtcaaaga 1500
ttaccacgaa ggacaagccg cggcgagcag caatggcggt ggcggattcg ggcatcccca 1560
catcaacctg tgctgcacgc tgctcgtgag caacgggagc ccggagctag ctccaccgta 1620
cgagacggtg accctgccgg cgcacgcgac ggtgggcgag ctgaagtggg aggcgcagag 1680
ggtgttcagc gagatgtacc tcggcctgag gagcttcgcg gcggactccg tcgtcggggt 1740
cggcgccgac caggagggcc tcccggtgct cgggctggtc gacgtcggaa gcgccgtcgt 1800
ggtgcaaggg agcgtgggcg agcagataaa cggggaggac cacgagagga aggaggaggc 1860
ggcggcggcg gccgtgtgcg aggggagcgg cggcggcgag cgcgtcgtgg actgcgcgtg 1920
cggcgcggtg gacgacgacg gcgagcgcat ggcgtgctgc gacatctgcg aggcgtggca 1980
gcacacgcgg tgcgccggga tcgcggacac cgaggacgcg ccgcacgtct tcctctgcag 2040
ccggtgcgac aacgacgtcg tgtcgttccc gtccttcaac tgttagatgt gatgctgctg 2100
ctgctactgc tactactact gcctctgctg ctatatatga tgctacctag tacaagtgat 2160
cgagaattca atttgttttc tcggcaaaac caaaatgaaa acgaaggtaa aaccaagtga 2220
acttcagatc aa 2232
<210> 8
<211> 2407
<212> DNA
<213> 水稻
<400> 8
cgttgattgg cagcaactag ctagctcgcc gtccggccgg ccggccatgg cgcctaagat 60
ggtgatcagc ctggggagct cgcggcggcg gaagcgcggc gagatgctgt tccggttcga 120
ggccttctgc cagcccggct acccggcgaa cttcgccggc gccggcggct tcagggacaa 180
cgtgaggacg ctgctcggct tcgcgcacct ggaggccggc gtccacggcg agaccaagtg 240
ctggtcgttc cagctcgagc tgcaccgcca cccccccacc gtcgtgaggc tcttcgtcgt 300
cgaggaggag gtcgccgcct cgccgcaccg ccagtgccac ctctgccgcc atattggtcc 360
gtcgaacaaa ctacaattaa tcaatcaacc tttacatagg attgatccga tcgatgccat 420
ggtgttgtag ggtgggggag gcatctgata tgcagcaaga ggtatcactt cttgctgccg 480
aggagggaat cggcggcgga agccgacggc ctgtgcttcg cgatcaacca cggcggcggc 540
ggtggcgcgg agaaagcgtc gtcgaaaggg acgacgacga cggcctccag cagaggccac 600
ctgctacacg gcgtcgtgca cctcaacggc tacggccacc tcgtcgccct ccacggcctc 660
gagggcggct ccgacttcgt ctccggccac cagatcatgg acctctggga ccgcatttgc 720
tcagccttgc acgtaaggta gtagtagtat acatgtgcgt gtgcatgcat gcaagcaatg 780
caacgatgtc gggctgcgtg tgagaacatt tgcttgggca tggtgtggtg tatgcaagga 840
cggtgagcct ggtggacacg gcgaggaagg gccacatgga gctgaggctg ctgcacggcg 900
tcgcgtacgg cgagacgtgg ttcgggcggt gggggtacag gtacggccgg ccgagctacg 960
gcgtcgcgct gccgtcgtac cggcagtcgc tgcacgtgct cggctccatg ccgctctgcg 1020
tgctggtgcc gcacctgtcg tgcttcagcc aggagctccc catggtggtc accaagtacc 1080
aggccatcag cggccacaag ctgctcagcc tcggcgacct cctccgcttc atgctcgagc 1140
tgcgcgcccg cctgccggcc acctccgtca cggccatgga ctaccggggc atcatgtcgg 1200
aggcctcgtg ccggtggtcg gcgaagcgcg tcgacatggc ggcgcgcgcc gtcgtggacg 1260
cgctccgccg cgccgagccg gcggcgcggt gggtcacgcg gcaggaggtg cgcgacgcgg 1320
cgcgcgccta catcggcgac acgggcctcc tcgacttcgt gctcaagtcc ctcggcaacc 1380
acatcgtcgg caactacgtc gtgcgccgca ccatgaaccc ggtgaccaag gtgctcgagt 1440
actgcctcga ggacgtctcc agcgtgctcc cggcggtcgc cgccggcggc ggcgtgccgg 1500
cgcagggcaa gatgagggtg aggttccagc tcacgcgtgc gcagctcatg agggacctgg 1560
tgcacctgta ccggcacgtg ctcaaggagc ccagccaggc gctcaccggc ggcgcgttcg 1620
gcgcgatccc ggtggcggtg cggatggtcc tggacatcaa gcacttcgtc aaagattacc 1680
acgaaggaca agccgcggcg agcagcaatg gcggtggcgg attcgggcat ccccacatca 1740
acctgtgctg cacgctgctc gtgagcaacg ggagcccgga gctagctcca ccgtacgaga 1800
cggtgaccct gccggcgcac gcgacggtgg gcgagctgaa gtgggaggcg cagagggtgt 1860
tcagcgagat gtacctcggc ctgaggagct tcgcggcgga ctccgtcgtc ggggtcggcg 1920
ccgaccagga gggcctcccg gtgctcgggc tggtcgacgt cggaagcgcc gtcgtggtgc 1980
aagggagcgt gggcgagcag ataaacgggg aggaccacga gaggaaggag gaggcggcgg 2040
cggcggccgt gtgcgagggg agcggcggcg gcgagcgcgt cgtggactgc gcgtgcggcg 2100
cggtggacga cgacggcgag cgcatggcgt gctgcgacat ctgcgaggcg tggcagcaca 2160
cgcggtgcgc cgggatcgcg gacaccgagg acgcgccgca cgtcttcctc tgcagccggt 2220
gcgacaacga cgtcgtgtcg ttcccgtcct tcaactgtta gatgtgatgc tgctgctgct 2280
actgctacta ctactgcctc tgctgctata tatgatgcta cctagtacaa gtgatcgaga 2340
attcaatttg ttttctcggc aaaaccaaaa tgaaaacgaa ggtaaaacca agtgaacttc 2400
agatcaa 2407
- 還沒有人留言評論。精彩留言會獲得點贊!