CNGC2基因和其編碼的氨基酸序列在培育植物新品種中的應用的制作方法
本發明屬于植物生物技術領域,具體涉及CNGC2基因和其編碼的氨基酸序列在培育植物新品種中的應用。
背景技術:
Ca是植物生長發育的必需礦質元素。2011年我國生態學家方精云院士領導的科研小組測定了1227種中國植物葉子中11種營養元素的含量,發現Ca的含量為9g/kg,僅次于氮(20g/kg)和鉀(10g/kg),是植物體內第三大礦質元素。對美國不同森林土壤中Ca含量進行長達26,54和74年的測定,發現土壤Ca含量下降50%到78%。土壤中植物可以利用的Ca的流失主要是兩個因素造成的:一是酸雨在世界范圍內的大量發生,導致大量非游離態Ca溶解在水中成為游離態Ca而流失;二是植物通過根吸收土壤中的Ca,其中的大部分構成了植物細胞壁,被固定在木材及樹皮當中,收獲農作物、砍伐森林使土壤中的Ca不能被循環利用,從而導致環境中Ca的缺失。植物是吸收土壤Ca的主體。植物Ca營養的兩個根本問題是根對Ca營養的吸收和Ca在葉片中的分布。
從全球大趨勢而言,酸雨影響使土壤中可利用Ca2+量越來越少,這就使植物吸收Ca2+能力問題成為了熱點話題,但是,目前關于影響植物Ca2+分布基因的研究程度還較低,達不到產業應用的水平。
技術實現要素:
為了解決現有技術存在的上述問題,本發明提供了CNGC2基因和其編碼的氨基酸序列在培育植物新品種中的應用。經本申請的發明人研究發現,所述CNGC2基因能夠影響植物Ca2+分布,促進植物對Ca2+的吸收,從而應用于培育植物新品種中。
本發明所采用的技術方案為:一種影響植物Ca2+分布的基因,定名為CNGC2基因。所述CNGC2基因的序列如SEQ ID NO:1所示。
與所述CNGC2基因序列對應的,其編碼的氨基酸序列如SEQ ID NO:2所示。
本申請的發明人,通過植物生理、細胞和遺傳學方法,確定一個編碼環核苷酸門控的陽離子通道基因,即CNGC2基因,是調節植物植物葉細胞鈣營養的核心基因。所述CNGC2基因在葉子小葉脈末端周邊的葉肉細胞內表達,這個位置是木質部中的鈣離子釋放到葉肉細胞外間隙的位點。缺失所述CNGC2基因的擬南芥突變體在低鈣水培條件下可以正常生長,但是在相對高鈣環境下,葉子的生長受到顯著抑制,同時葉子出現早衰、局部葉肉細胞死亡和光合暗反應受抑制的表型。利用鈣離子特異性膜不通透性熒光染料,結合熒光顯微鏡,本申請的發明人發現,在缺失CNGC2基因的植物葉子內,因為葉肉細胞對鈣離子的吸收被阻斷,大量鈣離子在細胞外積累,說明CNGC2基因編碼的蛋白是調節鈣離子在植物葉子內正確分布必需的重要因子。
基于以上原理,本發明還公開了所述CNGC2基因和其編碼的氨基酸序列在培育植物新品種中的應用。
本發明的有益效果為:本發明提供了CNGC2基因和其編碼的氨基酸序列在培育植物新品種中的應用,所述CNGC2基因能夠影響植物Ca2+分布,促進植物對Ca2+的吸收,從而應用于培育植物新品種中。
附圖說明
為了更清楚地說明本發明實施例或現有技術中的技術方案,下面將對實施例或現有技術描述中所需要使用的附圖作簡單地介紹,顯而易見地,下面描述中的附圖僅僅是本發明的一些實施例,對于本領域普通技術人員來講,在不付出創造性勞動的前提下,還可以根據這些附圖獲得其他的附圖。
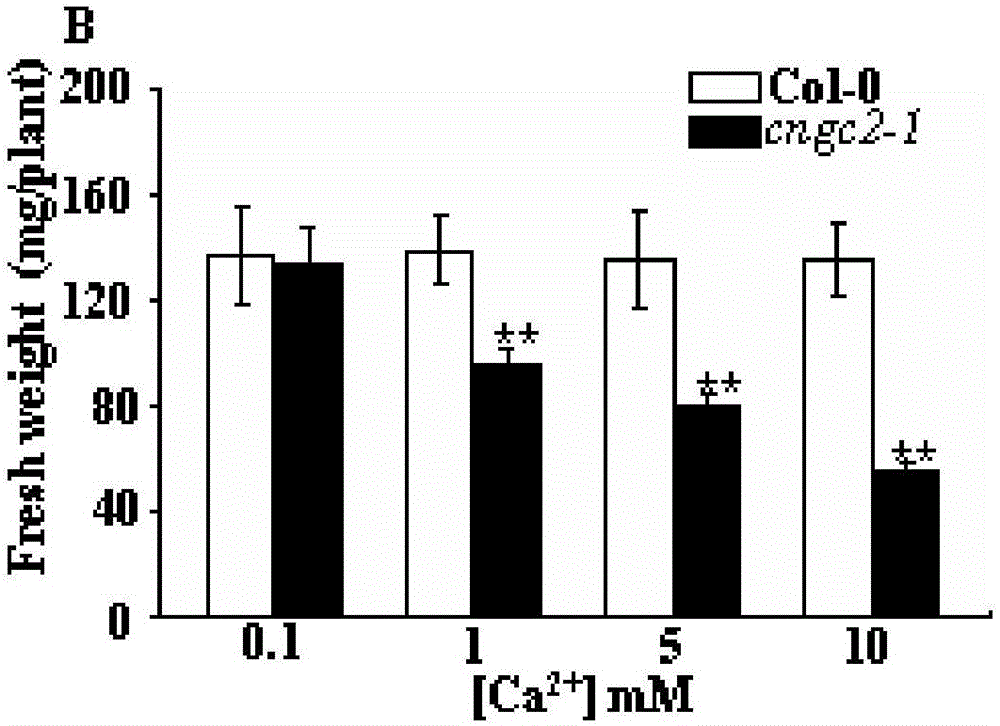
圖1是正常植物Col-0和cngc2-1擬南芥突變體在含有0.1mM Ca2+的水培溶液中培養3個星期,然后水培溶液更換為0.1,1,5或10mM Ca2+,繼續培養8天后,植物地上部分鮮重的統計分析圖;
圖2是正常植物Col-0和cngc2-1擬南芥突變體在含有0.1mM Ca2+的水培溶液中培養3個星期,然后水培溶液更換為0.1,1或10mM Ca2+,繼續培養8天后,葉中的Ca含量的統計分析圖;
圖3是正常植物Col-0和cngc2-1擬南芥突變體在含有0.1mM Ca2+的水培溶液中培養3個星期,然后水培溶液更換為0.1,1或10mM Ca2+,繼續培養8天后,根中的Ca含量的統計分析圖;
圖4是正常植物Col-0和cngc2-1擬南芥突變體在含有0.1mM Ca2+的水培溶液中培養3個星期,然后水培溶液更換為0.1或10mM Ca2+,繼續培養3天,植物葉子的葉柄插入含有膜不通透性、低親和性鈣離子熒光染料,Oregon Green BAPTA 488 5N(OGB)熒光染料的溶液中,在不同時間點,在藍色激光條件下,利用熒光顯微鏡觀察依賴鈣離子的綠色激發光的分布統計分析圖;
圖5是正常植物Col-0和cngc2-1擬南芥突變體在含有0.1mM Ca2+的水培溶液中培養3個星期,然后水培溶液更換為0.1或10mM Ca2+,繼續培養3天,快速收集各處理條件下成熟葉子進行液氮速凍,經過真空冷凍干燥以后,利用偶聯X能譜儀場發射掃描電子顯微鏡(Hitachi S-4800)測定葉片中表皮毛中的相對Ca含量的統計分析圖;
圖6是正常植物Col-0和cngc2-1擬南芥突變體在含有0.1mM Ca2+的水培溶液中培養3個星期,然后水培溶液更換為0.1或10mM Ca2+,繼續培養3天,快速收集各處理條件下成熟葉子進行液氮速凍,經過真空冷凍干燥以后,利用偶聯X能譜儀場發射掃描電子顯微鏡(Hitachi S-4800)測定葉片中各類細胞中的相對Ca含量的統計分析圖;
圖中,**表示顯著性差異,p<0.01。
具體實施方式
為使本發明的目的、技術方案和優點更加清楚,下面將對本發明的技術方案進行詳細的描述。顯然,所描述的實施例僅僅是本發明一部分實施例,而不是全部的實施例。基于本發明中的實施例,本領域普通技術人員在沒有做出創造性勞動的前提下所得到的所有其它實施方式,都屬于本發明所保護的范 圍。
在本發明中,若非特指,所有的份、百分比均為重量單位,所有的設備和原料等均可從市場購得或是本行業常用的。下述實施例中的方法,如無特別說明,均為本領域的常規方法。
實施例1
本實施例為CNGC2基因和其編碼的氨基酸序列對植物Ca2+分布的影響的驗證。
1.實驗材料與方法
1.1實驗材料
本實施例所用的材料為擬南芥野生型材料和cngc2-1擬南芥突變體,其中cngc2-1擬南芥突變體為不含有CNGC2基因的擬南芥植株,大腸桿菌菌株DH5α和根癌農桿菌菌株GV3101均由市場購得;Primer Star HS DNA Polymerase購自TaKaRa;EasyTaq PCR Supermix DNA聚合酶和T4DNA連接酶購自全式金公司;實驗所用MQA藥品及濃度為:5mM KNO3;1mM H3PO4;1mM MgSO4;0.1或10mM CaCl2;1/2MS微量元素;1/2MS Fe鹽;5mM MES;BTP調pH至5.7。
1.2實驗方法
1.2.1種子消毒與植株培養
分裝適量干燥好的擬南芥種子放于2ml離心管中,向管中加入1.8ml 75%乙醇并放置搖床上搖晃消毒20分鐘,然后在超凈工作臺中棄掉離心管中75%乙醇,倒入100%乙醇搖晃,將種子和乙醇倒在無菌濾紙上晾干,晾干后,用酒精燈火焰燒過的無菌鑷子點播在MQA固體培養基上,用醫用膠帶封皿放于4℃冰箱避光處理3天,最后將培養皿放在培養室培養,光周期為12小時光照/12小時黑暗,光強為75-100μmol m-2 s-1,培養溫度為22-25℃。
1.2.2擬南芥的水培培養
水培體系使用4×8的32孔4ml離心管盒為主要支持物。將同管架大小一 致并預先打孔的黑膠紙貼在離心管盒的管架上,孔大小隨苗大小而定,確保幼苗地上部分不可通過即可(一般是0.6mm×0.6mm圓形孔),盒里加入水培溶液650ml,將培養皿上生長10天的幼苗的根全部放入孔內的溶液中,幼苗地上部分架于溶液上,幼苗全部放好后蓋上離心管盒蓋,用于保濕。待植株再生長15-25天即可用于實驗。
1.2.6熒光染料觀察細胞外鈣的實驗方法
(1)用雙面刀片從葉柄基部剪取生長在MQA 0.1mM Ca2+水培溶液中的Col-0和cngc2-1擬南芥突變體葉片,將Col-0和cngc2-1葉柄分別插入到含有20μM Oregon Green 488BAPTA-5N(OGB)熒光染料的MQA 0.1mM Ca2+和MQA 10mM Ca2+溶液中,置于避光盒子內;
(2)在不同時間點將葉片用鑷子輕輕取出,放置在30%甘油鋪平的載玻片上,輕輕蓋上蓋玻片,用倒置熒光顯微鏡觀察,Oregon Green 488BAPTA-5N激發光為494nm。采用波長收集范圍是460-480nm,發射光收集范圍是500-560nm的濾光片進行拍照。
2.實驗驗證:
2.1缺失CNGC2基因的cngc2-1擬南芥突變體具有高鈣離子敏感表型
將種子播種在0.1mM Ca2+濃度下的培養皿中,生長21天后,移到0.1mM Ca2+水培溶液中生長21天,第22天更換0.1,1,5或10mM Ca2+溶液生長8天后采集數據,結果發現缺失CNGC2基因的cngc2-1擬南芥突變體在1mM Ca2+濃度下生長矮小,葉片邊緣黃化;生長在5,10mM Ca2+條件下的cngc2-1擬南芥突變體黃化及矮小現象更嚴重。對地上部分鮮重進行稱量,并對同一個濃度下的Col-0和cngc2-1擬南芥突變體進行統計學分析,結果如圖1所示,在0.1mM Ca2+濃度下,Col-0和cngc2-1擬南芥突變體鮮重相似,統計學分析沒有差異;在1,5,10mM Ca2+濃度下的水培體系下生長,cngc2-1擬南芥突變體植株鮮重明顯小于Col-0,統計學分析兩者具有顯著差異(p<0.01)。說明缺失CNGC2基因的cngc2-1擬南芥突變體具有高鈣離子敏感表型,所述CNGC2基 因能夠影響植物Ca2+分布,促進植物對Ca2+的吸收,從而應用于培育植物新品種中。
2.2缺失CNGC2基因的cngc2-1擬南芥突變體葉Ca含量顯著降低
將正常植物Col-0和cngc2-1擬南芥突變體在含有0.1mM Ca2+的水培條件下培養3個星期,然后分別給予植物0.1,1和10mM Ca2+處理,3天以后,利用原子吸收光譜分別測定葉和根的Ca含量,結果如圖2和圖3所示,圖2表示,0.1mM和1mM Ca2+濃度下Col-0和cngc2-1擬南芥突變體葉Ca含量沒有顯著區別,但是在10mM Ca2+濃度下,cngc2-1擬南芥突變體葉的Ca含量顯著低于Col-0;圖3表示,正常植物Col-0和cngc2-1擬南芥突變體根的Ca含量在三種不同Ca濃度培養條件下都沒有區別。這說明,CNGC2基因主要負責植物葉Ca的積累。
2.3高Ca2+條件下,Ca2+在cngc2-1擬南芥突變體細胞外過量積累
植物根吸收的營養元素通過維管系統向上運輸到葉片中,而后將營養物質卸載到質外體空間被葉片不同細胞吸收。Ca2+從細胞外空間到葉肉細胞轉運的過程,葉肉細胞對Ca2+吸收能力的減弱將會導致更多的Ca2+積累在細胞外空間。在增加Ca2+濃度下cngc2-1擬南芥突變體葉片早衰,可能原因是,葉肉細胞不能吸收Ca2+從而導致葉肉細胞Ca2+含量低而更多Ca2+積累在細胞外空間。為了驗證這個假說,本申請的發明人通過與Ca2+結合的熒光染料OGB觀察細胞外空間Ca2+的分布,將離體葉片放入能夠染細胞外Ca2+且不能跨細胞質膜的Ca2+特異性染料Oregon Green BAPTA 488 5N中,激發光波長/發射光波長=488nm/521nm,結果如圖4所示,10mM Ca2+濃度下,0分鐘時Col-0和cngc2-1擬南芥突變體的葉片均沒有檢測到熒光,20分鐘后能夠清晰的看到Col-0和cngc2-1擬南芥突變體的熒光,熒光表達的位置反應Ca2+的分布位置。2小時后0.1mM Ca2+濃度下,Col-0和cngc2-1擬南芥突變體細胞質外Ca2+主要分布在三級葉脈和小葉脈上,4小時后的觀察結果更為明顯,Col-0和cngc2-1擬南芥突變體熒光位置相似。在Col-0和cngc2-1擬南芥突變體的非葉脈區域沒有熒光信 號,說明Ca2+從維管束卸載到葉肉細胞是非常精準的過程使得游離于細胞外的Ca2+很少。
然而10mM Ca2+濃度下,我們觀測2小時和4小時處理后的熒光發現,cngc2-1擬南芥突變體葉脈與葉脈周圍都有很強的熒光信號值,在小葉脈周圍也檢測到較多的熒光信號。而Col-0在小葉脈周圍沒有檢測到信號,并且Col-0的熒光區域與非熒光區域有非常明確的界限,而cngc2-1擬南芥突變體中看不到明顯的界限。這說明cngc2-1擬南芥突變體非葉脈組織的細胞外空間中積累了更多的Ca2+,也說明在Ca2+從細胞外空間到葉肉細胞的分布過程中CNGC2基因扮演著重要的角色。
2.4.缺失CNGC2基因導致葉子中的Ca向葉表皮毛積累。
為了研究缺失CNGC2基因以后是否會影響植物葉和根的Ca含量,將正常植物Col-0和cngc2-1擬南芥突變體在含有0.1mM Ca2+的水培條件下培養3個星期,然后用含有0.1mM Ca2+和10mM Ca2+的水培溶液處理三天,剪下葉片,液氮速凍,用冷凍干燥機(EYELA FDU-2110)干燥24小時。用偶聯X能譜儀場發射掃描電子顯微鏡(Hitachi S-4800)測定葉片中某特定細胞的Ca含量,結果如圖5所示,cngc2-1擬南芥突變體葉表皮毛內的Ca含量顯著高于正常植物Col-0,這說明,缺失CNGC2基因以后,植物葉細胞吸收Ca的能力下降,更多的Ca積累到了葉表皮毛中。進一步對葉表面其他細胞內Ca含量進行測定,結果如圖5所示,在10mM Ca2+培養條件下,cngc2-1擬南芥突變體葉子表面如下細胞內的Ca含量顯著低于正常植物Col-0:葉表皮毛旁的蓮座細胞;葉表皮蓮座細胞旁葉肉細胞;獨立的葉肉細胞。這進一步證明,CNGC2調控Ca在植物葉片的分布。
以上所述,僅為本發明的具體實施方式,但本發明的保護范圍并不局限于此,任何熟悉本技術領域的技術人員在本發明揭露的技術范圍內,可輕易想到變化或替換,都應涵蓋在本發明的保護范圍之內。因此,本發明的保護范圍應以所述權利要求的保護范圍為準。
SEQUENCE LISTING
<110> 內蒙古和盛生態科技研究院有限公司
<120> CNGC2基因和其編碼的氨基酸序列在培育植物新品種中的應用
<130> 2010
<160> 2
<170> PatentIn version 3.3
<210> 1
<211> 2181
<212> DNA
<213> 未知
<400> 1
atgccctctc accccaactt catcttcagg tggattggac tgttttccga taagttccgt 60
cgacaaacga ctgggatcga tgaaaacagt aacctccaaa tcaacggtgg agattcgagc 120
agcagcggca gcgatgagac gccggtgcta agctccgtcg agtgttacgc ttgcacacaa 180
gtaggcgtcc cagctttcca ttcaactagc tgcgatcaag ctcacgcgcc ggagtggcgt 240
gcctccgccg gctcttctct agttccgatc caggaaggat ctgtccctaa cccagcccga 300
accagattcc gacgtctcaa aggtccgttt ggtgaagttc tcgatcctag gagcaagcgc 360
gtgcagagat ggaaccgcgc gttgctttta gctcgtggga tggctttagc ggtggatccg 420
ctcttcttct acgcgctttc catcggccga actaccggac cggcgtgtct ttacatggat 480
ggtgcgttcg ccgcggtggt cacggtgctc cgcacgtgtc tcgatgctgt tcatctttgg 540
cacgtgtggc ttcaattcag actggcctac gtctcgagag agtcgcttgt cgttggttgt 600
gggaagctcg tttgggatcc acgcgccatc gcgtctcact acgcacgctc tctcactggc 660
ttctggtttg atgttatcgt catcctccct gtccctcagg cagtgttttg gttagttgtg 720
ccgaaactga taagagaaga gaaggttaag ctgataatga cgattctgct gctaatattc 780
ttgttccagt tcctccccaa gatttatcac tgcatctgtt tgatgagaag gatgcagaag 840
gtcactggtt acatttttgg aactatttgg tggggttttg ctcttaatct catcgcatat 900
ttcatcgctt ctcatgttgc tgggggatgt tggtatgttc tcgcaataca gcgtgttgct 960
tcttgcataa gacaacaatg tatgagaacc gggaactgca atctgagtct ggcttgcaaa 1020
gaagaggtct gttaccaatt tgtgtcaccg acaagcacag ttggatatcc atgcttatct 1080
ggaaacctta ccagtgtggt caataagcct atgtgcttag actctaacgg accattccga 1140
tatggtatct accgttgggc acttccagtc atctccagca actctcttgc ggttaagatc 1200
ctttacccca tcttctgggg cctaatgact ctcagcacat ttgcgaatga tcttgagccc 1260
acaagcaact ggctcgaggt tattttcagt atagttatgg ttctaagtgg cttgttactt 1320
ttcacgctgt tgataggaaa cattcaggtg tttttgcatg cggtaatggc gaaaaaaagg 1380
aaaatgcaga tacggtgtag ggatatggaa tggtggatga aacgtaggca gttaccttcc 1440
cggttaagac agagggttag gcgatttgag cggcagagat ggaatgcctt gggtggtgaa 1500
gacgagctag aacttataca tgatttgcct ccgggtcttc gaagagatat caaacgatat 1560
ctttgctttg atctcattaa caaggtgcca ttgttcaggg gcatggacga cttgatcctc 1620
gacaacattt gcgatcgggc taagcctcga gtcttctcta aagacgaaaa gatcatccgt 1680
gaaggagatc ctgtacagag aatgatattc atcatgcgtg gacgagtcaa acgtatacag 1740
agcctaagca aaggcgtcct agccactagt acactagaac caggcggtta cttgggcgac 1800
gagctactct catggtgcct acgtcgcccg tttctggacc gtcttccccc ttcctcagca 1860
acatttgtct gcctagaaaa catcgaggca ttctccctcg gatccgaaga tcttaggtac 1920
attaccgatc atttccgtta taaattcgcg aacgagcggc ttaagcggac cgcaagatac 1980
tattcctcaa actggaggac gtgggcagcg gtaaatattc agatggcgtg gcgccggcgt 2040
aggaaaagaa cccgtggtga aaacatcggc ggttcgatga gtcctgtgtc ggagaatagc 2100
attgaaggta acagtgaacg ccggttactt cagtatgcag ctatgttcat gtccattcga 2160
ccgcatgatc atctcgaata a 2181
<210> 2
<211> 726
<212> PRT
<213> 未知
<400> 2
Met Pro Ser His Pro Asn Phe Ile Phe Arg Trp Ile Gly Leu Phe Ser
1 5 10 15
Asp Lys Phe Arg Arg Gln Thr Thr Gly Ile Asp Glu Asn Ser Asn Leu
20 25 30
Gln Ile Asn Gly Gly Asp Ser Ser Ser Ser Gly Ser Asp Glu Thr Pro
35 40 45
Val Leu Ser Ser Val Glu Cys Tyr Ala Cys Thr Gln Val Gly Val Pro
50 55 60
Ala Phe His Ser Thr Ser Cys Asp Gln Ala His Ala Pro Glu Trp Arg
65 70 75 80
Ala Ser Ala Gly Ser Ser Leu Val Pro Ile Gln Glu Gly Ser Val Pro
85 90 95
Asn Pro Ala Arg Thr Arg Phe Arg Arg Leu Lys Gly Pro Phe Gly Glu
100 105 110
Val Leu Asp Pro Arg Ser Lys Arg Val Gln Arg Trp Asn Arg Ala Leu
115 120 125
Leu Leu Ala Arg Gly Met Ala Leu Ala Val Asp Pro Leu Phe Phe Tyr
130 135 140
Ala Leu Ser Ile Gly Arg Thr Thr Gly Pro Ala Cys Leu Tyr Met Asp
145 150 155 160
Gly Ala Phe Ala Ala Val Val Thr Val Leu Arg Thr Cys Leu Asp Ala
165 170 175
Val His Leu Trp His Val Trp Leu Gln Phe Arg Leu Ala Tyr Val Ser
180 185 190
Arg Glu Ser Leu Val Val Gly Cys Gly Lys Leu Val Trp Asp Pro Arg
195 200 205
Ala Ile Ala Ser His Tyr Ala Arg Ser Leu Thr Gly Phe Trp Phe Asp
210 215 220
Val Ile Val Ile Leu Pro Val Pro Gln Ala Val Phe Trp Leu Val Val
225 230 235 240
Pro Lys Leu Ile Arg Glu Glu Lys Val Lys Leu Ile Met Thr Ile Leu
245 250 255
Leu Leu Ile Phe Leu Phe Gln Phe Leu Pro Lys Ile Tyr His Cys Ile
260 265 270
Cys Leu Met Arg Arg Met Gln Lys Val Thr Gly Tyr Ile Phe Gly Thr
275 280 285
Ile Trp Trp Gly Phe Ala Leu Asn Leu Ile Ala Tyr Phe Ile Ala Ser
290 295 300
His Val Ala Gly Gly Cys Trp Tyr Val Leu Ala Ile Gln Arg Val Ala
305 310 315 320
Ser Cys Ile Arg Gln Gln Cys Met Arg Thr Gly Asn Cys Asn Leu Ser
325 330 335
Leu Ala Cys Lys Glu Glu Val Cys Tyr Gln Phe Val Ser Pro Thr Ser
340 345 350
Thr Val Gly Tyr Pro Cys Leu Ser Gly Asn Leu Thr Ser Val Val Asn
355 360 365
Lys Pro Met Cys Leu Asp Ser Asn Gly Pro Phe Arg Tyr Gly Ile Tyr
370 375 380
Arg Trp Ala Leu Pro Val Ile Ser Ser Asn Ser Leu Ala Val Lys Ile
385 390 395 400
Leu Tyr Pro Ile Phe Trp Gly Leu Met Thr Leu Ser Thr Phe Ala Asn
405 410 415
Asp Leu Glu Pro Thr Ser Asn Trp Leu Glu Val Ile Phe Ser Ile Val
420 425 430
Met Val Leu Ser Gly Leu Leu Leu Phe Thr Leu Leu Ile Gly Asn Ile
435 440 445
Gln Val Phe Leu His Ala Val Met Ala Lys Lys Arg Lys Met Gln Ile
450 455 460
Arg Cys Arg Asp Met Glu Trp Trp Met Lys Arg Arg Gln Leu Pro Ser
465 470 475 480
Arg Leu Arg Gln Arg Val Arg Arg Phe Glu Arg Gln Arg Trp Asn Ala
485 490 495
Leu Gly Gly Glu Asp Glu Leu Glu Leu Ile His Asp Leu Pro Pro Gly
500 505 510
Leu Arg Arg Asp Ile Lys Arg Tyr Leu Cys Phe Asp Leu Ile Asn Lys
515 520 525
Val Pro Leu Phe Arg Gly Met Asp Asp Leu Ile Leu Asp Asn Ile Cys
530 535 540
Asp Arg Ala Lys Pro Arg Val Phe Ser Lys Asp Glu Lys Ile Ile Arg
545 550 555 560
Glu Gly Asp Pro Val Gln Arg Met Ile Phe Ile Met Arg Gly Arg Val
565 570 575
Lys Arg Ile Gln Ser Leu Ser Lys Gly Val Leu Ala Thr Ser Thr Leu
580 585 590
Glu Pro Gly Gly Tyr Leu Gly Asp Glu Leu Leu Ser Trp Cys Leu Arg
595 600 605
Arg Pro Phe Leu Asp Arg Leu Pro Pro Ser Ser Ala Thr Phe Val Cys
610 615 620
Leu Glu Asn Ile Glu Ala Phe Ser Leu Gly Ser Glu Asp Leu Arg Tyr
625 630 635 640
Ile Thr Asp His Phe Arg Tyr Lys Phe Ala Asn Glu Arg Leu Lys Arg
645 650 655
Thr Ala Arg Tyr Tyr Ser Ser Asn Trp Arg Thr Trp Ala Ala Val Asn
660 665 670
Ile Gln Met Ala Trp Arg Arg Arg Arg Lys Arg Thr Arg Gly Glu Asn
675 680 685
Ile Gly Gly Ser Met Ser Pro Val Ser Glu Asn Ser Ile Glu Gly Asn
690 695 700
Ser Glu Arg Arg Leu Leu Gln Tyr Ala Ala Met Phe Met Ser Ile Arg
705 710 715 720
Pro His Asp His Leu Glu
725
- 還沒有人留言評論。精彩留言會獲得點贊!
- 新的重組型抗體及其cdr氨基酸序列和編碼它們的基因的制作方法
- 包括一種抗原或包含氨基酸序列化合物的體內發生器的治療性組合物的制作方法
- 針對CXCR4和其他GPCRs的氨基酸序列以及包含所述氨基酸序列的多肽的制作方法
- 雞貧血病毒疫苗及診斷方法
- 用于控制致病體的核酸序列和氨基酸序列的制作方法
- 一種鐵氫化酶基因及其編碼的氨基酸序列和異源表達系統的制作方法
- 精氨酸在提高發酵培養氨基酸序列含有精氨酸-精氨酸的多肽的表達量中的應用及方法
- 一組可提高sod耐熱溫度和熱穩定性的氨基酸序列及其應用的制作方法
- 菜豆幾丁質酶基因及其編碼產物的氨基酸序列的制作方法
- 一種中藥有效成分的氨基酸序列獲得方法及其所確定的具體氨基酸序列的制作方法