四環素耐藥基因檢測并聯探針、基因芯片、試劑盒與檢測法的制作方法
本發明屬于分子生物學領域,涉及一種四環素耐藥基因檢測的并聯探針、基因芯片、試劑盒與檢測方法。
背景技術:
四環素類藥物屬廣譜抗生素,發現于20世紀40年代,是通過阻止氨酰tRNA與核糖體結合位點(A)的結合來阻止菌體蛋白合成的一類抗生素,具有廣泛的抗菌活性。在臨床中以其有效的殺菌作用及較小的副作用而被廣泛用于治療人和動物的細菌性感染,然而,近年來,耐藥性的出現限制了它們的使用。
1980年Menders等第1次研究腸桿菌科和假單胞菌質粒的四環素耐藥決定子的基因機制,到現在已經明確了29個不同的Tet基因和3個Otr基因,其中18種Tet基因和1種Otr基因編碼外輸泵,種Tet基因和1種Otr基因即Otr(A)編碼核糖體保護蛋白,1種Tet基因即Tet(X)編碼一種修飾或鈍化四環素的酶,但Tet(X)基因的功能在天然宿主中并不能起作用。
在革蘭氏陽性菌和革蘭氏陰性菌中都有外輸泵基因,而且大部分外輸泵基因都具有四環素抗性。四環素外輸泵蛋白屬于主要異化超家族(MFS),是目前Tet蛋白中研究最清楚的。所有Tet外輸泵基因都編碼膜相關蛋白可將四環素泵出胞外,降低了細胞內藥物濃度保護了胞內的核糖體,從而產生耐藥性。每個Tet外輸泵基因都編碼約46 KDa膜結合外輸泵蛋白,這些蛋白四環素常見耐藥基因為Tet(A)、Tet(B)、Tet(C)、Tet(D)、Tet(E)、Tet(G)、Tet(L)、Tet(M)、Tet(M)和Tet(P)。
生物芯片技術是九十年代發展起來的一項新興生物技術。按照檢測對象來分類,可以分為基因芯片和蛋白質芯片等兩大類。與蛋白質芯片相比,基因芯片利用的是核苷酸鏈之間的互補作用,特異性強,性好。本發明所提供的四環素耐藥基因芯片與傳統的細菌藥敏性檢測方法相比,直接針對病原體四環素耐藥基因進行檢測,其檢測通量大、所需樣本量少、靈敏度高、準確性高和特異性好等優勢。
技術實現要素:
本發明目的是提供一種特異性好、結果準確、假陽性率低、檢測效率高的四環素耐藥基因檢測的并聯探針、基因芯片、試劑盒與檢測方法。
本發明實現其目的技術方案為:
一種四環素耐藥基因檢測并聯探針,包括探針1~26,序列分別為SEQ ID NO.1~26。
一種四環素耐藥基因檢測芯片,包括固體相和權利要求1所述的探針組。
作為優選地,所述固相載體為氨基修飾的玻璃基片、尼龍膜或硝酸纖維素膜。
一種四環素耐藥基因檢測試劑盒,包括前述的基因芯片。
在上述四環素耐藥基因檢測試劑盒中,還包括四環素耐藥靶基因擴增引物對,引物序列如SEQ ID NO.27~44所示。
作為優選地,所述引物對的下游引物的5’端含有CY3熒光染料標記。
作為優選地,上述四環素耐藥基因檢測試劑盒,還包括PCR反應體系、雜交緩沖液、洗滌液。
在上述技術方案中,20 μL 的PCR反應體系包括:50~150 ng/μL的擴增模板 2 μL,Premix Taq 10 μL,濃度為8~12 μM的上、下游引物各1 μL,雙蒸水6 μL。
一種四環素耐藥基因檢測方法,使用前述的四環素耐藥基因檢測試劑盒進行檢測,當檢測對象的三個探針顯示的檢測結果一致時可以判斷樣品是否感染四環素耐藥基因;當檢測對象的三個探針顯示的檢測結果不一致時,不能判斷該待測樣品為陽性或陰性,使用該檢測試劑盒重新檢測或者使用現有其它檢測方法進行進一步驗證。
作為優選地,PCR擴增反應程序為95℃ 5 min;95℃10 s、55℃ 30 s、72℃30 s、30個循環;72℃ 10 min。
本發明的有益效果是:每個耐藥基因設計3條高特異性的探針,當3條探針的檢測結果均為陽性時才判斷該項指標為陽性,能極大的提高產品的準確性和精確性,減少其它方法容易出現的假陽性問題;本發明通過PCR技術對病原體四環素耐藥基因組進行擴增,檢測的特異性高,擴增產物與生物芯片上的寡核苷酸探針進行雜交,通過CY3標記的PCR反向擴增引物檢測雜交結果。操作簡單、結果易讀,滿足醫療等現場檢測需求,為臨床醫生快速診斷患者是否被感染四環素耐藥基因提供了便捷、快速和準確的理論依據,具有廣闊的臨床應用市場潛力。
附圖說明
圖1 檢測四環素耐藥基因的基因芯片的示意圖。
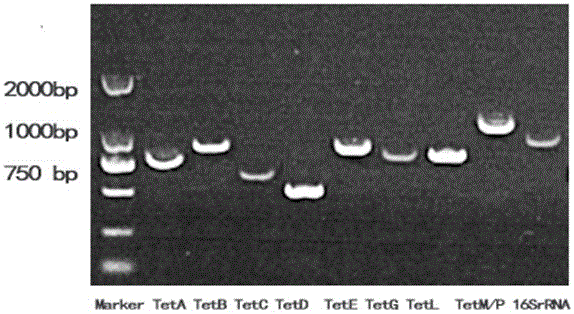
圖2待測樣品四環素耐藥基因擴增后PCR產物電泳條帶圖;Marker DL2000 (TAKARA)。
具體實施方式
下面將結合附圖,對本發明的優選實施例進行詳細的描述。實施例中未注明具體條件的實驗方法,通常按照常規條件,例如分子克隆實驗指南(第三版,J. 薩姆布魯克等著)中所述的條件,或按照制造廠商所建議的條件。以下實施例用于說明本發明,但不用于限制本發明的范圍。
實施例1目的片段引物與探針設計
查找NCBI數據庫中四環素耐藥基因的基因序列Tet(A)、Tet(B)、Tet(C)、Tet(D)、Tet(E)、Tet(G)、Tet(L)、Tet(M)和Tet(P),使用芯片探針設計軟件(ArrayDesigner4.2)針對每個耐藥基因設計3條探針,具體探針序列如SEQ ID NO .1~26所示。
使用NCBI在線設計軟件(Primer-BLAST)設計Tet(A)、Tet(B)、Tet(C)、Tet(D)、Tet(E)、Tet(G)、Tet(M)、Tet(L)和Tet(P)基因的PCR擴增引物,設計四環素耐藥基因靶基因的擴增引物與探針。為了使芯片結果用顯色液顯色后可直接通過肉眼判斷,在四環素耐藥基因靶基因擴增引物的反向(R)引物5’端用CY3熒光染料進行標記,具體序列如SEQ ID NO .27~44所示。
實施例2 芯片的制備
將實施例1中的并聯探針按照表1的順序點樣到氨基修飾的玻璃基片上,得到含有探針的基因芯片(如圖1所示)。探針濃度為30 μM,每個點0.2 μL,然后80℃孵育1.5 h。
表1 四環素耐藥基因檢測芯片探針排列順序
實施例3 檢測方法
1、待測樣品基因組提取
使用細菌基因組DNA提取試劑盒,按照操作說明書的步驟提取待測樣品的基因組作為擴增模板。
2、PCR擴增目的片段
經過大量實驗摸索,確定Tet檢測的靶基因PCR擴增體系和PCR反應程序。將每個Tet基因Tet(A)、Tet(B)、Tet(C)、Tet(D)、Tet(E)、Tet(G)、Tet(L)、Tet(M/P)和16SrRNA擴增的引物,分別配制PCR擴增緩沖液體系,總體積為20 μL的PCR擴增體系中含有試劑分別為:Premix LATaq 10μL、F引物(10μM)1 μL、F引物(10μM)1 μL、擴增模板2 μL、雙蒸水6 μL。
PCR反應程序為:95℃ 5 min;95℃10 s、55℃ 30 s、72℃30 s、30個循環;72℃ 10 min。
3、PCR產物與芯片雜交,照如下步驟操作:
a. 在實施例2中制備好的基因芯片上,將待測樣品的Tet(A)、Tet(B)、Tet(C)、Tet(D)、Tet(E)、Tet(G)、Tet(L)、Tet(M/P)和16SrRNA的PCR擴增產物混勻并點樣到芯片的1個檢測槽中。具體步驟為:PCR產物95℃解鏈5分鐘,迅速置于冰上,PCR產物與雜交緩沖液(50%甲酰胺,10×SSC,0.2%SDS)等體積混合,每孔點10 μL混合液,在濕盒中,于60℃雜交孵育2小時。
b. 取出孵育后的基因芯片,用洗滌液(0.3×SSC緩沖液)清洗3次。
c. 將芯片放入激光共聚焦檢測儀(GenePix)在650 nm處掃描芯片;軟件分析熒光信號強度值,給出結果。
實施例4 檢測待測樣品
從第三軍醫大學獲取四環素耐藥細菌感染的樣本5份(膝關節積液),無四環素耐藥基因感染樣本2份(對照)。按照實施例1和3中的引物和方法分別提取待測樣品的基因組,進行PCR擴增后,使用實施例2中的基因芯片進行檢測。PCR擴增后的產物電泳條帶表明本發明設計的擴增引物特異性高,能特異、靈敏地擴增出目標基因(如圖2所示)。
對于細菌感染的樣品,激光共聚焦檢測儀(GenePix)在650 nm處掃描芯片,探針點熒光信號較強(大于20),記為(+),熒光信號較弱(小于5),則記為(-),熒光信號強度難以分辨(5~20)則記為(*)。檢測中同時設計陽性和陰性質控對照,只有當陽性和陰性質控對照的檢測結果同時分為別陽性(+)和陰性(-),才認為該組檢測結果可信。
判斷檢測結果時,當Tet(A)、Tet(B)、Tet(C)、Tet(D)、Tet(E)、Tet(G)、Tet(L)、Tet(M/P)基因的3個探針均顯示陽性時才判斷該樣本帶有該耐藥基因,如果其中某一個或者兩個探針顯示陽性,則應對該檢測樣品重新檢測,3個探針結果須一致才能采信該檢測結果。3個探針檢測結果不一致時,也可使用現有其它技術(比如Elisa方法)進一步檢測驗證。檢測結果如表2所示。
表2 樣本檢測結果
結果顯示,陽性質控對照均為陽性(+),陰性質控對照均為陰性(-),說明實驗結果可信。其中,7份樣本芯片結果顯示:5份樣本中均檢測到四環素耐藥基因感染,樣本1被四環素耐藥基因Tet(A)感染,樣本2被四環素耐藥基因Tet(E)感染,樣本3被四環素耐藥基因Tet(L)感染,樣本4被四環素耐藥基因Tet(L)感染,樣本5被四環素耐藥基因Tet(E)感染。2份對照樣本均未檢測到四環素耐藥基因,與實際臨床樣本信息情況相符。
本發明公開的探針、基因芯片、試劑盒,特異性高、結果非常準確,檢測快速、工作效率高,操作簡單,滿足醫院等的現場檢測需求,為診斷四環素耐藥細菌感染提供可靠的實驗依據。
SEQUENCE LISTING
<110> 重慶高圣生物醫藥有限責任公司
<120> 四環素耐藥基因檢測并聯探針、基因芯片、試劑盒與檢測法
<130> 2016
<160> 44
<170> PatentIn version 3.3
<210> 1
<211> 55
<212> DNA
<213> 人工序列
<400> 1
tttttttttt tttttctgtc gactacgcca tcatggcgac ggcgcctttc ctttg 55
<210> 2
<211> 55
<212> DNA
<213> 人工序列
<400> 2
tttttttttt ttttttcgtg gccggcatca ccggggcgac tggggcggta gccgg 55
<210> 3
<211> 55
<212> DNA
<213> 人工序列
<400> 3
tttttttttt tttttctgat ggcgatgagc gcgcgcggca cttcggcttc atgag 55
<210> 4
<211> 55
<212> DNA
<213> 人工序列
<400> 4
tttttttttt tttttttgga acagctctat atggaaagct atcagaccag ctggg 55
<210> 5
<211> 55
<212> DNA
<213> 人工序列
<400> 5
tttttttttt tttttttggg attatggtaa atggcttagg gtcgatcatt ggatt 55
<210> 6
<211> 55
<212> DNA
<213> 人工序列
<400> 6
tttttttttt tttttctatt ctcattctag cccgatttat tcaaggaatt ggtgc 55
<210> 7
<211> 55
<212> DNA
<213> 人工序列
<400> 7
tttttttttt tttttttaac tcagcacgaa tattgtttgt tgaaaaaggc tatca 55
<210> 8
<211> 55
<212> DNA
<213> 人工序列
<400> 8
tttttttttt ttttttctcg ggaaaagcgt tggtgaccaa aggtgccttt tatca 55
<210> 9
<211> 55
<212> DNA
<213> 人工序列
<400> 9
tttttttttt tttttaatta ctcagtgcct gttataagca gcaattaatt atgat 55
<210> 10
<211> 55
<212> DNA
<213> 人工序列
<400> 10
tttttttttt tttttagttt catacaacgg ttatcaaaga tgttctatta tggat 55
<210> 11
<211> 55
<212> DNA
<213> 人工序列
<400> 11
tttttttttt tttttcttta ctgcttgatg atgtggcgaa taaagcgggt tatac 55
<210> 12
<211> 55
<212> DNA
<213> 人工序列
<400> 12
tttttttttt ttttttgttc aaaaaagtaa caggggtcac actggctagc tatat 55
<210> 13
<211> 55
<212> DNA
<213> 人工序列
<400> 13
tttttttttt tttttggtca gatcgcatag gtcgtcgccc tgtattatta ctttc 55
<210> 14
<211> 55
<212> DNA
<213> 人工序列
<400> 14
tttttttttt tttttactac gcattaatgg caacagccag cgtagtgtgg gtgtt 55
<210> 15
<211> 55
<212> DNA
<213> 人工序列
<400> 15
tttttttttt tttttctggt attaccggtg cgactggagc tgttgcagcc tcaac 55
<210> 16
<211> 55
<212> DNA
<213> 人工序列
<400> 16
tttttttttt ttttttgctt ggacaacttt cggatgctta cggtcggcgt ccggt 55
<210> 17
<211> 55
<212> DNA
<213> 人工序列
<400> 17
tttttttttt tttttgagcc gcagtcgatt acacgatcat ggcatctgcg ccggt 55
<210> 18
<211> 55
<212> DNA
<213> 人工序列
<400> 18
tttttttttt tttttgccga ctcatctccg gcgtcacggg cgcaaccgga gccgt 55
<210> 19
<211> 55
<212> DNA
<213> 人工序列
<400> 19
tttttttttt tttttctttt ctgatcatca gtattctgtc attcctgata tttgt 55
<210> 20
<211> 55
<212> DNA
<213> 人工序列
<400> 20
tttttttttt tttttcagaa ccttttgttg aacctgcact ggggaaaaat atttc 55
<210> 21
<211> 55
<212> DNA
<213> 人工序列
<400> 21
tttttttttt tttttgtgga ggacttatat ttggtacagt agcagggttt atctc 55
<210> 22
<211> 55
<212> DNA
<213> 人工序列
<400> 22
tttttttttt tttttagtgg gaaaatacga aggtgaacat catagacacg ccagg 55
<210> 23
<211> 55
<212> DNA
<213> 人工序列
<400> 23
tttttttttt tttttaagta tatcgttcat tatcagtttt agatggggca attct 55
<210> 24
<211> 55
<212> DNA
<213> 人工序列
<400> 24
tttttttttt tttttcactc aattgaaaaa taatgaggtc attcttagtg gagaa 55
<210> 25
<211> 34
<212> DNA
<213> 人工序列
<400> 25
tttttttttt tttttccgat agtgaaccag tacc 34
<210> 26
<211> 38
<212> DNA
<213> 人工序列
<400> 26
tttttttttt tttttctatc tagctagcta gctagcta 38
<210> 27
<211> 17
<212> DNA
<213> 人工序列
<400> 27
gtgaaaccca acagacc 17
<210> 28
<211> 17
<212> DNA
<213> 人工序列
<400> 28
gagcaagtag agggcag 17
<210> 29
<211> 18
<212> DNA
<213> 人工序列
<400> 29
caactttacg gcacaatc 18
<210> 30
<211> 15
<212> DNA
<213> 人工序列
<400> 30
caatcgcaat acccg 15
<210> 31
<211> 18
<212> DNA
<213> 人工序列
<400> 31
gattgtcacg accacatc 18
<210> 32
<211> 18
<212> DNA
<213> 人工序列
<400> 32
cataaaccag ccattgag 18
<210> 33
<211> 16
<212> DNA
<213> 人工序列
<400> 33
tgaacagcat tctcgc 16
<210> 34
<211> 18
<212> DNA
<213> 人工序列
<400> 34
gccgataata acttggtg 18
<210> 35
<211> 16
<212> DNA
<213> 人工序列
<400> 35
gaaccgcact gtgatg 16
<210> 36
<211> 15
<212> DNA
<213> 人工序列
<400> 36
ggtcagcaac ccttg 15
<210> 37
<211> 15
<212> DNA
<213> 人工序列
<400> 37
ttgtgccagc agagc 15
<210> 38
<211> 16
<212> DNA
<213> 人工序列
<400> 38
catccatccc tgtgtg 16
<210> 39
<211> 15
<212> DNA
<213> 人工序列
<400> 39
tttggctttg cgttc 15
<210> 40
<211> 18
<212> DNA
<213> 人工序列
<400> 40
tcaaggtcca taggtagc 18
<210> 41
<211> 20
<212> DNA
<213> 人工序列
<400> 41
gaacatcata gacacgccag 20
<210> 42
<211> 19
<212> DNA
<213> 人工序列
<400> 42
cttgaataca ccgagcagg 19
<210> 43
<211> 21
<212> DNA
<213> 人工序列
<400> 43
gagagtttga tyctggctca g 21
<210> 44
<211> 20
<212> DNA
<213> 人工序列
<400> 44
aaggaggtga tccarccgca 20
- 還沒有人留言評論。精彩留言會獲得點贊!