一株青霉素酰化酶及其編碼基因、產生菌和應用的制作方法
本發明涉及了一株青霉素酰化酶產生菌株,該菌產的一種青霉素酰化酶及其基因,以及酶法合成青霉素G類以及頭孢菌素類半合成抗生素的方法,屬于生物制藥技術領域。
背景技術:
青霉素酰化酶(penicillin acylase,PGA)又稱為青霉素氨基酰化酶,是一種N末端親核絲氨酸水解酶。該酶在酸性以及中性條件下,可用于催化6-氨基青霉烷酸(6-APA)和7-氨基脫乙酰頭孢烷酸(7-ADCA)與酰基供體發生酰基化反應,制備半合成抗生素;而在偏堿性條件下,催化青霉素鉀鹽等水解成為6-APA和苯乙酸(PAA),而6-APA是大量制備半合成β-內酰胺抗生素所需要的重要母核。并且PGA能合成的底物范圍廣,可以催化制備高效、廣譜、適用于不同用途的新型β-內酰胺抗生素。因此,青霉素酰化酶是工業上應用價值很高的酶。
目前生產最多、應用最廣的半合成抗生素是半合成青霉素類和頭孢菌素類抗生素。其中半合成青霉素類抗生素是以6-氨基青霉烷酸為母核,對其側鏈進行化學修飾等產生的,主要有氨芐青霉素、阿莫西林、阿洛西林、氨芐西林等;頭孢菌素類抗生素是以7-氨基去乙酰氧基頭孢烷酸為母核形成的抗生素藥物,主要有頭孢氨芐、頭孢羥氨芐、頭孢拉定、頭孢克洛等。
另外,頭孢替唑是第一代頭孢菌素類抗生素,通過抑制細菌細胞壁的合成而發揮抗菌活性。臨床上主要用于治療呼吸系統感染、泌尿系統感染、敗血癥、腹膜炎等。據現有文獻報道,頭孢替唑的合成方法主要是化學法,酶法合成即以7-氨基-3- (2-氨基-1,3,4-噻二唑)硫甲基頭孢烷酸為母核形成頭孢替唑尚未有文獻報道。
盡管青霉素酰化酶的研究已取得重要進展,但性能優良并能夠適用于多種新一代β-內酰胺抗生素合成的酶類仍然難以滿足需求,因此篩選性能更優越的青霉素酰化酶,如pH耐受性好、合成底物范圍廣泛的酶更適用于工業催化等,對酶法催化半合成β-內酰胺類抗生素具有重要意義。
技術實現要素:
本發明公開了一種青霉素酰化酶產生菌,該菌產的一種青霉素酰化酶及其基因,該青霉素酰化酶表達載體構建和該酶在大腸桿菌(E.coli)BL21(DE3)中的異源表達獲得重組酶,該青霉素酰化酶合成以6-氨基青霉烷酸、7-氨基去乙酰氧基頭孢烷酸、7-氨基-3- (2-氨基-1,3,4-噻二唑)硫甲基頭孢烷酸為母核的β-內酰胺類抗生素。
本發明的目的之一是提供一種青霉素酰化酶產生菌,該菌株為木糖氧化無色桿菌,命名為Achromobacter xylosoxidans PX02,保藏登記號為CCTCC NO:M 2016392。
本發明的又一目的是提供所述青霉素酰化酶產生菌產生的青霉素酰化酶,其特征在于所述青霉素酰化酶具有SEQ ID NO:2所示的氨基酸序列。
本發明的再一目的是提供所述青霉素酰化酶的編碼基因、重組載體或重組菌,所述編碼基因的核苷酸序列如SEQ ID NO:1所示,所述重組菌由所述編碼基因與載體pET-28a構建的表達載體轉化到大腸桿菌BL21(DE3)所得。
本發明的目的之一是提供一種青霉素酰化酶產生菌在合成β-內酰胺類抗生素中的應用。
為了實現本發明的目的,本發明首先從南京周邊藥廠土壤中篩選獲得青霉素酰化酶菌株PX02,通過16s rDNA基因序列對比分析,該菌株被鑒定為Achromobacter xylosoxidan,命名為Achromobacter xylosoxidans PX02,其保藏登記號為CCTCC NO:M 2016392。
本發明分離克隆到Achromobacter xylosoxidans PX02 菌株來源青霉素酰化酶PGAPX02的編碼基因,它具有SEQ ID NO:1所示的核苷酸序列,為此基因的改造并在各種外源基因表達系統中高效表達提供了優良的基因材料。通過PCR法分離克隆了這一青霉素酰化酶PGAPX02基因,DNA全序列分析結果表明,該青霉素酰化酶PGAPX02基因全長2583個核苷酸,編碼861個氨基酸,NCBI序列比對后發現與Achromobacter xylosoxidans A8 的序列相似性90%,成熟肽氨基酸同源性為94.7%。其全部氨基酸序列示于SEQ ID NO:2。
本發明構建了青霉素酰化酶PGAPX02基因的表達載體。它可以通過本領域常規方法將本發明所述基因連接于各種載體上構建而成。所述的載體可為本領域常規的各種載體,如市售的質粒、粘粒、噬菌體或病毒載體等,優選pET28a。較佳的,可通過下述方法制得本發明的重組表達載體:將通過PCR擴增所得的產物用限制性內切酶Nhe I和Xho I進行雙酶切,形成互補的粘性末端,同時將載體pET28a用限制性內切酶Nhe I和Xho I進行雙酶切經T4 DNA連接酶連接,構建了含有本發明的青霉素酰化酶PGAPX02基因的重組表達載體pET28a-pgapx02。
本發明將上述重組表達載體轉化至宿主細胞制得重組表達轉化體。所述的宿主可為本領域常規的各種宿主,只要能滿足重組表達載體可穩定地自行復制,且所攜帶的本發明的青霉素酰化酶PGAPX02基因能被有效表達即可。本發明優選大腸桿菌,更優選大腸埃希氏菌(E.coli)BL21(DE3)。將前述重組表達質粒pET28a-pgapx02轉化至(E.coli)BL21(DE3),即可得本發明優選的基因工程菌株,即大腸埃希氏菌(E.coli) BL21(DE3)/pET28a-pgapx02。并將表達載體成功導入大腸桿菌表達菌BL21中,加入IPTG誘導表達后,經SDS-PAGE驗證在BL21中成功表達了青霉素酰化酶PGAPX02,經檢測,該表達酶的活力達到4000U/L。
本發明對青霉素酰化酶PGAPX02進行了酶學性質的研究。青霉素酰化酶PX02的最適反應pH為8.0,在pH 6.5~9的范圍具有較高的穩定性(相對酶活90%以上)。其最佳反應溫度為55℃,在25℃-45℃下溫育2 h后都能保持較高的青霉素酰化酶活力(相對酶活90%以上)。實驗證明該青霉素酰化酶對多種有機溶劑都具有良好的耐受性,在部分有機溶劑中青霉素酰化酶的穩定性提高。
本發明還提供青霉素酰化酶在合成β-內酰胺類抗生素中的應用。所述青霉素酰化酶PGAPX02催化合成青霉素G類半合成抗生素如阿莫西林、氨芐西林和頭孢菌素類半合成抗生素如頭孢氨芐、頭孢羥氨芐、頭孢替唑。
所述的酶催化反應分別以6-氨基青霉烷酸、7-氨基去乙酰氧基頭孢烷酸和7-氨基-3- (2-氨基-1,3,4-噻二唑)硫甲基頭孢烷酸為母核,以對應的側鏈為酰基供體,在青霉素酰化酶PGAPX02的催化作用下進行縮合反應,以6-氨基青霉烷酸為母核合成阿莫西林、氨芐西林和以7-氨基去乙酰氧基頭孢烷酸為母核合成頭孢氨芐、頭孢羥氨芐,以及以7-氨基-3- (2-氨基-1,3,4-噻二唑)硫甲基頭孢烷酸為母核合成頭孢替唑,阿莫西林和氨芐西林的合成率分別為65%-70%和67%-72%,頭孢氨芐和頭孢羥氨芐的合成率分別為72%-76%和75%-80%,頭孢替唑的合成率為63%-69%。所述的母核與酰基供體的摩爾比為1:2~1:4,其中母核的濃度為20~100mmol/L,酰基供體的濃度為40~300 mmol/L。所述的體系為有機溶劑和水組成的混合溶劑,所述有機溶劑選自丙三醇、DMSO、聚乙二醇中的任意一種,其體積百分含量為10~80%。所述的酰基供體為羧酸及其甲酯、酰胺、酸酐及其衍生物等,所述羧酸可以為脂肪酸或芳香酸。
本發明的有益效果在于與已報道的木糖氧化無色桿菌相比,菌株Achromobacter xylosoxidans PX02的青霉素酰化酶PGAPX02單位菌體產酶量較高,發酵24 h青霉素酰化酶活力達到4000 U/L,比活力達到400 U/L/OD600;該青霉素酰化酶有耐高溫、pH穩定性好、有機溶劑耐受性強、底物范圍廣泛等優點,該青霉素酰化酶成功催化合成五種β-內酰胺類抗生素,并且具有較好的合成頭孢替唑的能力,表明其在催化合成β-內酰胺類抗生素方面有很好的應用前景。
附圖說明
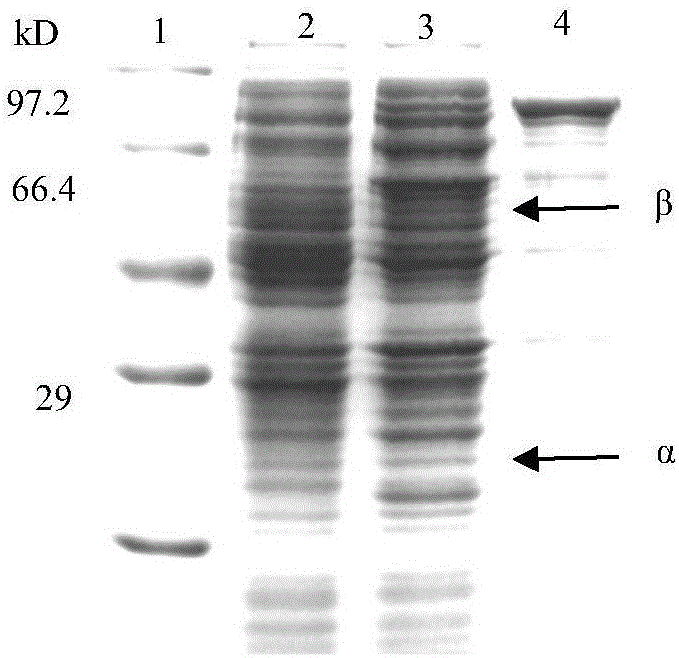
圖1顯示青霉素酰化酶PGAPX02的SDS-PAGE電泳分析。
圖2顯示青霉素酰化酶PGAPX02的最適反應pH。
圖3顯示青霉素酰化酶PGAPX02的pH穩定性(在各pH條件下37℃溫育2h后的相對酶活)。
圖4顯示青霉素酰化酶PGAPX02的最適反應溫度。
圖5顯示青霉素酰化酶PGAPX02的溫度穩定性(在各溫度條件下溫育2h后的相對酶活)。
圖6顯示20%有機溶劑對青霉素酰化酶PGAPX02的影響。
本發明所述的生物材料,其分類命名為 Achromobacter xylosoxidans PX02,已保藏于中國典型培養物保藏中心(簡稱CCTCC),保藏編號為:CCTCC NO:M 2016392,保藏日期為:2016年7月13日,保藏地址為:中國. 武漢. 武漢大學。
具體實施方式
實施例一
本實施例說明產青霉素酰化酶菌株的篩選程序。
初篩采用如下方法:以高濃度青霉素G為篩選壓力從藥廠周邊土壤等樣品中篩選獲得耐高濃度青霉素微生物。具體培養基配方為:酵母膏5 g/L,蛋白胨 5 g/L,NaCl 2 g/L,苯乙酸2 g/L,青霉素 106U/ml ,pH 7.0。培養溫度為30℃,培養時間為24~48 h,搖床轉速為180 rmp。此方法可篩選到大量耐高濃度青霉素微生物。
對初篩的候選菌株進行復篩,采用如下方法:用50mM磷酸緩沖液(pH7.5)配置2mg/ml底物類似物2-硝基-5-苯乙酰胺苯甲酸2-nitro-5-phenylacetamidobenzoic acid(NIPAB),并加到透明的48孔板中,將候選菌株挑到48孔板中,以不加菌株的為空白對照,37℃反應,可直接觀察反應液的顏色變化,如有產青霉素酰化酶菌株,反應液會變為黃色。將所篩選到的產青霉素酰化酶菌株接種到產酶發酵培養基,具體配方為:酵母膏5 g/L,蛋白胨 5 g/L,NaCl 2 g/L,苯乙酸2 g/L ,pH 7.0。培養溫度為30℃,培養時間為48 h,搖床轉速為180 rmp。發酵結束后,測定其青霉素酰化酶活力,選取酶活力最高的菌株作為產青霉素酰化酶菌株。將該菌保藏于中國典型培養物保藏中心(簡稱CCTCC),保藏編號為:CCTCC NO:M 2016392。
青霉素酰化酶活力檢測方法(以NIPAB為底物)為:取發酵液于12000 rpm、4℃離心10 min,棄上清,菌體用等體積的50mM磷酸緩沖液(pH7.5)重懸,超聲破碎細胞后,12000 rpm,4℃離心10 min,取上清為酶液;將酶液用50mM磷酸緩沖液(pH7.5)稀釋適當倍數,并用相應的緩沖液配置2mg/ml NIPAB底物溶液;反應體系中先加入100 μL酶液,以滅活的酶液為空白對照,再加入100 μL底物溶液,在酶標儀中進行反應,反應溫度為37℃,反應時間為4min,在405 nm波長下檢測反應結束時生產的5-氨基-2-硝基苯甲酸5-Amino-2-nitrobenzoic acid(ANBA)的量。每1個單位(U)青霉素酰化酶定義為,在相應條件下,每分鐘催化水解NIPAB分解產生1 μmol ANBA所需的酶量。
實施例二
本實驗說明青霉素酰化酶PGAPX02編碼基因的分離克隆程序。
采用酚-氯仿法抽提菌體總DNA。選取Achromobacter xylosoxidans A8(NCBI Reference Sequence:NC_014640.1)的全基因測序結果進行分析,獲得編碼無色桿菌來源的青霉素酰化酶的基因,根據該基因序列設計引物:PX02-F1和PX02-R1。
PX02-F1(SEQ ID:3):5′-ATGAAGCAGCAATGGTTGTCGGC-3′;
PX02-R1(SEQ ID:4):5′-TCAGTAGCGCAACGTCTCTACCG-3′。
以木糖氧化無色桿菌Achromobacter xylosoxidans PX02總DNA為模板,以引物PX02-F1和PX02-R1擴增青霉素酰化酶PGAPX02的CDS編碼序列。將擴增到的2500 bp的PCR片段連接到pMD18-T載體,進行序列測定和分析,青霉素酰化酶的基因序列如SEQ ID NO:1所示,氨基酸序列如SEQ ID NO:2所示。木糖氧化無色桿菌Achromobacter xylosoxidans PX02與Achromobacter xylosoxidans A8青霉素酰化酶基因同源性為 90%,成熟肽氨基酸序列同源性為94.7 %。
實施例三
本實驗說明青霉素酰化酶PGAPX02的表達載體的構建及其在大腸桿(E.coli)BL21(DE3)中的表達。
以PGAPX02總DNA為模板,設計引物,如下:
PX02-F2(SEQ ID:5):5′-ATAGCTAGCATGAAGCAGCAATGGTTGTCGGC-3′(下劃線部分為Nhe I酶切位點);
PX02-R2(SEQ ID:6):5′-ATA CTCGAGTCAGTAGCGCAACGTCTCTACCG-3′(下劃線部分為Xho I酶切位點)
以PX02-F2和PX02-F2擴增青霉素酰化酶PGAPX02基因,經純化后用限制性內切酶Nhe I與Xho I進行雙酶切,同時以相同限制性內切酶酶切載體pET28a(購自Novagen公司),將酶切產物進行瓊脂糖凝膠電泳,回收基因片段和載體,并將基因片段與載體通過T4 DNA連接酶連接,經酶切鑒定及PCR篩選出陽性克隆命名為pET28a-pgapx02,從而構建了表達載體pET28a-pgapx02。
將構建的重組質粒pET28a-pgapx02通過熱休克轉化法轉化大腸桿菌(E.coli)BL21(本實驗室保存,特征在于蛋白酶缺陷,T7RNA聚合酶基因)感受態細胞,得到重組菌BL21/ pET28a-pgapx02。同時將pET28a質粒同樣轉化入BL21感受態細胞,命名為BL21/pET28a,用于青霉素酰化酶PGAPX02表達時的對照菌株。將重組菌BL21/ pET28a-pgapx02及BL21/pET28a接種至含100mg mL-1卡那霉素的LB液體培養基,37℃,180rpm培養過夜后,按2%接種量接種至新鮮LB培養基中(含100mg mL-1卡那霉素),37℃,180rpm培養OD600為0.8~1.0時,加入誘導劑IPTG(終濃度0.1mmol L-1),誘導24 h。分別將誘導后的青霉素酰化酶PGAPX02重組菌破碎后離心,分別取上清和沉淀進行SDS-PAGE,以同樣條件下誘導的BL21/pET28a破碎液作為對照,電泳結果如圖1所示,其中第一欄為Marker,第二欄為BL21/pET28a加IPTG誘導后細胞破碎液,第三為重組菌BL21/ pET28a-pgapx02加IPTG誘導表達產物破碎上清,第四為重組菌BL21/pET28a-pgapx02加IPTG誘導表達產物破碎沉淀。由此可見經誘導后的重組菌BL21/pET28a-pgapx02破碎后的上清中在27 kD和65 kD左右處均有一明顯條帶,分別對應α和β亞基。同時分別測定破碎液上清中青霉素酰化酶PGAPX02的酶活,加入誘導劑IPTG誘導后重組菌BL21/pET28a-pgapx02的酶活力為4000U/L,而BL21/pET28a基本不存在青霉素酰化酶PGAPX02酶活。
實施例四
本實驗說明青霉素酰化酶PGAPX02的酶學性質。
青霉素酰化酶PGAPX02最適反應pH和pH穩定性的檢測:分別配制4.0、5.0、6.0、6.5、7.0、7.5、8.0、8.5、9.0、10.0的緩沖液,將酶液與不同pH緩沖液溶解的NIPAB反應液反應,以pH8.0條件下的青霉素酰化酶活力為對照(100%),分別檢測青霉素酰化酶PGAPX02活力,不同pH體系中的酶活如圖2所示。從圖2中可以看出pH 6.5-8.5范圍內具有較高的酶活力,其最適反應pH為8.0。pH穩定性測定:將青霉素酰化酶PGAPX02在不同pH的緩沖溶液中37℃保溫2 h后,以最高酶活為100%,測殘余酶活,從圖3中可以看出該青霉素酰化酶在pH 5.0~10的范圍內具有較高的穩定性,在pH 10的溶液中保溫2h后,仍然保留最高活力的82%。
青霉素酰化酶PGAPX02最適反應溫度和熱穩定性的檢測:最適反應溫度的測定在0.05 M Na2HPO4?12H2O-NaH2PO4?2H2O緩沖體系(pH 7.5)中進行,在不同的溫度下以NIPAB為底物進行酶促反應。結果如圖4所示,該酶的最適反應溫度為55℃。熱穩定性測定:青霉素酰化酶PGAPX02在25 °C,35 °C,45 °C,55 °C,65 °C,75 °C下處理2h以后,以最高酶活為100%,測定殘留酶活。由圖5可以看出該青霉素酰化酶具有較好的熱穩定性,該酶在25℃-45℃范圍內都能保持較高的青霉素酰化酶活力(相對酶活90%以上)。
青霉素酰化酶PGAPX02的有機溶劑耐受性:考察8種有機溶劑對青霉素酰化酶PGAPX02穩定性的影響,分別將8種有機溶劑緩緩加入酶液中至有機溶劑終濃度為20%(V/V),37℃,180 rpm振蕩,定時取樣,以NIPAB為底物檢測青霉素酰化酶活力。以0h不添加有機溶劑的青霉素酰化酶酶活為對照(100%),測定酶的相對酶活。有機溶劑對青霉素酰化酶PGAPX02酶活影響結果如圖6所示。圖6結果表明該酶在不添加有機溶劑時,24h后酶活剩80%,而在聚乙二醇和丙三醇中,于37℃處理24h后,酶活分別為101%和112%,說明這兩種有機溶劑對酶有一定的激活作用,在DMSO中也表現出一定的耐受性,24h后酶活為60%,由實驗結果說明,青霉素酰化酶PGAPX02具有良好的有機溶劑耐受性。
實施例六
本實驗說明青霉素酰化酶PGAPX02在催化合成β-內酰胺類抗生素的應用。
青霉素酰化酶PGAPX02催化合成青霉素G類半合成抗生素:以40%丙三醇-0.05 mM磷酸緩沖液(pH 7.0)為反應介質,反應總體積為2mL,其中含有20 mM 6-氨基青霉烷酸,50 mM對羥基苯甘氨酸甲酯(或苯甘氨酸甲酯),加入青霉素酰化酶PGAPX02酶液(實施例三制備)200 μL(8000U/L),在25℃,轉速200 rpm條件下反應,定時取樣。樣品用溶劑(水︰甲醇︰乙酸=60︰32︰8(V/V))稀釋10倍,采用HPLC進行檢測。結果反應6 h(或4 h),阿莫西林的最大合成率可達65%-70%(或氨芐西林最大合成率可達67%-72%)。
實施例七
本實驗說明青霉素酰化酶PGAPX02在催化合成β-內酰胺類抗生素的應用。
青霉素酰化酶PGAPX02催化合成頭孢菌素類半合成抗生素:以40%丙三醇-0.05 mM磷酸緩沖液(pH 7.0)為反應介質,反應總體積為2 mL,其中含有20 mM 7-氨基去乙酰氧基頭孢烷酸,50 mM對羥基苯甘氨酸甲酯(或苯甘氨酸甲酯),加入青霉素酰化酶PGAPX02酶液(實施例三制備)200 μL(8000U/L),在25℃,轉速200 rpm條件下反應,定時取樣。樣品用溶劑(水︰甲醇︰乙酸=60︰32︰8(V/V))稀釋10倍,采用HPLC進行檢測。結果反應2 h,頭孢氨芐的最大合成率可達72%-76%(或頭孢羥氨芐最大合成率可達75%-80%)。
以40%丙三醇-0.05 mM磷酸緩沖液(pH 7.0)為反應介質,反應總體積為2 mL,其中含有20 mM 7-氨基-3- (2-氨基-1,3,4-噻二唑)硫甲基頭孢烷酸,50 mM四氮唑乙酸甲酯,加入青霉素酰化酶PGAPX02酶液(實施例三制備)200 μL(8000U/L),在25℃,轉速200 rpm條件下反應,定時取樣。樣品用溶劑(水︰甲醇︰乙酸=60︰32︰8(V/V))稀釋10倍,采用HPLC進行檢測。結果反應2 h,頭孢替唑的最大合成率可達42%-47%。
實施例八
本實驗說明擴大反應體系青霉素酰化酶PGAPX02催化合成頭孢替唑。
以40%丙三醇-0.05 mM磷酸緩沖液(pH 7.0)為反應介質,反應總體積為20 mL,其中含有100 mM 7-氨基-3- (2-氨基-1,3,4-噻二唑)硫甲基頭孢烷酸,250 mM四氮唑乙酸甲酯,加入青霉素酰化酶PGAPX02酶液(實施例三制備)2 mL(8000U/L),在25℃,轉速400 rpm條件下反應,自動電位滴定儀檢測并用10%的氨水滴定,維持pH 7.0,定時取樣。樣品用溶劑(水︰甲醇︰乙酸=60︰32︰8(V/V))稀釋10倍,采用HPLC進行檢測。結果反應6h,頭孢替唑的合成率最高可達63%-69%。
結果表明青霉素酰化酶PGAPX02具有良好的合成青霉素G類以及頭孢菌素類半合成抗生素的能力,表明該酶將來在合成多種新型β-內酰胺類抗生素中具有巨大的潛力,在生物制藥工業中具有廣闊的應用前景。
序列表
<110> 南京工業大學
<120> 一株青霉素酰化酶及其編碼基因、產生菌和應用
<130> xb16092301
<160> 2
<170> PatentIn version 3.3
<210> 1
<211> 2583
<212> DNA
<213> Achromobacter xylosoxidans PX02
<400> 1
atgaagcagc aatggttgtc ggccgccatc ctggcggcct gtgcggccgt gggcgctcat 60
gcccaggccc aggaaatccc gcagaagact tcgccgcagg ccggtgcctc ggcgcgtccc 120
gctggagcca ccagcggaca ggtcacgata cggcgcgatg gttatggcat gccgcacgtg 180
tacgccaaca ccgtgtatgg cattttctat ggctatggct acgcggtggc gcaggatcgc 240
ccgttccaga tggagatggc gcggcgcagc acccagggcc gcgtggctga agtgcttgga 300
cagcccatgg tggcgttcga caagtccatc cggggaaatt tctcgccaga gcgcatccag 360
cgccagctcg cggcgttgcc ggcatcggag cgccagatcc tggacggcta cgcggccggc 420
atgaatgcct ggatcgcgcg ggtgcgcgcc gagcctggcg gactgatgcc caaggaattc 480
aacgacctgc gcttccagcc ggcggactgg acggcctacg acgtggcgat ggtgttcgtg 540
ggcaccatgg ccaaccgctt ctccgacgcc aatagcgaga tcgacaatct ggcgctgctg 600
accgcgctga aggacaagca cggcgatgag cgcgccatgc agatcttcaa ccagctgcgc 660
tggatgaccg acagccgcgc gcccaccacc gtgccggagg aagagggggt gtaccaaccg 720
gacgccgctc gtccttccgc caggctctcc tatgcgttgc cgcgctacga gggcacgccg 780
cccatgctgg aacgcgtggc gcgcgatccg caaacccgcg gtgtgctgga cgaagcgcct 840
gccgcggtgc cggcgcgact gctggcccag tttgcggaat cggggcaacc gggcatcgcc 900
ggctttccca cgaccagcaa catgtggatc gtgggccgcg accatgccaa ggacgcgcgc 960
gccatcctgc tgaacggacc gcagttcggc tggtggaacc cggcgtatac ctacgggatc 1020
ggcttgcacg gggccggatt cgacgtggtc ggcaatacgc cgtttgccta tcccagcatc 1080
ttgttcggcc acaatgcgca cgtgacctgg ggctcgactg ccggctttgg cgatgacgtg 1140
gacatctacg ccgaaaagct ggatcccaac gaccgcaccc gctatttcca tgatggcgtc 1200
tggaagacga tggaaaagcg caccgagctc atcgaggtga aggacgcgca gccggtggtg 1260
atggatgtct accgcaccgt gcacggcatc gtcacgaagt tcgacgacaa gcagcgcgtg 1320
gcctacgcca aggcgcgcgc ctgggagggc tatgagctgc aatcgctgat ggcctggacc 1380
cacaaggcgc agtcccgcaa ctgggaccag tggaaacagc aggccgcgcg ccacgccctg 1440
accatcaact ggtattacac ggacgacaag ggcaatatcg gctacgcgca tacggggttc 1500
taccccaaac gccgtccggg ccacgatccg cgcctgcccg tgcctggcac cggcgagatg 1560
gattgggacg gcatactgcc tttctcgacc aacccgcagg tgtacaaccc gcgccagggt 1620
tttatcgcca actggaacaa ccagcccatg cgcggctatc cctccaccga ccttttcgcc 1680
atcgtgtggg gccaggcgga tcgctatgcc gaaatcgaga cgcgcctgaa ggccatgaca 1740
gccaatggcg gcaaggtcag cgcgcaacag atgtgggacc tgatccgcac gaccagctat 1800
gccgacgtga accgccgcca tttcctgccg ttcctgcagc aggcggtgca agggctgccc 1860
gcggatgacg cgcgggcgcg gctggttgcc gggctggcat cgtgggacgg catgggtacc 1920
agcgacaagc aaccgggcta ctacgaccac acgggccccg cggtgatgga cgcctggctg 1980
cgcgccatgc tcaagcgcgc gctggccgac gagatgcccg ccgacttctt caagtggtac 2040
agcgccaccg gttatccgac gcaggcggcg cctgctaccg ggtcggtcaa cctgacggtg 2100
ggggtgaagg tgttgttcaa tgcgctggcg ggtcgcgatg cgggcgtgcc acagcagtac 2160
gatttcttca acggccaacg accccaggat gtaacgctgg cggccctgga cgacgcgctg 2220
gcggctttgc gcaaggccta tggcgaggat cccgcccagt ggcgcatacc tgcgcctccg 2280
atggtgttcg cgcccaagaa cttcctgggc gtgccgcagg cggacgacaa ggccgtgttg 2340
agcttcccgg ccacccagaa ccgcggcacc gagaacaaca tgacggtgtt cgatggcaag 2400
ggcgtgcgcg cggtggacgt ggtggcgccg ggacaaagcg gttttgtcgc cccggacggc 2460
accccgtcac cccacgcgcg cgaccagttc gacctttaca ccagtttcgg cagcaagcgg 2520
gtctggttca cggatgcgga agtgcgcgcc cacgctaagt cggtagagac gttgcgctac 2580
tga 2583
<210> 2
<211>860
<212>PRT
<213> Achromobacter xylosoxidans PX02
<400> 2
Met Lys Gln Gln Trp Leu Ser Ala Ala Ile Leu Ala Ala Cys Ala Ala
1 5 10 15
Val Gly Ala His Ala Gln Ala Gln Glu Ile Pro Gln Lys Thr Ser Pro
20 25 30
Gln Ala Gly Ala Ser Ala Arg Pro Ala Gly Ala Thr Ser Gly Gln Val
35 40 45
Thr Ile Arg Arg Asp Gly Tyr Gly Met Pro His Val Tyr Ala Asn Thr
50 55 60
Val Tyr Gly Ile Phe Tyr Gly Tyr Gly Tyr Ala Val Ala Gln Asp Arg
65 70 75 80
Pro Phe Gln Met Glu Met Ala Arg Arg Ser Thr Gln Gly Arg Val Ala
85 90 95
Glu Val Leu Gly Gln Pro Met Val Ala Phe Asp Lys Ser Ile Arg Gly
100 105 110
Asn Phe Ser Pro Glu Arg Ile Gln Arg Gln Leu Ala Ala Leu Pro Ala
115 120 125
Ser Glu Arg Gln Ile Leu Asp Gly Tyr Ala Ala Gly Met Asn Ala Trp
130 135 140
Ile Ala Arg Val Arg Ala Glu Pro Gly Gly Leu Met Pro Lys Glu Phe
145 150 155 160
Asn Asp Leu Arg Phe Gln Pro Ala Asp Trp Thr Ala Tyr Asp Val Ala
165 170 175
Met Val Phe Val Gly Thr Met Ala Asn Arg Phe Ser Asp Ala Asn Ser
180 185 190
Glu Ile Asp Asn Leu Ala Leu Leu Thr Ala Leu Lys Asp Lys His Gly
195 200 205
Asp Glu Arg Ala Met Gln Ile Phe Asn Gln Leu Arg Trp Met Thr Asp
210 215 220
Ser Arg Ala Pro Thr Thr Val Pro Glu Glu Glu Gly Val Tyr Gln Pro
225 230 235 240
Asp Ala Ala Arg Pro Ser Ala Arg Leu Ser Tyr Ala Leu Pro Arg Tyr
245 250 255
Glu Gly Thr Pro Pro Met Leu Glu Arg Val Ala Arg Asp Pro Gln Thr
260 265 270
Arg Gly Val Leu Asp Glu Ala Pro Ala Ala Val Pro Ala Arg Leu Leu
275 280 285
Ala Gln Phe Ala Glu Ser Gly Gln Pro Gly Ile Ala Gly Phe Pro Thr
290 295 300
Thr Ser Asn Met Trp Ile Val Gly Arg Asp His Ala Lys Asp Ala Arg
305 310 315 320
Ala Ile Leu Leu Asn Gly Pro Gln Phe Gly Trp Trp Asn Pro Ala Tyr
325 330 335
Thr Tyr Gly Ile Gly Leu His Gly Ala Gly Phe Asp Val Val Gly Asn
340 345 350
Thr Pro Phe Ala Tyr Pro Ser Ile Leu Phe Gly His Asn Ala His Val
355 360 365
Thr Trp Gly Ser Thr Ala Gly Phe Gly Asp Asp Val Asp Ile Tyr Ala
370 375 380
Glu Lys Leu Asp Pro Asn Asp Arg Thr Arg Tyr Phe His Asp Gly Val
385 390 395 400
Trp Lys Thr Met Glu Lys Arg Thr Glu Leu Ile Glu Val Lys Asp Ala
405 410 415
Gln Pro Val Val Met Asp Val Tyr Arg Thr Val His Gly Ile Val Thr
420 425 430
Lys Phe Asp Asp Lys Gln Arg Val Ala Tyr Ala Lys Ala Arg Ala Trp
435 440 445
Glu Gly Tyr Glu Leu Gln Ser Leu Met Ala Trp Thr His Lys Ala Gln
450 455 460
Ser Arg Asn Trp Asp Gln Trp Lys Gln Gln Ala Ala Arg His Ala Leu
465 470 475 480
Thr Ile Asn Trp Tyr Tyr Thr Asp Asp Lys Gly Asn Ile Gly Tyr Ala
485 490 495
His Thr Gly Phe Tyr Pro Lys Arg Arg Pro Gly His Asp Pro Arg Leu
500 505 510
Pro Val Pro Gly Thr Gly Glu Met Asp Trp Asp Gly Ile Leu Pro Phe
515 520 525
Ser Thr Asn Pro Gln Val Tyr Asn Pro Arg Gln Gly Phe Ile Ala Asn
530 535 540
Trp Asn Asn Gln Pro Met Arg Gly Tyr Pro Ser Thr Asp Leu Phe Ala
545 550 555 560
Ile Val Trp Gly Gln Ala Asp Arg Tyr Ala Glu Ile Glu Thr Arg Leu
565 570 575
Lys Ala Met Thr Ala Asn Gly Gly Lys Val Ser Ala Gln Gln Met Trp
580 585 590
Asp Leu Ile Arg Thr Thr Ser Tyr Ala Asp Val Asn Arg Arg His Phe
595 600 605
Leu Pro Phe Leu Gln Gln Ala Val Gln Gly Leu Pro Ala Asp Asp Ala
610 615 620
Arg Ala Arg Leu Val Ala Gly Leu Ala Ser Trp Asp Gly Met Gly Thr
625 630 635 640
Ser Asp Lys Gln Pro Gly Tyr Tyr Asp His Thr Gly Pro Ala Val Met
645 650 655
Asp Ala Trp Leu Arg Ala Met Leu Lys Arg Ala Leu Ala Asp Glu Met
660 665 670
Pro Ala Asp Phe Phe Lys Trp Tyr Ser Ala Thr Gly Tyr Pro Thr Gln
675 680 685
Ala Ala Pro Ala Thr Gly Ser Val Asn Leu Thr Val Gly Val Lys Val
690 695 700
Leu Phe Asn Ala Leu Ala Gly Arg Asp Ala Gly Val Pro Gln Gln Tyr
705 710 715 720
Asp Phe Phe Asn Gly Gln Arg Pro Gln Asp Val Thr Leu Ala Ala Leu
725 730 735
Asp Asp Ala Leu Ala Ala Leu Arg Lys Ala Tyr Gly Glu Asp Pro Ala
740 745 750
Gln Trp Arg Ile Pro Ala Pro Pro Met Val Phe Ala Pro Lys Asn Phe
755 760 765
Leu Gly Val Pro Gln Ala Asp Asp Lys Ala Val Leu Ser Phe Pro Ala
770 775 780
Thr Gln Asn Arg Gly Thr Glu Asn Asn Met Thr Val Phe Asp Gly Lys
785 790 795 800
Gly Val Arg Ala Val Asp Val Val Ala Pro Gly Gln Ser Gly Phe Val
805 810 815
Ala Pro Asp Gly Thr Pro Ser Pro His Ala Arg Asp Gln Phe Asp Leu
820 825 830
Tyr Thr Ser Phe Gly Ser Lys Arg Val Trp Phe Thr Asp Ala Glu Val
835 840 845
Arg Ala His Ala Lys Ser Val Glu Thr Leu Arg Tyr
850 855 860
- 還沒有人留言評論。精彩留言會獲得點贊!