用于鑒定刀鱭和短頜鱭的引物和分子生物學方法與流程
本發明涉及生物
技術領域:
,特別涉及一種用于鑒定刀鱭和短頜鱭的引物和分子生物學方法。
背景技術:
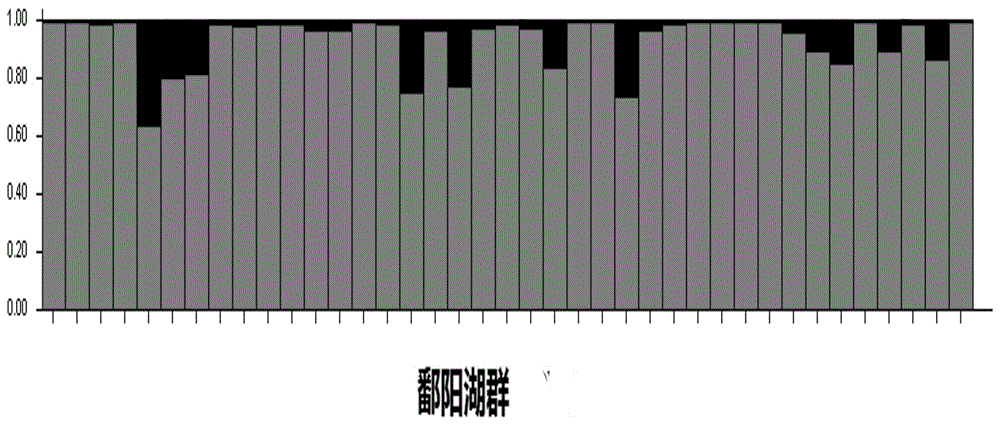
:長江刀鱭是長江中一種重要的漁業資源,也是長江特有的一種名貴水產品。而在長江中,同時還分布另一種與之形態極為類似的物種短頜鱭。短頜鱭的口感和價值,與刀鱭相差較大。市面上常常利用短頜鱭個體來冒充刀鱭出售,而由于兩者主要區別的下頜部分,差距較小難以辨別,而且往往由于刺網的捕撈破損而更加難以甄別;另一方面,由于目前刀鱭尚未實現人工繁育,養殖需要依賴長江中撈取的苗種,而刀鱭和短頜鱭的苗種常常出現在同一區域混雜生存而難以區分,常常要等養到較大個體才可以區分剔除,增加了養殖的成本。現有技術中,通常采用線粒體DNA技術對刀鱭、鳳鱭和短頜鱭基因甄別,但是此方法主要存在以下缺陷:首選經遺傳學調查顯示,刀鱭和鳳鱭差異較大,較易使用線粒體基因序列加以區別,而刀鱭和短頜鱭是姐妹種,分化時間短,線粒體差異不明顯,甚至還有一定的遺傳漸滲現象,因此,不適合使用線粒體基因組序列對這兩者進行辨別。其次,線粒體是母系遺傳,其序列只能反應其母系祖先的特征,而長江中由于這兩者具有共分布區和遺傳漸滲現象,有可能使得鑒定產生錯誤。同時,一些水系的短頜鱭的遺傳背景與長江刀鱭更加接近,因此僅適用線粒體基因序列分析,很難區別其物種歸屬。另一方面,線粒體序列測定如序列數據較少,信息量少,如序列數據較多,信息量大,費用也較大。為了解決以上存在的問題,人們一直在尋求一種理想的技術解決方案。技術實現要素:有鑒于此,本發明的目的在于提供一種鑒別效率高、敏感度高、方便快捷且、費用低廉的用于鑒定刀鱭和短頜鱭的引物和分子生物學方法,以解決上述問題。為了實現上述目的,本發明所采用的技術方案是:一種引物組合物,它包含擴增引物組和延伸引物組;所述擴增引物組的核酸序列如SEQIDNO:1-16所示,所述延伸引物組的核酸序列如SEQIDNO:17-24所示。本發明還提供一種所述的引物組合物的應用,所述引物組合物用于鑒定刀鱭和短頜鱭。本發明還提供一種構建包括待測刀鱭和短頜鱭的SNP位點區域核酸測序文庫的方法,包括以下步驟:利用擴增引物組對包含SNP位點區域的待測刀鱭和短頜鱭核酸樣本進行第一PCR擴增,獲得第一擴增產物,分別利用磷酸化酶和外切酶對所述第一擴增產物進行純化;利用延伸引物組對純化后的所述第一擴增產物進行延伸反應,獲得延伸產物,采用磷酸化酶對所述延伸產物進行純化,純化后的所述延伸產物構成所述待測刀鱭和短頜鱭SNP位點區域核酸測序文庫;其中,所述擴增引物組的核酸序列如SEQIDNO:1-16所示,所述延伸引物組的核酸序列如SEQIDNO:17-24所示。基于上述,制得所述包含SNP位點區域的待測刀鱭和短頜鱭核酸樣本步驟包括:首先取待鑒定樣品的肌肉或者活體尾鰭尖部組織;然后采用酚氯仿法、高鹽法或層析柱法提取待測刀鱭和短頜鱭樣品的DNA。基于上述,構建包括待測刀鱭和短頜鱭的SNP位點區域核酸測序文庫的方法中,按照20微升反應體系計算,所述第一PCR擴增的反應體系為:試劑體積(微升)PrimerMix4MgCl1.6dNTPMix0.4ExTaq酶10待測刀鱭和短頜鱭DNA2ddH2O補至20所述第一PCR擴增的反應程序為:基于上述,所述構建包括待測刀鱭和短頜鱭的SNP位點區域核酸測序文庫的方法中,按照10微升反應體系計算,所述延伸反應的反應體系為:試劑體積(微升)SNaPshotMultiplexKit5多重第一擴增產物2延伸引物組1ddH2O補至10所述延伸反應的反應程序為:本發明還提供一種利用上述引物組合鑒定刀鱭和短頜鱭的分子生物學方法,包括以下步驟:首先取待測刀鱭和短頜鱭樣品的肌肉或者活體尾鰭尖部組織,然后采用酚氯仿法、高鹽法或層析柱法提取待測刀鱭和短頜鱭樣品的DNA;然后利用擴增引物組對包含SNP位點區域的待測刀鱭和短頜鱭核酸樣本進行第一PCR擴增,獲得第一擴增產物;利用磷酸化酶和外切酶對所述第一擴增產物進行純化;利用延伸引物組對純化后的所述第一擴增產物進行延伸反應,獲得延伸產物;采用磷酸化酶對所述延伸產物進行純化;最后對純化后的延伸產物進行測序,獲得測序結果;基于所述測序結果,確定所述待測刀鱭和短頜鱭SNP位點區域核酸序列;基于所述刀鱭和短頜鱭SNP位點區域核酸序列,利用Structure軟件,繪制群體遺傳差異的柱形圖和個體遺傳差異的柱形圖,利用該分析判斷所述待測刀鱭和短頜鱭物種種類;其中,所述擴增引物組的核酸序列如SEQIDNO:1-16所示,所述延伸引物組的核酸序列如SEQIDNO:17-24所示。基于上述,所述的利用上述引物組合鑒定刀鱭和短頜鱭的分子生物學方法中,分別采用ABI3730xl及GeneMapper4.0對來所述待測刀鱭和短頜鱭SNP位點區域核酸測序文庫進行測序和分析統計。本發明相對現有技術具有突出的實質性特點和顯著的進步,具體的說,僅選取8個SNP位點進行樣品鑒定即可有效的區分刀鱭和短頜鱭物種,達到物種鑒定的良好效果。同時由于SNP分型是按照反應孔計費,而每個孔同時容納的檢測位點的多少是根據位點的相互位置關系來確定的,本發明選取的8個位點能夠滿足在一個反應孔完成SNP分型的技術要求,每個孔即可鑒定1個樣品,可以實現單次96-384個樣品的反應,鑒定效率高;并且本發明的鑒定結果的軟件可視化分析,方便快捷,結果直觀、易讀。同時,利用本發明提供的分子生物學方法可降低鑒定刀鱭和短頜鱭的鑒定費用。附圖說明圖1為實施例1從選自洞庭湖的39個樣品中任選的1個樣本測試的8個SNP位點在分型峰圖中的位置分布。圖2為實施例1中利用結構分析軟件得到的選自洞庭湖中39個樣本個體遺傳組成柱狀圖。圖3為實施例2從選自潘陽湖的39個樣品中任選的1個樣本測試的8個SNP位點在分型峰圖中的位置分布。圖4為實施例2中利用結構分析軟件得到的選自潘陽湖中39個樣本個體遺傳組成柱狀圖。圖5為實施例3中選自蕪湖9個樣本選中任選1個樣本測試的8個SNP位點在分型峰圖中的位置分布。圖6為實施例3中利用結構分析軟件得到的9個選自蕪湖樣本個體遺傳組成柱狀圖。圖7為實施例4中選自太湖40個樣本選中任選1個樣本測試的8個SNP位點在分型峰圖中的位置分布。圖8為實施例4中利用結構分析軟件得到的40個選自太湖樣本個體遺傳組成柱狀圖。圖9-圖10為實施例5中選自某養殖場20個樣本選中任選2個樣本測試的8個SNP位點在分型峰圖中的位置分布。圖11為實施例5中利用結構分析軟件得到的選自某養殖場20個樣本個體遺傳組成柱狀圖。圖12-圖13為實施例6中選自靖江19個樣本選中任選2個樣本測試的8個SNP位點在分型峰圖中的位置分布。圖14為實施例3中利用結構分析軟件得到的19個選自靖江樣本個體遺傳組成柱狀圖。具體實施方式下面通過具體實施方式,對本發明的技術方案做進一步的詳細描述。本發明提供的鑒定刀鱭和短頜鱭的分子生物學方法,具體包括以下步驟:1、樣本DNA的提取(1)分別取新鮮或冰凍的不同種群的待測刀鱭和短頜鱭肌肉或者活體的尾鰭尖部組織0.1g,剪碎并置于玻璃勻漿器中,向其中加入1mL的細胞裂解緩沖液勻漿至組織塊消失,然后將玻璃勻漿器中的液體置于1.5mL的離心管中,并向離心管加入蛋白酶,混均。在65oC恒溫水浴鍋中水浴30min,也可轉入37oC水浴12-24小時,間歇振蕩離心管數次。然后將離心管置于臺式離心機以12000rpm/min離心5分鐘,取上清液置于第二離心管中。(2)向第二離心管中加入2倍上清液體積的異丙醇,倒轉混均后,將生成的絲狀物挑出并晾干,然后用200微升的TE試劑重新溶解所述絲狀物。(3)向溶解有絲狀物的TE試劑中加入等體積的酚-氯仿-異戊醇并振蕩混均,然后以12000rpm/min轉速離心5分鐘,取上清液置于第三離心管中。(4)向第三離心管中加入等體積的氯仿-異戊醇,并振蕩混均,然后以12000rpm/min轉速離心5分鐘,取上清液置于第四離心管中。(5)向第四離心管中加入一半體積的7.5mol/L的乙酸氨和2倍體積的無水乙醇,混勻后室溫沉淀2min,然后以12000rpm/min轉速離心10分鐘,棄上清后,向第四離心管中加入200微升的TE試劑重新溶解沉淀物,即得到刀鱭或短頜鱭的DNA提取液,然后將其置于4oC保存備用。2、多重PCR擴增及延伸反應(1)引物設計基于篩選的刀鱭和短頜鱭8個SNP位點基因設計能與之配對的擴增引物SEQIDNO:1-16和延伸引物SEQIDNO:17-24;(2)多重PCR擴增反應首先利用上述擴增引物對DNA提取液進行擴增,獲得PCR產物;然后取15微升的PCR產物,并向其中加入5USAP酶和2UExonucleaseI酶,并將其置于37oC水浴條件下1小時,然后在75oC滅活15分鐘,對PCR產物進行純化處理。具體地,按照20微升反應體系計算,所述第一PCR擴增的反應體系為:試劑體積(微升)PrimerMix4MgCl1.6dNTPMix,0.4ExTaq10待測刀鱭和短頜鱭DNA2ddH2O補至20所述第一PCR擴增的反應程序為:(3)SNaPshot多重單堿基延伸反應首先采用本發明設計的延伸引物組對純化后的PCR產物進行多重單堿基延伸反應,獲得延伸產物;然后取10微升的延伸產物,并向其中加入1USAP酶,并將其置于37oC水浴條件下1小時,然后在85oC滅活15分鐘,對延伸產物進行純化處理。具體地,按照10微升反應體系計算,所述延伸反應的反應體系為:試劑體積(微升)SNaPshotMultiplexKit(ABI)5多重第一擴增產物2延伸引物組1ddH2O補至10所述延伸反應的反應程序為:3、延伸產物核酸測序及群體鑒別取1微升純化后的延伸產物置于測序容器中,然后向其中加入0.5微升的Liz120SIZESTANDARD試劑和8.5微升的Hi-Di試劑并進行混勻,并置于95oC溫度下變性5分鐘,變性后采用ABI3730XL測序儀進行測序。經ABI3730XL測序儀上收集的原始數據用GeneMapper4.0軟件進行分析統計。最后,設置分群參數K=2,利用Structure2.3.2軟件繪制群體遺傳差異的柱形圖和個體遺傳差異的柱形圖,利用群體遺傳差異的柱形圖和個體遺傳差異的柱形圖分析判斷樣本的物種歸屬。實施例1本實施例本根據發明提供的鑒定刀鱭和短頜鱭的分子生物學方法的具體步驟,對選自洞庭湖中39個樣本進行分子生物學鑒定,具體鑒定結果如下:利用ABI3730XL測序平臺,從選自洞庭湖中39個樣本中任選的1個樣本測試的8個SNP位點在分型峰圖中的位置分布如圖1所示。選自洞庭湖中39個樣本的遺傳組成柱狀圖如圖2所示,柱狀圖中,黑色組分占大多數的個體即為刀鱭,灰色組分占大多數的個體為短頜鱭。鑒定結果顯示,選自洞庭湖中39個樣本個體中全部為短頜鱭。實施例2本實施例根據本發明提供的鑒定刀鱭和短頜鱭的分子生物學方法,利用ABI3730XL測序平臺,對選自潘陽湖中39個樣本進行分子生物學鑒定具體操作步驟同實施例1中的測序和鑒定步驟。從選自潘陽湖的39個樣品中任選的1個樣本測試的8個SNP位點在分型峰圖中的位置分布如圖3所示。選自潘陽湖中39個樣本的遺傳組成柱狀圖如圖4所示,柱狀圖中,黑色組分占大多數的個體即為刀鱭,灰色組分占大多數的個體為短頜鱭。鑒定結果顯示,選自潘陽湖中39個樣本個體中全部為短頜鱭。實施例3本實施例根據本發明提供的鑒定刀鱭和短頜鱭的分子生物學方法,利用ABI3730XL測序平臺,對選自蕪湖樣的9個樣本進行分子生物學鑒定具體操作步驟同實施例1中的測序和鑒定步驟。選自蕪湖樣的9個樣本中任選1個樣本測試的8個SNP位點在分型峰圖中的位置分布如圖5所示。選自蕪湖樣的9個樣本的遺傳組成柱狀圖如圖6所示,柱狀圖中,淺白色組分占大多數的個體即為刀鱭,灰色組分占大多數的個體為短頜鱭。鑒定結果顯示,選自蕪湖樣的9個樣本個體中全部為刀鱭。實施例4本實施例根據本發明提供的鑒定刀鱭和短頜鱭的分子生物學方法,利用ABI3730XL測序平臺,對選自太湖40個樣本進行分子生物學鑒定具體操作步驟同實施例1中的測序和鑒定步驟。選自太湖40個樣本中任選1個樣本測試的8個SNP位點在分型峰圖中的位置分布如圖7所示。選自太湖40個樣本的遺傳組成柱狀圖如圖8所示,柱狀圖中,淺白色組分占大多數的個體即為刀鱭,灰色組分占大多數的個體為短頜鱭。鑒定結果顯示,選自太湖40個樣本個體中全部為刀鱭。實施例5本實施例根據本發明提供的鑒定刀鱭和短頜鱭的分子生物學方法,利用ABI3730XL測序平臺,對選自某養殖場20個樣本進行分子生物學鑒定具體操作步驟同實施例1中的測序和鑒定步驟。選自某養殖場20個樣本任選2個樣本測試的8個SNP位點在分型峰圖中的位置分布如圖9和圖10所示。選自某養殖場20個樣本的遺傳組成柱狀圖如圖11所示,柱狀圖中,淺白色組分占大多數的個體即為刀鱭,灰色組分占大多數的個體為短頜鱭。鑒定結果顯示,選自某養殖場20個樣本存在兩個物種的共分布區,分析認為養殖群體的種源由于是從長江下游一帶撈苗,且撈苗活動并非一次,因此兩個物種在群體里混雜程度較高。實施例6本實施例根據本發明提供的鑒定刀鱭和短頜鱭的分子生物學方法,利用ABI3730XL測序平臺,對選自某養殖場20個樣本進行分子生物學鑒定具體操作步驟同實施例1中的測序和鑒定步驟。選自靖江19個樣本任選2個樣本測試的8個SNP位點在分型峰圖中的位置分布如圖12和圖13所示。選自靖江19個樣本的遺傳組成柱狀圖如圖14所示,柱狀圖中,淺白色組分占大多數的個體即為刀鱭,灰色組分占大多數的個體為短頜鱭。鑒定結果顯示,選自靖江19個樣本存在兩個物種的共分布區,因此短頜鱭群體里混雜少量刀鱭。最后應當說明的是:以上實施例僅用以說明本發明的技術方案而非對其限制;盡管參照較佳實施例對本發明進行了詳細的說明,所屬領域的普通技術人員應當理解:依然可以對本發明的具體實施方式進行修改或者對部分技術特征進行等同替換;而不脫離本發明技術方案的精神,其均應涵蓋在本發明請求保護的技術方案范圍當中。當前第1頁1 2 3
相關技術
網友詢問留言
已有0條留言
- 還沒有人留言評論。精彩留言會獲得點贊!
1
- 一種基于熒光PCR檢測ESR1基因突變的引物及探針的制造方法與工藝
- 一種用于檢測乳制品中熒光假單胞菌的分子標記及其應用的制造方法與工藝
- 一種用于檢測水產品中副溶血弧菌的分子標記及其應用的制造方法與工藝
- 一種用于檢測草莓灰霉病菌的分子標記及其應用的制造方法與工藝
- 一種用于檢測B族鏈球菌四環素耐藥基因的分子標記及其應用的制造方法與工藝
- 一種用于檢測糞腸球菌慶大霉素耐藥基因的分子標記及其應用的制造方法與工藝
- 一種用于檢測奶牛乳腺炎性金黃色葡萄球菌青霉素耐藥基因的分子標記及其應用的制造方法與工藝
- 與甘藍葉片表皮毛有無緊密相關的分子標記及其應用的制造方法與工藝
- 檢測黃杉大小蠹的引物、試劑盒及檢測方法與流程
- 一種用于紫斑牡丹與中原牡丹的分子鑒定方法與流程