一種在總狀毛霉休眠孢子中表達蛋白質的方法與流程
本發明屬于生物技術領域,具體涉及一種在總狀毛霉休眠孢子中表達蛋白質的方法。
背景技術:
分子生物學的中心法則是指遺傳信息從DNA傳遞給RNA,再從RNA傳遞給蛋白質,即完成遺傳信息的轉錄和翻譯的過程。現代基因工程中,常常使用宿主來表達外源蛋白,常用的做法就是構建質粒載體,把外源蛋白的編碼DNA序列放在一個強啟動子后面,導入宿主細胞后,編碼DNA序列被轉錄成mRNA序列(單鏈RNA),mRNA序列又被細胞內的翻譯機器表達成蛋白質分子,完成外源蛋白表達的全過程。
DNA不論是作為游離于宿主基因組之外的質粒,還是插入到宿主基因組之中,都可以穩定地復制自身,并傳遞給子代。也就是說,DNA的信息可以遺傳。同時,DNA可以源源不斷地轉錄出新生RNA,并翻譯成蛋白質。
RNA由核糖核苷酸經磷酸二酯鍵縮合而成長鏈狀分子。一個核糖核苷酸分子由磷酸,核糖和堿基構成。RNA的堿基主要有4種,即A(腺嘌呤)、G(鳥嘌呤)、C(胞嘧啶)、U(尿嘧啶)。RNA作為單鏈分子,與雙鏈結構的DNA相比,RNA很不穩定,半衰期很短,而且在自然界,RNA酶廣泛存在,RNA非常容易被降解。RNA具有以下眾所周知的特征:游離在細胞質的RNA不會遺傳;RNA半衰期很短,不能長期存在;RNA不會插入到宿主的基因組DNA中。
由于半衰期短,因此,一個RNA分子存在于細胞內的時長,也很短暫,該RNA只能在這個短暫的時間內,與核糖體相互作用,表達蛋白質分子。而被表達出來的蛋白質分子,也是有壽命的,一個蛋白質分子在細胞內存在多久,也可以用半衰期指標來衡量。
在傳統的基因工程技術中,如果要在一個宿主細胞內部表達外源的蛋白質,常用的做法是把蛋白質的編碼基因構建在DNA載體上,把DNA載體轉入宿主細胞內部,DNA載體源源不斷地轉錄出編碼蛋白質的RNA,RNA不斷翻譯出目的蛋白,實現了在細胞內的蛋白表達。
如果不借助DNA載體,在體外制備編碼蛋白的單鏈RNA分子,僅僅把編碼蛋白質的外源單鏈RNA導入到宿主細胞內部,只要細胞內沒有對應的DNA源源不斷地轉錄出新的該RNA,那么,這個RNA與其所表達出的蛋白質分子,只能在特定的時間窗口存在。換言之,這個RNA與其所表達出的蛋白質分子,只能在細胞生命周期中的某一個時間段存在。
因此,這個特性有個很重要的作用,可以在特定的時間周期窗口,瞬時表達某個蛋白或者RNA,可以實現表達蛋白質而不影響或者盡可能少影響細胞的正常生理周期和遺傳信息。比如:可以在細胞生命活動的某個時間節點,轉入外源RNA,表達某個細胞因子,調控細胞的生命周期;可以把TALENs、ZFNs和CRISPR/Cas9等Genome Editing的工具酶蛋白分子,用外源單鏈RNA在宿主細胞內做表達,避免使用質粒或其它DNA形式的載體時,將DNA序列引入細胞內部,并造成潛在的外源DNA與宿主染色體相互作用(例如同源重組),影響細胞的遺傳,等等。這個特性在基因工程領域,具有重要價值。
在哺乳動物細胞實驗中,已經有先例,可以把外源單鏈RNA直接轉入哺乳動物細胞中。主要方法包括:1,使用諸如Lipo等試劑的化學法,Lipo等化學試劑可以與哺乳動物細胞的細胞膜發生相互作用,把外源RNA導入細胞內部。2,傳統的電轉化法,比如方波電擊,這是一種用電脈沖短暫作用于接觸外源RNA的哺乳動物細胞,使外源RNA進入細胞。
總狀毛霉,屬于毛霉目,毛霉科,可以生產蛋白酶、羥基黃體酮、殼聚糖等各種具有經濟價值的產物,是一種重要的工業發酵菌種。對其進行科學研究和基因工程改造,都涉及到在其細胞內表達蛋白質,但是在這種真菌未萌發孢子中表達蛋白質非常困難。
真菌孢子是真菌的主要繁殖器官,孢子呈休眠狀態且可以生存很長時間。孢子在適宜的外界條件下,能夠蘇醒并萌發,形成菌絲而進行分裂繁殖。最為重要的是,很多類型真菌的孢子,是天然的單倍體,孢子是基因工程領域良好的起始生物材料。
但是,由于孢子一般處于休眠狀態,其細胞壁非常厚實,休眠孢子的細胞壁、細胞膜的狀態,與萌發孢子、與菌絲體是不同的。且休眠孢子細胞內的生命活動也處于最不旺盛的狀態,因此,本領域公知,休眠孢子的細胞通透性不如萌發孢子和菌絲,休眠孢子幾乎不與外界交流物質,閉關鎖國,一般處于睡眠狀態。而萌發孢子或菌絲體,需要從外界吸收各種營養分子供給生命活動,因此細胞壁和細胞膜的通透性大于休眠孢子。所以,非常難以將外源RNA分子直接導入到睡眠孢子的內部。目前也尚未有任何方法可以做到直接把外源RNA分子在無介質介導的情況下,導入休眠的(未萌發)真菌孢子內部。這也是本行業內一直沒有攻克的技術難題。
目前尚未有把外源單鏈RNA導入總狀毛霉細胞內部的報道,更沒有把外源RNA導入總狀毛霉孢子(無論是萌發孢子,還是未萌發孢子)的報道。目前用基因工程技術在總狀毛霉細胞內表達目的蛋白質的方法,仍然需要借助DNA載體,且一般需要使用總狀毛霉成熟菌絲體的原生質體作為宿主細胞,或者借助農桿菌作為介質,用DNA載體運載基因信息進入宿主細胞。
此外,編碼蛋白質的外源RNA導入未萌發孢子內部后,還需要與孢子內部的蛋白翻譯因子等各種細胞因子、酶系相互作用,才能翻譯出蛋白質分子。但是,眾所周知,睡眠孢子的生命活動處于最不旺盛的狀態,細胞內各種蛋白翻譯因子和酶系的活力是很低的,各種因子和酶系分子的數量也是很少的,即便外源RNA進入了未萌發孢子內部,也需要比較長的相互作用時間,才能翻譯出目的蛋白。而時間長,就意味著外源RNA在細胞內,被細胞內源性RNA酶降解的概率加大,因此,就要求要有足夠多數量的外源RNA,可以進入未萌發孢子內部。
如何讓真菌的孢子萌發,本領域已經有現成的技術方案,但是無論哪種萌發方法,都需要消耗時間、精力、人工操作、以及化學試劑。業內已經有用萌發的真菌孢子,導入外源DNA的案例,因此,按照本領域的基礎理論,一般認為,用細胞通透性更強的萌發孢子導入外源分子(例如外源的RNA)的成功率更大,因為萌發的孢子已經蘇醒,細胞內各種因子和酶系已經充分調動和活躍起來了,細胞要吸收養分,細胞內外物質交流活躍。可惜的是,目前為止,尚未有用孢子(無論是萌發還是未萌發)導入外源編碼RNA的先例。但是,顯而易見,用未萌發的孢子來作為導入外源分子的起始材料,非常簡便,因為可以省去做孢子萌發這一復雜的步驟,更重要的是,萌發的孢子,可能已經產生了多倍體,而未萌發的孢子,可以保證是單倍體,很多基因工程的應用,要求宿主細胞必須是單倍體,才能達到預想的結果或者效率。因此本發明嘗試繞過真菌孢子萌發這個步驟,直接用休眠的孢子,來導入外源RNA。
另外,眾所周知真菌能產生大量的RNA酶,并且能夠大量外分泌這些RNA酶,真菌是自然界RNA酶的主要來源之一。真菌孢子采集自菌體,采集真菌孢子時,孢子會攜帶有菌體分泌的大量的外源性RNA酶,而且很難在不殺滅孢子的情況下,把RNA酶徹底清洗干凈(如果使用DEPC等化合物來做徹底清洗,勢必會殺滅真菌孢子)。而且孢子自身也會產生和外分泌一些RNA酶。因此,使用外源單鏈RNA來轉化真菌細胞,非常困難,無異于雞蛋碰石頭,單鏈RNA還未接觸到真菌細胞就會被RNA酶降解了。
綜上所述,將編碼蛋白質的RNA分子,直接導入真菌(無論是菌絲體還是孢子,無論是萌發的孢子還是未萌發的孢子)內部,是本行業內一直沒有攻克的技術難題。
電擊轉化(Electroporation)是一種用電脈沖短暫作用于接觸了外源核酸的細胞,使外源核酸進入細胞的方法。電擊轉化法又根據各自特點,有許多細分。已經有文獻報道了用電擊法,遞送DNA進入某些種類真菌的細胞。但是,DNA與編碼蛋白的單鏈RNA相比,在結構上、在生物化學和分子生物學屬性上,均完全不同,它們與細胞壁、細胞膜的相互作用方式也是不同的,因此讓它們通過真菌的細胞壁、細胞膜并進入細胞內部的機制,也是不同的。DNA分子與單鏈RNA相比,DNA半衰期長,不容易被降解,非常穩定。用于遞送DNA的方法,不能直接用于遞送單鏈RNA,目前也沒有相關報道。
2013年面世的HDEN電擊轉化技術,是一種專門針對哺乳動物細胞的特性,而開發的電轉化技術,其開發目的是為了提高向哺乳動物細胞內傳送外源DNA和藥物的效率。該技術包含3大部分內容,一是施加于細胞樣品的電波形式,二是細胞樣品在電擊時的溶液環境,三是細胞樣品的培養方法和電擊前預處理方法。由于該技術是專門針對哺乳動物細胞開發的,因此,上述3大部分內容,都是針對哺乳動物細胞的特性而設計。
現有文獻報道了HDEN技術在HEK-293A,Hela,Neuro-2A,MCF-7,C2C12,3T3-L1,CHO,MDCK,HL-60,HUVEC,A375,U251等哺乳動物細胞中,遞送DNA進入細胞的應用案例。目前尚未有該技術在哺乳動物細胞以外的物種中應用的案例報道。因此,HDEN電轉化技術能不能應用于哺乳動物細胞以外的物種,也是一個未知數。
技術實現要素:
本發明的目的在于針對背景技術現狀,提供一種在總狀毛霉休眠孢子中表達蛋白質的方法,該方法繞過真菌孢子萌發這個步驟,采用HDEN電轉化技術,將體外的單鏈編碼蛋白RNA遞送穿過細胞壁和細胞膜,在總狀毛霉休眠孢子內表達蛋白質。
為實現上述目的,本發明采用如下技術方案:
一種在總狀毛霉休眠孢子中表達蛋白質的方法,包括以下步驟:
1)總狀毛霉培養和孢子收集
在固體瓊脂培養基表面接種總狀毛霉,培養至培養基表面長滿總狀毛霉孢子,洗下培養基表面的總狀毛霉孢子,吸出孢子懸液并過濾去除菌絲,收集含有孢子的濾液,將濾液離心,收集沉淀的休眠孢子;
2)總狀毛霉孢子預處理
用電擊緩沖液重懸孢子,離心并收集孢子沉淀,重復上述重懸和離心步驟3-4次后,將最后收集的孢子沉淀重懸于電擊緩沖液中,得到孢子濃度為104—1011個/ml的總狀毛霉孢子懸液;
所述電擊緩沖液由終濃度為0.01-100mmol/L的4-羥乙基哌嗪乙磺酸(HEPES)和終濃度為0.5-5000mmol/L的甘露醇組成,電擊緩沖液的pH為3.0-9.5;
3)使用HDEN法電擊總狀毛霉孢子
在細胞培養板的孔中加入上述步驟得到的總狀毛霉孢子懸液以及待轉化RNA,混勻,得到孢子與RNA混合物,將細胞培養板置于冰上冰浴10-15min,然后采用Etta Biotech X-Porator H1電轉儀進行HDEN法電擊,將帶有矩陣式電極的電擊頭插入孢子與RNA混合物內部,通電,使得孢子與RNA混合物內部產生電場,電擊之后再將細胞培養板置于冰上冰浴10-15min,然后將孢子和RNA混合物吸出,得到導入外源RNA的休眠的總狀毛霉孢子;
所述總狀毛霉懸液與待轉化RNA的比例為:0.6-60000μl的總狀毛霉孢子懸液:0.1-10000μg的待轉化RNA;
所述電擊參數如下:電壓1—6000V,脈寬2-2000000ms,重復1-100次,每次間隔5-50000ms。
進一步,所述步驟1)的培養基為PDA培養基、YPD培養基或察氏培養基,優選為PDA培養基的效果最好,產孢子最快最多。
所述步驟1,)總狀毛霉的培養條件為:溫度16-40℃,濕度15-85%,培養3-15日。
優選的,所述步驟1),總狀毛霉的培養條件為:溫度30℃,濕度50-60%,培養5日。
進一步,所述步驟2),電擊緩沖液由終濃度為1-10mmol/L的HEPES和終濃度為50-100mmol/L的甘露醇組成,電擊緩沖液的pH為5.0-7.0;
優選的,所述步驟2),電擊緩沖液由終濃度為1mmol/L的HEPES和終濃度為50mmol/L的甘露醇組成,電擊緩沖液的pH為7.0。
所述步驟2)的總狀毛霉孢子懸液在電擊前,于顯微鏡下觀察,確認孢子懸液中無菌絲體混雜污染并且孢子均未萌發,然后再進行電擊。
進一步,所述步驟3),待轉化RNA為外源單鏈編碼蛋白質的RNA,可以是綠色熒光蛋白的編碼RNA、紅色熒光蛋白的編碼RNA或黃色熒光蛋白的編碼RNA等等。
本發明擴增綠色熒光蛋白基因或黃色熒光蛋白基因的PCR引物如下:
F:5'AGATGACGTCGCTAGCATGGTGAGCAAGGGC 3';(SEQ ID NO.1)
R:5'ACGCGTCGACTTACTTGTACAGCTCGT3';(SEQ ID NO.2)
其中,引物F中靠近5’端的GCTAGC序列,用于促進該DNA轉錄成RNA后,翻譯起始因子與RNA結合,該序列與所表達的目的基因的起始密碼子ATG緊鄰。
本發明擴增紅色熒光蛋白基因用的PCR引物如下:
上游引物:RFP-F:5'CGGAATTCGCCACCATGGCCTCCTCCGAGGACGT 3';(SEQ ID NO.3)
下游引物:RFP-R:5'TCGAGCTCGTTAGGCGCCGGTGGAGTGG 3';(SEQ ID NO.4)
其中,引物RFP-F中靠近5’端的GCCACC序列,用于促進該DNA轉錄成RNA后,翻譯起始因子與RNA結合,該序列與所表達的目的基因的起始密碼子ATG緊鄰。
優選的,所述步驟3),總狀毛霉懸液與待轉化RNA的比例為:60μl的總狀毛霉孢子懸液:10μg的待轉化RNA。
進一步,所述步驟3),電壓300-1000V,脈寬1500-20000ms,重復1-50次,每次間隔500-5000ms;優選為:電壓600V,脈寬2500ms,重復3次,每次間隔500ms。
本發明所述電擊采用Etta Biotech X-Porator H1電轉儀,購買自蘇州壹達生物公司。
本發明收集未萌發的孢子,以及后續的電轉實驗過程,若無特殊說明,實驗操作均在不高于23攝氏度的恒溫實驗室中操作,離心步驟均為4℃冷凍離心,接觸未萌發孢子的各種液體均事先在冰上預冷。未萌發孢子嚴禁接觸到任何含有可促使其萌發的因素和物質(比如以YEPD等為代表的萌發培養基),以保證孢子的休眠狀態。
本發明總狀毛霉培養過程,當總狀毛霉表面長滿孢子后,向培養基表面倒入無菌水,洗下培養基表面的總狀毛霉孢子,用移液器吸出孢子懸液,使用滅菌過的擦鏡紙(或者砂芯漏斗、濾紙等)過濾,去除菌絲,保留孢子,如果沒有過濾步驟,那么孢子懸液中會混雜有菌絲,后續步驟得到的轉化子,無法判斷究竟是由孢子轉化DNA后形成的陽性克隆,還是菌絲轉化DNA后形成的假陽性。得到的孢子還要用染色體染色法對其染色體染色,并觀察和確認孢子內的染色體是單倍體狀態。
本發明制備固體瓊脂培養基的水應該是MillQ級別的分子生物學用高純水或雙蒸水,水的電阻率不低于18.2MΩ-cm。
使用HDEN法電擊總狀毛霉孢子時,在細胞培養板的孔中加入適當比例的總狀毛霉孢子懸液以及待轉化RNA,混合勻勻,將容器置于冰上進行冰浴。例如在96孔細胞培養板的孔中(NUNC公司的Nunclon Surface 96孔細胞培養板,貨號Cat.No.167008),加入60μl的總狀毛霉孢子懸液,以及不少于10μg的RNA。所述細胞培養板也可以是384孔板、24孔板、6孔板、或者其它更大、更小的容器,然后按照容器體積大小比例,放大或縮小孢子和RNA混合物體系。
本發明采用以上技術方案,用未萌發的孢子來作為導入外源分子的起始材料,非常簡便,因為可以省去做孢子萌發這一復雜的步驟,更重要的是,萌發的孢子,可能已經產生了多倍體,而未萌發的孢子,可以保證是單倍體,很多基因工程的應用,要求宿主細胞必須是單倍體,才能達到預想的結果或者效率。同時,本發明應用HDEN電轉化技術將外源RNA導入總狀毛霉休眠孢子。HDEN電轉化技術采用了高密度矩陣式電極,它能產生高度均勻且強度足夠的電場,電場內每個細胞接受電擊的條件幾乎完全一致,操作時,只要把細胞放在通用容器中,比如細胞培養板,再把帶有矩陣式電極的電擊頭插入容器中,通電即可。而傳統的電轉技術一般是使用專用電擊杯,在兩塊平行放置的金屬板之間放置細胞,并對金屬板加電,形成電場電擊。因此,二者的放電方式完全不同,前者的電極插入細胞懸液內部,在內部產生電場,而后者是在細胞懸液整體的外部產生電場;前者的電極頭由許多金屬針組成,在針與針之間產生電壓,而后者只有兩塊金屬板,一塊正極,一塊負極,在二者之間產生電壓。HDEN技術還基本消除了傳統電擊技術的陰極效應,避免產生大量氫氧根離子,避免殺傷細胞,提高電擊后的細胞存活率。而傳統電擊技術很難消除陰極效應。本發明的HDEN法,電擊多次,可以疊加多次電擊的效果,并且不會大幅增加細胞死亡率,而傳統的電擊方法一般只電擊1次,因為傳統電擊方法如果電擊多次,會導致細胞死亡率大幅度增加。
HDEN電轉化技術是一種專門針對哺乳動物細胞的特性而開發的電轉化技術,其開發目的是為了提高向哺乳動物細胞內傳送外源DNA和藥物的效率。而在本領域內眾所周知,微生物細胞的特性和培養方法,與哺乳動物細胞相比,千差萬別。哺乳動物細胞和微生物細胞的結構不同,哺乳動物細胞沒有細胞壁,微生物細胞(比如絕大部分真菌、包括總狀毛霉)有細胞壁。哺乳動物細胞的細胞膜和微生物的細胞膜也不一樣。即便是同一種生物,它的不同組織、不同器官或不同的細胞類型,各自的細胞膜、細胞壁的結構、狀態、化學成分,也都不一樣。哪怕是同一種生物的同樣的組織、同樣的器官、同樣的細胞類型,在不同的生長發育階段,或者在不同的外界環境中,各自的細胞膜、細胞壁的結構、狀態、化學成分,也都不一樣。而細胞壁和細胞膜,是阻擋外源分子進入細胞的屏障,屏障不同(結構不同,化學成分不同),決定了突破屏障的方法也是不同的。此外,不同的外源分子,因為具有不同的結構、分子量、體積和化學成分,面對相同的屏障時,不同外源分子突破屏障的方法也是不一樣的。如果面對不同的屏障,那么不同外源分子突破不同屏障的方法和機制,更是千差萬別了。
因此,任何應用于哺乳動物細胞的技術,都很難直接應用于微生物細胞。目前尚未有HDEN技術在哺乳動物細胞以外的物種中應用的案例報道。本發明針對總狀毛霉細胞的特性,采用HDEN技術將體外的單鏈編碼蛋白RNA導入總狀毛霉休眠孢子,該方法確定了細胞樣品的培養方法和電擊前預處理方法,細胞樣品在電擊時的溶液環境,以及施加于細胞樣品的電波形式。
此外,RNA翻譯成蛋白質,需要有調控序列與與翻譯起始因子結合并介導蛋白翻譯的起始。什么樣的調控序列可以在真菌(例如本發明的總狀毛霉)細胞內使用,是一個未知數,沒有任何文獻報道過。本發明還披露了可以在總狀毛霉細胞內使用的2種調控序列,見本發明實施例部分。
采用本發明方法,實現了歷史上首次將編碼蛋白質的RNA直接導入總狀毛霉休眠孢子并表達蛋白質,且步驟非常簡便快速,另外,效果極佳,可以得到至少90%的轉化率。
附圖說明
以下結合附圖和具體實施方式對本發明作進一步詳細的說明:
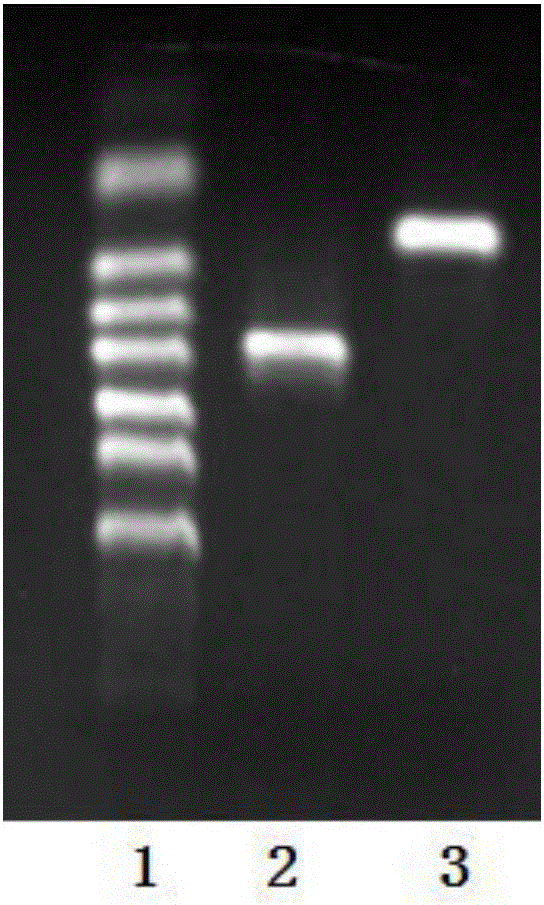
圖1為實施例1轉錄出的RNA產物的電泳檢測結果。
具體實施方式
以下的實施例便于更好的理解本發明,但并不限定本發明。
實驗過程的所有操作均要遵循微生物實驗的無菌操作原則,器皿、耗材、試劑均要滅菌處理。
本發明所用的所有內切酶均為Fermentas公司fastdigest系列限制性內切酶產品,以下實施例用到的T4DNA ligase為Fermentas公司產品,酶切和連接及膠回收和DNA純化操作,均按照產品說明書操作。
實施例1
在總狀毛霉細胞中表達綠色熒光蛋白(GFP):
一、質粒構建
使用GFP基因編碼序列,GFP基因如SEQ ID NO.5所示,
上述GFP基因的蛋白序列如SEQ ID NO.6所示,
擴增GFP基因用的PCR引物:
F:波浪線是Aat II酶切位點
R:波浪線是Sal I酶切位點
引物F中帶下劃線的GCTAGC序列,用于促進該DNA轉錄成RNA后,翻譯起始因子與RNA結合,該序列與所表達的目的基因的起始密碼子ATG緊鄰。用上述這對引物做PCR后,可以讓GFP基因上游帶上GCTAGC,上下游均帶上酶切位點。
瓊脂糖凝膠電泳檢測PCR產物無誤后,使用Thermo GeneJET Gel Extraction and DNA Cleanup Micro Kit回收純化PCR產物。
將上述PCR產物用Aat II和Sal I雙酶切,用T4DNA ligase將它連接在也經過Aat II和Sal I雙酶切的Promega公司pGEM-T easy質粒上,轉化大腸桿菌,挑選陽性克隆,用質粒上的通用測序引物測序驗證插入序列無誤后,大量提取重組質粒。上述步驟正好可以讓該GFP基因位于該質粒載體T7promoter的下游。
體外轉錄
用Fermentas公司的Fastdigest Nde1內切酶對上述提取的重組質粒做單酶切線性化。使用Thermo Scientific TranscriptAid T7High Yield Transcription Kit,對上述重組質粒做體外轉錄,均按照說明書操作。
轉錄出的RNA產物純化
1)在20μl轉錄反應體系中加入115μl的DEPC-H2O和15μl的3M NaAc溶液(pH5.2),混勻;
2)加入等體積的酚氯仿異戊醇混合物(酚:氯仿:異戊醇=25:24:1),混勻;
3)高速離心,使液體上下分層,取上層水相至另一個1.5ml離心管。
4)在水相中加入2倍體積無水乙醇,置于-20℃至少30min,高速離心,沉淀RNA,去上清。
5)加入1ml預冷的70%乙醇,輕輕顛倒后高速離心1min;
6)將沉淀重懸于合適體積的DEPC-H2O中,使RNA濃度不低于10μg/μL;
電泳檢測結果如圖1所示:
泳道1:RiboRuler RNA Ladder,high range,ready-to-use;
泳道2:positive control,2222nt;
泳道3:體外轉錄出的GFP的RNA,~750nt
二、使用上述綠色熒光蛋白的編碼RNA在總狀毛霉休眠孢子中表達綠色熒光蛋白,步驟如下:
1)總狀毛霉培養和孢子收集
在15cm培養皿中,制備固體瓊脂培養基(PDA培養基),在固體瓊脂培養基表面接種總狀毛霉CICC 40714,溫度30℃,濕度50-60%,培養5日,讓培養基表面長滿總狀毛霉孢子。
向培養基表面倒入無菌水,洗下(震蕩,或者用光滑的無菌玻璃涂布棒輕輕刮蹭)培養基表面的總狀毛霉孢子,用移液器吸出孢子懸液,使用滅菌過的擦鏡紙(或者砂芯漏斗、濾紙等)過濾,去除菌絲,保留孢子,濾過的液體裝入離心管,離心后,收集沉淀的休眠孢子,去除上清液體。得到的孢子還要用染色體染色法對其染色體染色,并觀察和確認孢子內的染色體是單倍體狀態。
2)總狀毛霉孢子預處理
用電擊緩沖液重懸孢子沉淀(加入電擊緩沖液的體積應能填充滿離心管),再離心,收集孢子沉淀,去除上清液體。重復上述步驟兩次后,再次將孢子重懸于電擊緩沖液中,顯微鏡觀察,確認孢子懸液中無菌絲體混雜污染,以及確認孢子均未萌發。最后一次將孢子重懸于電擊緩沖液時,應控制好電擊緩沖液的體積,保持總狀毛霉孢子懸液中孢子濃度為108個/ml。
所述電擊緩沖液由終濃度為1mmol/L的HEPES和終濃度為50mmol/L的甘露醇組成,電擊緩沖液的pH為7.0。
3)使用HDEN法電擊總狀毛霉孢子
在96孔細胞培養板的孔中加入60μl的總狀毛霉孢子懸液,及10μg綠色熒光蛋白的編碼RNA,混勻,得到孢子與RNA混合物,將容器置于冰上冰浴10min,然后采用Etta Biotech X-Porator H1電轉儀進行HDEN法電擊,將帶有矩陣式電極的電擊頭插入孢子與RNA混合物內部,通電,使得孢子與RNA混合物內部產生電場,電擊之后再將細胞培養板置于冰上冰浴10min,然后將孢子和RNA混合物吸出,得到導入外源RNA的休眠的總狀毛霉孢子;
本實施例電擊參數如下:電壓600V,脈寬2500ms,重復3次,每次間隔500ms。
4)驗證實驗
將上述吸出的孢子和RNA混合物,加入到100倍體積的YPD培養基中,30℃培養20小時后,用激光共聚焦顯微鏡觀察。
在進行上述實驗步驟的同時,需要制備對照組:將未電擊的、相同的“孢子和RNA混合物”,加入到另一YPD培養基中,相同條件下培養,然后用激光共聚焦顯微鏡進行觀察。
結果顯示:對照組的細胞無熒光,樣品組至少95%的細胞有綠色熒光,表明有成功的RNA轉化。
實施例2
在總狀毛霉細胞中表達紅色熒光蛋白(RFP):
一、質粒構建
RFP的核酸序列如SEQ ID NO.7所示,
RFP的蛋白序列如SEQ ID NO.8所示,
擴增RFP基因的引物如下:
上游引物:RFP-F:5'CGGAATTCGCCACCATGGCCTCCTCCGAGGACGT 3'
下游引物:RFP-R:5'TCGAGCTCGTTAGGCGCCGGTGGAGTGG 3'
上游引物5端帶有EcoR I酶切位點,帶下劃線的GCCACC序列,用于促進該DNA轉錄成RNA后,翻譯起始因子與RNA結合,該序列與所表達的目的基因的起始密碼子ATG緊鄰。
下游引物5端帶有Sal I酶切位點。
按照實施例1的做法,把RFP基因通過PCR擴增,做分子克隆,連接到pGEM-T easy載體上,再做體外轉錄,并將所得RNA轉化宿主孢子。
二、使用紅色熒光蛋白的編碼RNA在總狀毛霉休眠孢子中表達紅色熒光蛋白,步驟如下:
1)總狀毛霉培養和孢子收集
在15cm培養皿中,制備固體瓊脂培養基(YPD培養基),在固體瓊脂培養基表面接種總狀毛霉CICC 40714,在溫度16℃,濕度15-50%下,培養15日,讓培養基表面長滿總狀毛霉孢子。
向培養基表面倒入無菌水,洗下(震蕩,或者用光滑的無菌玻璃涂布棒輕輕刮蹭)培養基表面的總狀毛霉孢子,用移液器吸出孢子懸液,使用滅菌過的擦鏡紙(或者砂芯漏斗、濾紙等)過濾,去除菌絲,保留孢子,濾過的液體裝入離心管,離心后,收集沉淀的休眠孢子,去除上清液體。得到的孢子還要用染色體染色法對其染色體染色,并觀察和確認孢子內的染色體是單倍體狀態。
2)總狀毛霉孢子預處理
用電擊緩沖液重懸孢子沉淀(加入電擊緩沖液的體積應能填充滿離心管),再離心,收集孢子沉淀,去除上清液體。重復上述步驟兩次后,再次將孢子重懸于電擊緩沖液中,顯微鏡觀察,確認孢子懸液中無菌絲體混雜污染,以及確認孢子均未萌發。最后一次將孢子重懸于電擊緩沖液時,應控制好電擊緩沖液的體積,保持總狀毛霉孢子懸液中孢子濃度為1011個/ml。
所述電擊緩沖液由終濃度為0.01mmol/L的HEPES和終濃度為0.5mmol/L的甘露醇組成,電擊緩沖液的pH為3.0。
3)使用HDEN法電擊總狀毛霉孢子
在96孔細胞培養板的孔中加入0.6μl的總狀毛霉孢子懸液,及0.1μg紅色熒光蛋白的編碼RNA,混勻,得到孢子與RNA混合物,將容器置于冰上冰浴15min,然后采用Etta Biotech X-Porator H1電轉儀進行HDEN法電擊,將帶有矩陣式電極的電擊頭插入孢子與RNA混合物內部,通電,使得孢子與RNA混合物內部產生電場,電擊之后再將細胞培養板置于冰上冰浴15min,然后將孢子和RNA混合物吸出,得到導入外源RNA的休眠的總狀毛霉孢子;
本實施例電擊參數如下:電壓1V,脈寬2000000ms,重復100次,每次間隔5ms。
4)驗證實驗
將上述吸出的孢子和RNA混合物,加入到10倍體積的YPD培養基中,10℃培養25小時后,用激光共聚焦顯微鏡觀察。
在進行上述實驗步驟的同時,需要制備對照組:將未電擊的、相同的“孢子和RNA混合物”,加入到另一YPD培養基中,相同條件下培養,然后用激光共聚焦顯微鏡進行觀察。
結果顯示:對照組的細胞無熒光,樣品組至少90%以上的細胞有紅色熒光,表明有成功的RNA轉化。
實施例3
總狀毛霉細胞中表達黃色熒光蛋白(YFP):
一、質粒構建
YFP的核酸序列如SEQ ID NO.9所示,
YFP的蛋白序列如SEQ ID NO.10所示,
采用與實施例1中GFP相同的引物,并按照同樣的實驗步驟和方法把GFP基因通過PCR擴增,做分子克隆,連接到pGEM-T easy載體上,再做體外轉錄,并將所得RNA轉化宿主孢子。
二、使用黃色熒光蛋白的編碼RNA在總狀毛霉休眠孢子中表達黃色熒光蛋白,步驟如下:
1)總狀毛霉培養和孢子收集
在15cm培養皿中,制備固體瓊脂培養基(PDA培養基),在固體瓊脂培養基表面接種總狀毛霉CICC 40714,在溫度40℃,濕度60-85%下,培養3日,讓培養基表面長滿總狀毛霉孢子。
向培養基表面倒入無菌水,洗下(震蕩,或者用光滑的無菌玻璃涂布棒輕輕刮蹭)培養基表面的總狀毛霉孢子,用移液器吸出孢子懸液,使用滅菌過的擦鏡紙(或者砂芯漏斗、濾紙等)過濾,去除菌絲,保留孢子,濾過的液體裝入離心管,離心后,收集沉淀的休眠孢子,去除上清液體。得到的孢子還要用染色體染色法對其染色體染色,并觀察和確認孢子內的染色體是單倍體狀態。
2)總狀毛霉孢子預處理
用電擊緩沖液重懸孢子沉淀(加入電擊緩沖液的體積應能填充滿離心管),再離心,收集孢子沉淀,去除上清液體。重復上述步驟兩次后,再次將孢子重懸于電擊緩沖液中,顯微鏡觀察,確認孢子懸液中無菌絲體混雜污染,以及確認孢子均未萌發。最后一次將孢子重懸于電擊緩沖液時,應控制好電擊緩沖液的體積,保持總狀毛霉孢子懸液中孢子濃度為104個/ml。
所述電擊緩沖液由終濃度為100mmol/L的HEPES和終濃度為5000mmol/L的甘露醇組成,電擊緩沖液的pH為7.0。
3)使用HDEN法電擊總狀毛霉孢子
在96孔細胞培養板的孔中加入60000μl的總狀毛霉孢子懸液,及10000μg紅色熒光蛋白的編碼RNA,混勻,得到孢子與RNA混合物,將容器置于冰上冰浴10min,然后采用Etta Biotech X-Porator H1電轉儀進行HDEN法電擊,將帶有矩陣式電極的電擊頭插入孢子與RNA混合物內部,通電,使得孢子與RNA混合物內部產生電場,電擊之后再將細胞培養板置于冰上冰浴10min,然后將孢子和質粒混合物吸出,得到導入外源RNA的休眠的總狀毛霉孢子;
本實施例電擊參數如下:電壓6000V,脈寬2ms,重復1次,間隔50000ms。
4)驗證實驗
將上述吸出的孢子和RNA混合物,加入到10倍體積的YPD培養基中,40℃培養24小時后,用激光共聚焦顯微鏡觀察。
在進行上述實驗步驟的同時,需要制備對照組:將未電擊的、相同的“孢子和RNA混合物”,加入到另一YPD培養基中,相同條件下培養,然后用激光共聚焦顯微鏡進行觀察。
結果顯示:對照組的細胞無熒光,樣品組至少90%以上的細胞有黃色熒光,表明有成功的RNA轉化。
實施例4
本實施例電擊參數為:電壓30V,脈寬1000000ms,電擊50次,間隔5000ms,其它同實施例1。
實施例5
本實施例電擊參數為:電壓3000V,脈寬100ms,電擊5次,間隔25000ms,其它同實施例1。
本發明所述的將編碼蛋白質的線性RNA導入細胞、在細胞內表達的蛋白質,可以是該細胞、該物種自身就能編碼能表達的蛋白質,也可以是其它生物物種來源的蛋白質,或者是人工設計的蛋白質。
所有的外源單鏈編碼蛋白質的RNA,都可以用本發明的方法做轉化,不僅限于實施例所述綠色、紅色或黃色熒光蛋白的編碼RNA。
SEQUENCE LISTING
<110> 福州大學
<120> 一種在總狀毛霉休眠孢子中表達蛋白質的方法
<130> 1
<160> 10
<170> PatentIn version 3.3
<210> 1
<211> 31
<212> DNA
<213> 人工序列
<400> 1
agatgacgtc gctagcatgg tgagcaaggg c 31
<210> 2
<211> 27
<212> DNA
<213> 人工序列
<400> 2
acgcgtcgac ttacttgtac agctcgt 27
<210> 3
<211> 34
<212> DNA
<213> 人工序列
<400> 3
cggaattcgc caccatggcc tcctccgagg acgt 34
<210> 4
<211> 28
<212> DNA
<213> 人工序列
<400> 4
tcgagctcgt taggcgccgg tggagtgg 28
<210> 5
<211> 720
<212> DNA
<213> 人工序列
<400> 5
atggtgagca agggcgagga gctgttcacc ggggtggtgc ccatcctggt cgagctggac 60
ggcgacgtaa acggccacaa gttcagcgtg tccggcgagg gcgagggcga tgccacctac 120
ggcaagctga ccctgaagtt catctgcacc accggcaagc tgcccgtgcc ctggcccacc 180
ctcgtgacca ccctgaccta cggcgtgcag tgcttcagcc gctaccccga ccacatgaag 240
cagcacgact tcttcaagtc cgccatgccc gaaggctacg tccaggagcg caccatcttc 300
ttcaaggacg acggcaacta caagacccgc gccgaggtga agttcgaggg cgacaccctg 360
gtgaaccgca tcgagctgaa gggcatcgac ttcaaggagg acggcaacat cctggggcac 420
aagctggagt acaactacaa cagccacaac gtctatatca tggccgacaa gcagaagaac 480
ggcatcaagg tgaacttcaa gatccgccac aacatcgagg acggcagcgt gcagctcgcc 540
gaccactacc agcagaacac ccccatcggc gacggccccg tgctgctgcc cgacaaccac 600
tacctgagca cccagtccgc cctgagcaaa gaccccaacg agaagcgcga tcacatggtc 660
ctgctggagt tcgtgaccgc cgccgggatc actctcggca tggacgagct gtacaagtaa 720
<210> 6
<211> 239
<212> PRT
<213> 人工序列
<400> 6
Met Val Ser Lys Gly Glu Glu Leu Phe Thr Gly Val Val Pro Ile Leu
1 5 10 15
Val Glu Leu Asp Gly Asp Val Asn Gly His Lys Phe Ser Val Ser Gly
20 25 30
Glu Gly Glu Gly Asp Ala Thr Tyr Gly Lys Leu Thr Leu Lys Phe Ile
35 40 45
Cys Thr Thr Gly Lys Leu Pro Val Pro Trp Pro Thr Leu Val Thr Thr
50 55 60
Leu Thr Tyr Gly Val Gln Cys Phe Ser Arg Tyr Pro Asp His Met Lys
65 70 75 80
Gln His Asp Phe Phe Lys Ser Ala Met Pro Glu Gly Tyr Val Gln Glu
85 90 95
Arg Thr Ile Phe Phe Lys Asp Asp Gly Asn Tyr Lys Thr Arg Ala Glu
100 105 110
Val Lys Phe Glu Gly Asp Thr Leu Val Asn Arg Ile Glu Leu Lys Gly
115 120 125
Ile Asp Phe Lys Glu Asp Gly Asn Ile Leu Gly His Lys Leu Glu Tyr
130 135 140
Asn Tyr Asn Ser His Asn Val Tyr Ile Met Ala Asp Lys Gln Lys Asn
145 150 155 160
Gly Ile Lys Val Asn Phe Lys Ile Arg His Asn Ile Glu Asp Gly Ser
165 170 175
Val Gln Leu Ala Asp His Tyr Gln Gln Asn Thr Pro Ile Gly Asp Gly
180 185 190
Pro Val Leu Leu Pro Asp Asn His Tyr Leu Ser Thr Gln Ser Ala Leu
195 200 205
Ser Lys Asp Pro Asn Glu Lys Arg Asp His Met Val Leu Leu Glu Phe
210 215 220
Val Thr Ala Ala Gly Ile Thr Leu Gly Met Asp Glu Leu Tyr Lys
225 230 235
<210> 7
<211> 678
<212> DNA
<213> 人工序列
<400> 7
atggcctcct ccgaggacgt catcaaggag ttcatgcgct tcaaggtgcg catggagggc 60
tccgtgaacg gccacgagtt cgagatcgag ggcgagggcg agggccgccc ctacgagggc 120
acccagaccg ccaagctgaa ggtgaccaag ggcggccccc tgcccttcgc ctgggacatc 180
ctgtcccctc agttccagta cggctccaag gcctacgtga agcaccccgc cgacatcccc 240
gactacttga agctgtcctt ccccgagggc ttcaagtggg agcgcgtgat gaacttcgag 300
gacggcggcg tggtgaccgt gacccaggac tcctccctgc aggacggcga gttcatctac 360
aaggtgaagc tgcgcggcac caacttcccc tccgacggcc ccgtaatgca gaagaagacc 420
atgggctggg aggcctccac cgagcggatg taccccgagg acggcgccct gaagggcgag 480
atcaagatga ggctgaagct gaaggacggc ggccactacg acgccgaggt caagaccacc 540
tacatggcca agaagcccgt gcagctgccc ggcgcctaca agaccgacat caagctggac 600
atcacctccc acaacgagga ctacaccatc gtggaacagt acgagcgcgc cgagggccgc 660
cactccaccg gcgcctaa 678
<210> 8
<211> 225
<212> PRT
<213> 人工序列
<400> 8
Met Ala Ser Ser Glu Asp Val Ile Lys Glu Phe Met Arg Phe Lys Val
1 5 10 15
Arg Met Glu Gly Ser Val Asn Gly His Glu Phe Glu Ile Glu Gly Glu
20 25 30
Gly Glu Gly Arg Pro Tyr Glu Gly Thr Gln Thr Ala Lys Leu Lys Val
35 40 45
Thr Lys Gly Gly Pro Leu Pro Phe Ala Trp Asp Ile Leu Ser Pro Gln
50 55 60
Phe Gln Tyr Gly Ser Lys Ala Tyr Val Lys His Pro Ala Asp Ile Pro
65 70 75 80
Asp Tyr Leu Lys Leu Ser Phe Pro Glu Gly Phe Lys Trp Glu Arg Val
85 90 95
Met Asn Phe Glu Asp Gly Gly Val Val Thr Val Thr Gln Asp Ser Ser
100 105 110
Leu Gln Asp Gly Glu Phe Ile Tyr Lys Val Lys Leu Arg Gly Thr Asn
115 120 125
Phe Pro Ser Asp Gly Pro Val Met Gln Lys Lys Thr Met Gly Trp Glu
130 135 140
Ala Ser Thr Glu Arg Met Tyr Pro Glu Asp Gly Ala Leu Lys Gly Glu
145 150 155 160
Ile Lys Met Arg Leu Lys Leu Lys Asp Gly Gly His Tyr Asp Ala Glu
165 170 175
Val Lys Thr Thr Tyr Met Ala Lys Lys Pro Val Gln Leu Pro Gly Ala
180 185 190
Tyr Lys Thr Asp Ile Lys Leu Asp Ile Thr Ser His Asn Glu Asp Tyr
195 200 205
Thr Ile Val Glu Gln Tyr Glu Arg Ala Glu Gly Arg His Ser Thr Gly
210 215 220
Ala
225
<210> 9
<211> 720
<212> DNA
<213> 人工序列
<400> 9
atggtgagca agggcgagga gctgttcacc ggggtggtgc ccatcctggt cgagctggac 60
ggcgacgtaa acggccacaa gttcagcgtg tccggcgagg gcgagggcga tgccacctac 120
ggcaagctga ccctgaagtt catctgcacc accggcaagc tgcccgtgcc ctggcccacc 180
ctcgtgacca ccttcggcta cggcctgcag tgcttcgccc gctaccccga ccacatgaag 240
cagcacgact tcttcaagtc cgccatgccc gaaggctacg tccaggagcg caccatcttc 300
ttcaaggacg acggcaacta caagacccgc gccgaggtga agttcgaggg cgacaccctg 360
gtgaaccgca tcgagctgaa gggcatcgac ttcaaggagg acggcaacat cctggggcac 420
aagctggagt acaactacaa cagccacaac gtctatatca tggccgacaa gcagaagaac 480
ggcatcaagg tgaacttcaa gatccgccac aacatcgagg acggcagcgt gcagctcgcc 540
gaccactacc agcagaacac ccccatcggc gacggccccg tgctgctgcc cgacaaccac 600
tacctgagct accagtccgc cctgagcaaa gaccccaacg agaagcgcga tcacatggtc 660
ctgctggagt tcgtgaccgc cgccgggatc actctcggca tggacgagct gtacaagtga 720
<210> 10
<211> 239
<212> PRT
<213> 人工序列
<400> 10
Met Val Ser Lys Gly Glu Glu Leu Phe Thr Gly Val Val Pro Ile Leu
1 5 10 15
Val Glu Leu Asp Gly Asp Val Asn Gly His Lys Phe Ser Val Ser Gly
20 25 30
Glu Gly Glu Gly Asp Ala Thr Tyr Gly Lys Leu Thr Leu Lys Phe Ile
35 40 45
Cys Thr Thr Gly Lys Leu Pro Val Pro Trp Pro Thr Leu Val Thr Thr
50 55 60
Phe Gly Tyr Gly Leu Gln Cys Phe Ala Arg Tyr Pro Asp His Met Lys
65 70 75 80
Gln His Asp Phe Phe Lys Ser Ala Met Pro Glu Gly Tyr Val Gln Glu
85 90 95
Arg Thr Ile Phe Phe Lys Asp Asp Gly Asn Tyr Lys Thr Arg Ala Glu
100 105 110
Val Lys Phe Glu Gly Asp Thr Leu Val Asn Arg Ile Glu Leu Lys Gly
115 120 125
Ile Asp Phe Lys Glu Asp Gly Asn Ile Leu Gly His Lys Leu Glu Tyr
130 135 140
Asn Tyr Asn Ser His Asn Val Tyr Ile Met Ala Asp Lys Gln Lys Asn
145 150 155 160
Gly Ile Lys Val Asn Phe Lys Ile Arg His Asn Ile Glu Asp Gly Ser
165 170 175
Val Gln Leu Ala Asp His Tyr Gln Gln Asn Thr Pro Ile Gly Asp Gly
180 185 190
Pro Val Leu Leu Pro Asp Asn His Tyr Leu Ser Tyr Gln Ser Ala Leu
195 200 205
Ser Lys Asp Pro Asn Glu Lys Arg Asp His Met Val Leu Leu Glu Phe
210 215 220
Val Thr Ala Ala Gly Ile Thr Leu Gly Met Asp Glu Leu Tyr Lys
225 230 235
- 還沒有人留言評論。精彩留言會獲得點贊!