順式?5?羥基?L?哌可酸的制造方法與流程
本發明涉及利用了具有生產順式-5-羥基-l-哌可酸的能力的酶的順式-5-羥基-l-哌可酸的制造方法。
背景技術:
順式-5-羥基-l-哌可酸(以下有時稱為“5oh-pa”)是作為藥品的中間體等而有用的化合物。已知順式-5-羥基-l-哌可酸可以利用生物學方法由l-哌可酸進行制造。
已經報道了百脈根中慢生根瘤菌(mesorhizobiumloti)maff303099來源的bab52605蛋白質、苜蓿中華根瘤菌(sinorhizobiummeliloti)1021來源的cac47686蛋白質(以下有時稱為“smph”)具有將l-脯氨酸轉變為順式-4-羥基脯氨酸的能力(專利文獻1)。
bab52605蛋白質雖然具有將l-哌可酸轉變為順式-5-羥基-l-哌可酸的能力,但據報道,其生產率比較低(專利文獻2)。
雖然報道了cac47686蛋白質也具有將l-哌可酸轉變為順式-5-羥基-l-哌可酸的能力,但其在由l-哌可酸生產順式-5-羥基-l-哌可酸的同時,還生產出幾乎等量的順式-3-羥基哌可酸(以下有時稱為“3oh-pa”)(專利文獻2)。
另外,專利文獻2中報道了表達由segniliparusrugosusatccbaa-974來源的efv12517蛋白質的注釋的更上游的48個堿基(相當于16個氨基酸)所表達的多核苷酸(cis基因)編碼的蛋白質(以下有時稱為“sruph”)的大腸桿菌具有l-哌可酸的順式-5位羥化酶活性,能夠將l-哌可酸轉變為順式-5-羥基-l-哌可酸。但是,如本說明書的實施例中所示,該蛋白質在由l-哌可酸生產順式-5-羥基-l-哌可酸的同時,生產出約2%的順式-3-羥基哌可酸。
專利文獻3中報道了由l-哌可酸生產順式-5-羥基-l-哌可酸的方法。在專利文獻3中報道了,為了抑制不需要的順式-3-羥基哌可酸的生產而改變了smph的基因,但即使改變smph的基因,也會生產出約9%的順式-3-羥基哌可酸。
在非專利文獻1中也報道了smph由l-哌可酸生產順式-5-羥基-l-哌可酸和順式-3-羥基哌可酸。在非專利文獻1中報道了,為了提高順式-5-羥基-l-哌可酸的生產率而改變了smph基因,但即使改變smph基因,也會生產出約4%的順式-3-羥基哌可酸。
現有技術文獻
專利文獻
專利文獻1:wo2009/139365
專利文獻2:wo2013/187438
專利文獻3:wo2013/169725
非專利文獻
非專利文獻1:koketsu等、acssynth.biol,doi:10.1021/sb500247a
技術實現要素:
發明所要解決的問題
由此可見,雖然利用生物學方法由l-哌可酸制造順式-5-羥基-l-哌可酸的方法是已知的,但每種方法的順式-5-羥基-l-哌可酸的生產率均低,并且,會一定程度地生產出順式-3-羥基-l-哌可酸。因此,作為要求高純度的藥品的中間體等的制造方法,期望更高效地制造高純度的順式-5-羥基-l-哌可酸的方法。
本發明的課題在于提供順式-3-羥基-l-哌可酸的生成少、更高效地制造光學純度高的順式-5-羥基-l-哌可酸的新方法。
用于解決問題的手段
本發明人們為了解決上述問題進行了深入研究,結果發現,xenorhabdusdoucetiaefrm16株來源的蛋白質(以下有時稱為“xdph”)和xenorhabdusromaniistr.pr06-a株來源的蛋白質(以下有時稱為“xrph”)具有高的l-哌可酸的順式-5位羥化酶活性。而且發現,通過使用編碼這些蛋白質的dna制作轉化體并使該轉化體細胞、其制備物和/或培養液與l-哌可酸發生作用,能夠以高光學純度及高濃度制造順式-5-羥基-l-哌可酸。本發明是基于這些發現而完成的。
即,本發明的主旨如下所述。
[1]一種順式-5-羥基-l-哌可酸的制造方法,其特征在于,使α-酮戊二酸依賴型l-哌可酸羥化酶、具有生產該酶的能力的微生物或細胞、該微生物或細胞的處理物、和/或對該微生物或細胞進行培養而得到的含有該酶的培養液與l-哌可酸發生作用,生成順式-5-羥基-l-哌可酸,
上述α-酮戊二酸依賴型l-哌可酸羥化酶包含下述(a)、(b)或(c)所示的多肽:
(a)具有序列編號4或11所表示的氨基酸序列的多肽;
(b)具有在序列編號4或11所表示的氨基酸序列中缺失、置換和/或添加了1個或幾個氨基酸而得到的氨基酸序列、且具有α-酮戊二酸依賴型l-哌可酸羥化酶活性的多肽;或
(c)具有與序列編號4或11所表示的氨基酸序列具有60%以上的同一性的氨基酸序列、且具有α-酮戊二酸依賴型l-哌可酸羥化酶活性的多肽。
[2]如[1]所述的順式-5-羥基-l-哌可酸的制造方法,其中,編碼上述α-酮戊二酸依賴型l-哌可酸羥化酶的dna包含下述(d)、(e)或(f)所示的dna:
(d)具有序列編號1、9或10所表示的堿基序列的dna;
(e)包含在序列編號1、9或10所表示的堿基序列中置換、缺失和/或添加了1個或幾個堿基而得到的堿基序列、且編碼具有α-酮戊二酸依賴型l-哌可酸羥化酶活性的多肽的dna;或
(f)包含在嚴格條件下與序列編號1、9或10所表示的堿基序列的互補鏈雜交的堿基序列、且編碼具有α-酮戊二酸依賴型l-哌可酸羥化酶活性的多肽的dna。
[3]如[1]或[2]所述的順式-5-羥基-l-哌可酸的制造方法,其中,使α-酮戊二酸依賴型l-哌可酸羥化酶、具有生產該酶的能力的微生物或細胞、該微生物或細胞的處理物、和/或對該微生物或細胞進行培養而得到的含有該酶的培養液在α-酮戊二酸和二價鐵離子的存在下與上述l-哌可酸發生作用。
[4]一種α-酮戊二酸依賴型l-哌可酸羥化酶蛋白質,其具有與l-哌可酸發生作用而生成順式-5-羥基-l-哌可酸的活性,并且包含下述(a)、(b)或(c)所示的多肽:
(a)具有序列編號4或11所表示的氨基酸序列的多肽;
(b)具有在序列編號4或11所表示的氨基酸序列中缺失、置換和/或添加了1個或幾個氨基酸而得到的氨基酸序列、且具有α-酮戊二酸依賴型l-哌可酸羥化酶活性的多肽;或
(c)具有與序列編號4或11所表示的氨基酸序列具有60%以上的同一性的氨基酸序列、且具有α-酮戊二酸依賴型l-哌可酸羥化酶活性的多肽。
[5]一種多肽,其具有序列編號11所表示的氨基酸序列。
發明效果
根據本發明,能夠由l-哌可酸更高效地制造光學純度高的順式-5-羥基-l-哌可酸。此外,由于順式-3-羥基-l-哌可酸的生成少,因此在工業規模的制造中能夠以低成本制造高純度的順式-5-羥基-l-哌可酸。
附圖說明
圖1是示出導入有各種l-哌可酸羥化酶基因的大腸桿菌的sds-聚丙烯酰胺電泳法的結果的圖(電泳照片)。箭頭示出l-哌可酸羥化酶的條帶。
圖2是示出由導入有各種l-哌可酸羥化酶基因的大腸桿菌得到的反應產物的分析結果的圖。
圖3是示出各種l-哌可酸羥化酶的活性的溫度依賴性的圖。
具體實施方式
以下,對本發明進行詳細說明。
本說明書中,“將l-哌可酸轉變為順式-5-羥基-l-哌可酸的能力”是指以α-酮戊二酸依賴的方式在l-哌可酸的5位碳原子上添加羥基的能力。
是否具有“將l-哌可酸轉變為順式-5-羥基-l-哌可酸的能力”例如可以通過如下方法進行確認:在含有l-哌可酸作為底物、進而含有α-酮戊二酸作為輔酶的反應體系中,使作為測定對象的酶與l-哌可酸發生作用,直接測定由l-哌可酸轉變而來的順式-5-羥基-l-哌可酸的量。
另外,本說明書中的“酶”也包括純化酶(包括部分純化的酶)、使用公知的固定化技術固定化的酶、例如固定化于聚丙烯酰胺、卡拉膠等載體上的酶等。
本說明書中,“具有將l-哌可酸轉變為順式-5-羥基-l-哌可酸的能力的微生物或細胞”(以下有時稱為“本發明的微生物或細胞”)只要具有“能夠在l-哌可酸的5位碳原子上添加羥基的能力”,則沒有特別限制,可以是內源性地具有上述能力的微生物或細胞,也可以是通過育種而賦予了上述能力的微生物或細胞。作為通過育種而賦予了上述能力的手段,可以采用基因重組處理(轉化)、突變處理等公知的方法。作為轉化的方法,可以使用導入目的基因、通過在染色體上改變啟動子等表達調節序列等來強化目的基因的表達等方法。
需要說明的是,作為“微生物或細胞”的種類,可以列舉后述的宿主生物或宿主細胞中記載的微生物或細胞。另外,本說明書中,作為“具有能夠在l-哌可酸的5位碳原子上添加羥基的能力的微生物或細胞”,不限定于活的微生物或細胞,也包括作為生物體來說已死亡但仍具有酶能力的微生物或細胞。
本說明書中,作為“宿主生物”的生物的種類,沒有特別限定,可以列舉大腸桿菌、枯草桿菌、棒狀桿菌、假單胞菌屬細菌、芽孢桿菌屬細菌、根瘤菌屬細菌、乳桿菌屬細菌、琥珀酸桿菌屬細菌(サクシノバチルス屬細菌)、厭氧螺菌屬細菌、放線桿菌屬細菌等原核生物、酵母、絲狀真菌等菌類、植物、動物等真核生物。其中,優選為大腸桿菌、酵母、棒狀桿菌,特別優選為大腸桿菌。
本說明書中,作為“宿主細胞”的細胞的種類,沒有特別限定,可以使用動物細胞、植物細胞、昆蟲細胞等。
本說明書中,“表達載體”是指用于通過整合入編碼具有期望功能的蛋白質的多核苷酸并導入宿主生物而在上述宿主生物中復制和表達具有期望功能的蛋白質的遺傳因子。可以列舉例如質粒、病毒、噬菌體、粘粒等,但不限定于此。優選表達載體為質粒。
本說明書中,“轉化體”是指使用上述表達載體等導入目的基因、從而變得能夠表現出與具有期望功能的蛋白質相關的期望性狀的微生物或細胞。
本說明書中,“微生物或細胞的處理物”是指下述物質等:對微生物或細胞進行培養并將該微生物或細胞1)利用有機溶劑等進行處理而得到的含有具有期望功能的蛋白質的物質、2)進行冷凍干燥而得到的含有具有期望功能的蛋白質的物質、3)固定化于載體等中而得到的含有具有期望功能的蛋白質的物質、4)利用物理方法或利用酶破壞而得到的含有具有期望功能的蛋白質的物質。
本說明書中,“對微生物或細胞進行培養而得到的含有酶的培養液”是指,1)微生物或細胞的培養液、2)利用有機溶劑等對微生物或細胞的培養液進行處理而得到的培養液、3)利用物理方法或利用酶將微生物或細胞的細胞膜破壞而得到的培養液。
<使用α-酮戊二酸依賴型l-哌可酸羥化酶的順式-5-羥基-l-哌可酸的制造方法>
本發明的順式-5-羥基-l-哌可酸的制造方法的特征在于,使α-酮戊二酸依賴型l-哌可酸羥化酶、具有生產該酶的能力的微生物或細胞、該微生物或細胞的處理物、和/或對該微生物或細胞進行培養而得到的含有該酶的培養液與l-哌可酸發生作用。如后所述,本發明的制造方法優選在α-酮戊二酸和二價鐵離子的存在下進行。
本發明中使用的α-酮戊二酸依賴型l-哌可酸羥化酶(以下有時稱為“本發明的l-哌可酸羥化酶”)將l-哌可酸羥化時的位置選擇性和立體選擇性高,因此,通過使用該酶,能夠高效地得到光學純度高的順式-5-羥基-l-哌可酸。
本發明的α-酮戊二酸依賴型l-哌可酸羥化酶只要是具有將l-哌可酸轉變為順式-5-羥基-l-哌可酸的能力的酶則沒有特別限制,優選為具有序列編號4或11記載的氨基酸序列的酶、或為該氨基酸序列的同源物且具有α-酮戊二酸依賴型l-哌可酸羥化酶活性的酶。即,本發明的l-哌可酸羥化酶優選為包含下述(a)、(b)或(c)所示的多肽的酶。
(a)具有序列編號4或11所表示的氨基酸序列的多肽;
(b)具有在序列編號4或11所表示的氨基酸序列中缺失、置換和/或添加了1個或幾個氨基酸而得到的氨基酸序列、且具有α-酮戊二酸依賴型l-哌可酸羥化活性的多肽;或
(c)具有與序列編號4或11所表示的氨基酸序列具有60%以上的同一性的氨基酸序列、且具有α-酮戊二酸依賴型l-哌可酸羥化活性的多肽。
作為本發明中可使用的、具有序列編號4或11記載的氨基酸序列的α-酮戊二酸依賴型l-哌可酸羥化酶的同源物,如上述(b)所述,只要保持α-酮戊二酸依賴型l-哌可酸羥化活性,可以列舉具有在序列編號4或11記載的氨基酸序列中缺失、置換或添加了1個或幾個氨基酸而得到的氨基酸序列的同源物。在此,“1個或幾個氨基酸”例如為1個~100個、優選為1個~50個、更優選為1個~20個、進一步優選為1個~10個、特別優選為1個~5個氨基酸。
另外,作為上述同源物,如上述(c)所述,只要保持α-酮戊二酸依賴型l-哌可酸羥化活性,也可以為與序列編號4或11所示的氨基酸序列全長具有至少60%以上、優選80%以上、更優選90%以上、進一步優選95%、特別優選99%以上的序列同一性的蛋白質。
序列編號4記載的氨基酸序列是基于xenorhabdusdoucetiaefrm16株的公知的基因組信息而得到的氨基酸序列。
序列編號11記載的氨基酸序列是基于利用使用pcr的公知方法從xenorhabdusromaniistr.pr06-a株克隆化而得的基因信息而得到的氨基酸序列。
包含序列編號4或11的氨基酸序列及其同源物的本發明的l-哌可酸羥化酶可選擇性地將l-哌可酸的5位碳原子羥化,因此能夠以高效率生成順式-5-羥基-l-哌可酸。
需要說明的是,在本發明的制造方法中,也可以并用多個α-酮戊二酸依賴型l-哌可酸羥化酶。
本發明中可使用的α-酮戊二酸依賴型l-哌可酸羥化酶可以由xenorhabdusdoucetiaefrm16株或xenorhabdusromaniistr.pr06-a株純化得到,也可以通過對于編碼α-酮戊二酸依賴型l-哌可酸羥化酶的dna利用pcr、雜交等公知的方法將該基因克隆化、并使其在適當的宿主中進行表達而得到。
作為編碼具有序列編號4或11所示的氨基酸序列的α-酮戊二酸依賴型l-哌可酸羥化酶的dna,可以分別列舉包含序列編號1、9或10的堿基序列的dna,只要編碼具有α-酮戊二酸依賴型l-哌可酸羥化活性的蛋白質,也可以為包含序列編號1、9或10的堿基序列的dna的同源物。即,作為本發明的編碼l-哌可酸羥化酶的dna,可以列舉下述(d)、(e)或(f)所示的堿基序列。
(d)具有序列編號1、9或10所表示的堿基序列的dna;
(e)包含在序列編號1、9或10所表示的堿基序列中置換、缺失和/或添加了1個或幾個堿基而得到的堿基序列、且編碼具有α-酮戊二酸依賴型l-哌可酸羥化活性的多肽的dna;或
(f)包含在嚴格條件下與序列編號1、9或10所表示的堿基序列的互補鏈雜交的堿基序列、且編碼具有α-酮戊二酸依賴型l-哌可酸羥化活性的多肽的dna。
作為同源物,可以列舉例如如上述(e)所述包含在序列編號1、9或10的堿基序列中置換、缺失或添加1個或幾個堿基而得到的堿基序列的dna。在此所述的1個或幾個堿基例如為1個~300個、優選為1個~150個、更優選為1個~60個、進一步優選為1個~30個、特別優選為1個~15個堿基。
需要說明的是,序列編號1是為了用于大腸桿菌表達而對編碼序列編號4的氨基酸序列的xenorhabdusdoucetiaefrm16株的基因進行密碼子優化而得到的堿基序列。這樣根據作為轉化對象的宿主的不同對密碼子進行了優化的dna當然也包含在本發明中可使用的編碼α-酮戊二酸依賴型l-哌可酸羥化酶的dna中。
此外,作為上述dna的同源物,如上述(f)所述,只要編碼具有α-酮戊二酸依賴型l-哌可酸羥化活性的蛋白質,可以為在嚴格條件下與序列編號1、9或10的堿基序列的互補鏈雜交的dna。在此,“在嚴格條件下雜交的堿基序列”是指使用dna作為探針,通過在嚴格條件下利用菌落雜交法、噬菌斑雜交法或southern印記雜交法等而得到的dna的堿基序列。作為嚴格條件,可以列舉例如下述條件:在菌落雜交法和噬菌斑雜交法中,使用固定有來源于菌落或噬菌斑的dna或該dna的片段的過濾器,在0.7mol/l~1.0mol/l的氯化鈉水溶液的存在下、在65℃進行雜交后,使用0.1~2×ssc溶液(1×ssc的組成為150mmol/l氯化鈉水溶液、15mmol/l檸檬酸鈉水溶液),在65℃的條件下對過濾器進行清洗。
各雜交可以基于molecularcloning:alaboratorymannual(分子克隆實驗指南),第2版,冷泉港實驗室,冷泉港,紐約,1989.等記載的方法進行。
本領域技術人員可以通過使用定點突變法(nucleicacidsres.10,pp.6487(1982)、methodsinenzymol.100,pp.448(1983)、molecularcloning、pcrapracticalapproachirlpresspp.200(1991))等在序列編號1、9或10的dna中適當導入置換、缺失、插入和/或添加突變而得到如上所述的dna同源物。
另外,也可以基于序列編號4或11的氨基酸序列或其一部分、序列編號1、9或10所表示的堿基序列或其一部分,對例如dnadatabankofjapan(日本dna數據庫)(ddbj)等數據庫進行同源性檢索而獲得α-酮戊二酸依賴型l-哌可酸羥化酶的氨基酸信息或編碼該酶的dna的堿基序列信息。
在本發明的羥基-l-哌可酸的制造方法中,可以將α-酮戊二酸依賴型l-哌可酸羥化酶直接用于反應,但優選使用含有α-酮戊二酸依賴型l-哌可酸羥化酶的微生物或細胞、該微生物或細胞的處理物、和/或對該微生物或細胞進行培養而得到的含有該酶的培養液。
作為含有α-酮戊二酸依賴型l-哌可酸羥化酶的微生物或細胞,可以使用原本就具有α-酮戊二酸依賴型l-哌可酸羥化酶的微生物或細胞,但優選使用利用編碼α-酮戊二酸依賴型l-哌可酸羥化酶的基因進行了轉化的微生物或細胞。
另外,作為含有α-酮戊二酸依賴型l-哌可酸羥化酶的微生物或細胞的制備物,例如可以使用將該微生物或細胞用丙酮、二甲亞砜(dmso)、甲苯等有機溶劑、表面活性劑進行處理而得到的制備物、進行冷凍干燥處理而得到的制備物、利用物理方法或利用酶進行破碎而得到的制備物等微生物或細胞的制備物、將微生物或細胞中的酶級分以粗制物或純化物的形式取出而得到的制備物、以及將這些制備物固定于以聚丙烯酰胺凝膠、卡拉膠等為代表的載體上而得到的制備物等。
作為對含有α-酮戊二酸依賴型l-哌可酸羥化酶的微生物或細胞進行培養而得到的培養液,例如可以使用該微生物或細胞與液體培養基的混懸液,在該微生物或細胞為分泌表達型微生物或細胞的情況下,可以使用通過離心分離等除去該微生物或細胞后而得到的上清或其濃縮物。
通過將如上所述分離出的編碼α-酮戊二酸依賴型l-哌可酸羥化酶的dna以可表達的方式插入到公知的表達載體中,可提供α-酮戊二酸依賴型l-哌可酸羥化酶表達載體。然后,通過利用該表達載體對宿主細胞進行轉化,能夠得到導入有編碼α-酮戊二酸依賴型l-哌可酸羥化酶的dna的轉化體。轉化體也可以通過利用同源重組等方法將編碼α-酮戊二酸依賴型l-哌可酸羥化酶的dna以可表達的狀態整合到宿主的染色體dna中而得到。
作為轉化體的制作方法,具體而言,可以例示下述方法:將編碼α-酮戊二酸依賴型l-哌可酸羥化酶的dna導入到在微生物等宿主細胞中穩定存在的質粒載體、噬菌體載體、病毒載體中并將所構建的表達載體導入到該宿主細胞中,或者直接將該dna導入到宿主基因組中,對其遺傳信息進行轉錄、翻譯。此時,優選在宿主中在dna的5’-側上游連接適當的啟動子,更優選進一步在3’-側下游連接終止子。作為這樣的啟動子和終止子,只要是已知在用作宿主的細胞中發揮功能的啟動子和終止子則沒有特別限定,例如,在“微生物學基礎講座8基因工程、共立出版”等中詳細記載了在宿主微生物中可利用的載體、啟動子和終止子。
作為用于表達α-酮戊二酸依賴型l-哌可酸羥化酶的作為轉化對象的宿主微生物,只要宿主本身不會對l-哌可酸的反應造成不利影響則沒有特別限定,具體可以列舉如下所示的微生物。
屬于埃希氏菌(escherichia)屬、芽孢桿菌(bacillus)屬、假單胞菌(pseudomonas)屬、沙雷氏菌(serratia)屬、短桿菌(brevibacterium)屬、棒狀桿菌(corynebacterium)屬、鏈球菌(streptococcus)屬、乳桿菌(lactobacillus)屬等的已建立了宿主載體系統的細菌。
屬于紅球菌(rhodococcus)屬、鏈霉菌(streptomyces)屬等的已建立了宿主載體系統的放線菌。屬于酵母菌(saccharomyces)屬、克魯維酵母(kluyveromyces)屬、裂殖酵母(schizosaccharomyces)屬、接合酵母(zygosaccharomyces)屬、解脂耶氏酵母菌(yarrowia)屬、絲孢酵母菌(trichosporon)屬、紅冬孢酵母(rhodosporidium)屬、漢遜酵母(hansenula)屬、畢赤酵母(pichia)屬、念珠菌(candida)屬等已建立了宿主載體系統的酵母。
屬于脈孢菌(neurospora)屬、曲霉(aspergillus)屬、頭孢霉(cephalosporium)屬、木霉(trichoderma)屬等的已建立了宿主載體系統的霉菌。
用于轉化體制作的程序、適合于宿主的重組載體的構建和宿主的培養方法可以基于分子生物學、生物工程、基因工程的領域中慣用的技術來進行(例如,molecularcloning(分子克隆)中記載的方法)。
以下,具體地列舉優選的宿主微生物、各微生物的優選轉化方法、載體、啟動子、終止子等的示例,但本發明不限定于這些示例。
在埃希氏菌屬、特別是大腸桿菌(escherichiacoli)中,作為質粒載體,可以列舉pbr、puc系質粒等,可以列舉lac(β-半乳糖苷酶)、trp(色氨酸操縱子)、tac、trc(lac、trp的融合)、λ噬菌體pl、pr等來源的啟動子等。另外,作為終止子,可以列舉trpa來源、噬菌體來源、rrnb核糖體rna來源的終止子等。
在芽孢桿菌屬中,作為載體,可以列舉pub110系質粒、pc194系質粒等,另外,也可以整合到染色體中。作為啟動子和終止子,可以利用堿性蛋白酶、中性蛋白酶、α-淀粉酶等的酶基因的啟動子、終止子等。
在假單胞菌屬中,作為載體,可以列舉在惡臭假單胞菌(pseudomonasputida)、洋蔥假單胞菌(pseudomonascepacia)等中建立的通常的宿主載體系統、參與甲苯化合物的分解的質粒、以tol質粒為基礎的廣宿主載體(包含來源于rsf1010等的自主復制所需的基因)pkt240(gene,26,273-82(1983))等。
在短桿菌屬、特別是乳糖發酵短桿菌(brevibacteriumlactofermentum)中,作為載體,可以列舉paj43(gene39,281(1985))等質粒載體。作為啟動子和終止子,可以利用在大腸桿菌中使用的各種啟動子和終止子。
在棒狀桿菌屬、特別是谷氨酸棒桿菌(corynebacteriumglutamicum)中,作為載體,可以列舉pcs11(日本特開昭57-183799號公報)、pcb101(mol.gen.genet.196,175(1984))等質粒載體。
在酵母菌(saccharomyces)屬、特別是釀酒酵母菌(saccharomycescerevisiae)中,作為載體,可以列舉yrp系、yep系、ycp系、yip系質粒等。另外,可以利用醇脫氫酶、甘油醛-3-磷酸脫氫酶、酸性磷酸酶、β-半乳糖苷酶、磷酸甘油酸激酶、烯醇酶等各種酶基因的啟動子、終止子。
在裂殖酵母(schizosaccharomyces)屬中,作為載體,可以列舉mol.cell.biol.6,80(1986)中記載的粟酒裂殖酵母來源的質粒載體等。特別是paur224由寶生物株式會社銷售,可以容易地利用。
在曲霉(aspergillus)屬中,黑曲霉(aspergillusniger)、米曲霉(aspergillusoryzae)等在霉菌中研究得最多,可用于向質粒、染色體中整合,可以利用菌體外蛋白酶、淀粉酶來源的啟動子(trendsinbiotechnology7,283-287(1989))。
另外,除了上述以外,已建立了適應于各種微生物的宿主載體系統,可以適當使用這些宿主載體系統。
另外,除了微生物以外,在植物、動物中也建立了各種宿主、載體系統,特別是建立了在昆蟲(例如蠶)等動物中(nature315,592-594(1985))、菜籽、玉米、土豆等植物中大量表達異種蛋白質的系統、使用大腸桿菌無細胞提取液或小麥胚芽等的無細胞蛋白質合成系統的系統,可以適當利用。
在本發明的制造方法中,通過使α-酮戊二酸依賴型l-哌可酸羥化酶、具有生產該酶的能力的微生物或細胞、該微生物或細胞的處理物、和/或對該微生物或細胞進行培養而得到的含有該酶的培養液在α-酮戊二酸的存在下與作為反應底物的l-哌可酸發生作用,能夠制造順式-5-羥基-l-哌可酸。
本發明的制造方法只要使α-酮戊二酸、以及α-酮戊二酸依賴型l-哌可酸羥化酶、具有生產該酶的能力的微生物或細胞、該微生物或細胞的處理物、和/或對該微生物或細胞進行培養而得到的含有該酶的培養液與l-哌可酸發生作用,則沒有特別限制,但通常優選在水性介質中或該水性介質與有機溶劑的混合物中進行。本發明的制造方法優選進一步在二價鐵離子的存在下進行。
作為上述水性介質,可以列舉例如水或緩沖液。
另外,作為上述有機溶劑,可以使用甲醇、乙醇、1-丙醇、2-丙醇、1-丁醇、叔丁醇等醇類溶劑、丙酮、二甲亞砜等反應底物的溶解度高的溶劑。另外,作為上述有機溶劑,也可以使用對除去反應副產物等有效的乙酸乙酯、乙酸丁酯、甲苯、氯仿、正己烷等。
作為反應底物的l-哌可酸通常在底物濃度為0.01%w/v~90%w/v、優選為0.1%w/v~30%w/v的范圍內使用。反應底物可以在反應開始時一次性添加,但從減少存在酶的底物抑制時的影響的觀點和提高產物的蓄積濃度的觀點出發,優選連續地或間歇地添加。
α-酮戊二酸通常在與底物為等摩爾或更多、優選為等摩爾~1.2倍摩爾的范圍內添加。α-酮戊二酸可以在反應開始時一次性添加,但從減少對酶具有抑制作用時的影響的觀點和提高產物的蓄積濃度的觀點出發,優選連續地或間歇地添加。另外,也可以添加葡萄糖等宿主可代謝的廉價的化合物來代替α-酮戊二酸,使宿主進行代謝,將該過程中產生的α-酮戊二酸用于反應。
本發明的制造方法優選在二價鐵離子的存在下進行。二價鐵離子通常優選在0.01mmol/l~100mmol/l、優選在0.1mmol/l~10mmol/l的范圍內使用。二價鐵離子可以以硫酸鐵等的形式在反應開始時一次性添加,但在反應中添加的二價鐵離子被氧化為三價、形成沉淀而減少的情況下,追加添加也是有效的。另外,在本發明的l-哌可酸羥化酶、具有生產該酶的能力的微生物或細胞、該微生物或細胞的處理物、和/或對該微生物或細胞進行培養而得到的含有該酶的培養液中已經含有足夠量的二價鐵離子的情況下,也不是一定要進行添加。
反應在通常為4℃~60℃、優選為15℃~45℃、特別優選為20℃~40℃的反應溫度下在通常為ph3~ph11、優選為ph5~ph8下進行。反應時間通常為約1小時~約72小時。
關于向反應液中添加的微生物或細胞、該微生物或細胞的處理物、和/或對該微生物或細胞進行培養而得到的含有該酶的培養液的量,例如在添加細胞的情況下,按照該細胞的濃度以濕菌體重量計通常為約0.1%w/v~約50%w/v、優選為1%w/v~20%w/v的方式添加到反應液中,在使用處理物的情況下,求出酶的比活性,以添加后達到上述細胞濃度的量進行添加。
通過本發明的制造方法生成的羥基-l-哌可酸可以通過在反應結束后利用離心分離、膜處理等對反應液中的菌體、蛋白質等進行分離、然后將利用1-丁醇、叔丁醇等有機溶劑進行的萃取、蒸餾、使用離子交換樹脂或硅膠等的柱層析、等電點晶析或利用一鹽酸鹽、二鹽酸鹽、鈣鹽等進行的晶析等適當組合而進行純化。
[實施例]
以下,利用實施例進一步詳細地對本發明進行說明,但本發明并不限定于此。
實施例1
α-酮戊二酸依賴型哌可酸羥化酶基因的克隆
由dna2.0公司人工合成了編碼xenorhabdusdoucetiaefrm16株來源的推測的l-脯氨酸順式-4-羥化酶xdph(genbank登錄號cdg16639、序列編號4)的為了用于大腸桿菌表達而進行了密碼子優化的基因序列(xdph_ecodon、序列編號1),插入到pjexpress411(dna2.0)中,制作成質粒pj411xdph。
同樣地,也進行了公知的顯示出哌可酸5位羥化活性的代表性的酶基因的克隆。由dna2.0公司人工合成了編碼segniliparusrugosusnbrc101839株來源的l-哌可酸順式-5-羥化酶sruph(genbank登錄號efv12517、序列編號6)和苜蓿中華根瘤菌(sinorhizobiummeliloti)1021株來源的l-脯氨酸順式-4-羥化酶smph(genbank登錄號cac47686、序列編號5)的、為了用于大腸桿菌表達而進行了密碼子優化的基因序列sruph_ecodon(序列編號3)和smph_ecodon(序列編號2),分別插入到pjexpress411(dna2.0)和pjexpress401中,制作成質粒pj411sruph、pj401smph。合成針對smph_ecodon的引物smph_f(序列編號7)和smph_r(序列編號8),使用它們以質粒dna為模板按照常規方法進行pcr反應,得到約1.0kbp的dna片段。利用限制性內切酶ndei、hindiii對所得到的dna片段進行消化,按照常規方法與利用ndei、hindiii消化后的pet24a(novagen)連接,由此分別得到pet24smph。
實施例2
α-酮戊二酸依賴型哌可酸羥化酶基因表達菌體的獲取和表達量的確認
接著,使用所得到的各質粒按照常規方法對大腸桿菌(escherichiacoli)bl21(de3)(invitrogen制)進行轉化,得到重組大腸桿菌bl21(de3)/pj411xdph、bl21(de3)/pj411sruph、bl21(de3)/pet24smph。為了得到表達所導入的基因的菌體,對于各重組大腸桿菌,使用含有卡那霉素和lac啟動子誘導物質的液體lb培養基在28℃下培養4小時~6小時后,在15℃下進一步培養約40小時,然后收集菌體。
將所得到的重組大腸桿菌以濁度od630為約10的方式懸浮到ph7的50mmol/lmes(2-嗎啉乙磺酸)緩沖液中。在冰上對該懸浮液0.5ml進行超聲波破碎處理,然后以12000rpm的轉速進行離心分離,由此分離成上清和殘渣。將所得到的上清作為可溶性級分,將殘渣作為不溶性級分。
按照常規方法對所得到的可溶性級分和不溶性級分進行處理后,利用sds-聚丙烯酰胺電泳法確認所表達的蛋白質表達量。將結果示于圖1。
實施例3
α-酮戊二酸依賴型哌可酸羥化酶的活性確認
在塑料管內將5mmol/ll-哌可酸、10mmol/lα-酮戊二酸、1mmol/ll-抗壞血酸、0.5mmol/l硫酸鐵、以及以約2mg/ml的蛋白質濃度添加有實施例2中得到的各粗制酶液的液體0.2ml在30℃下振蕩1小時。
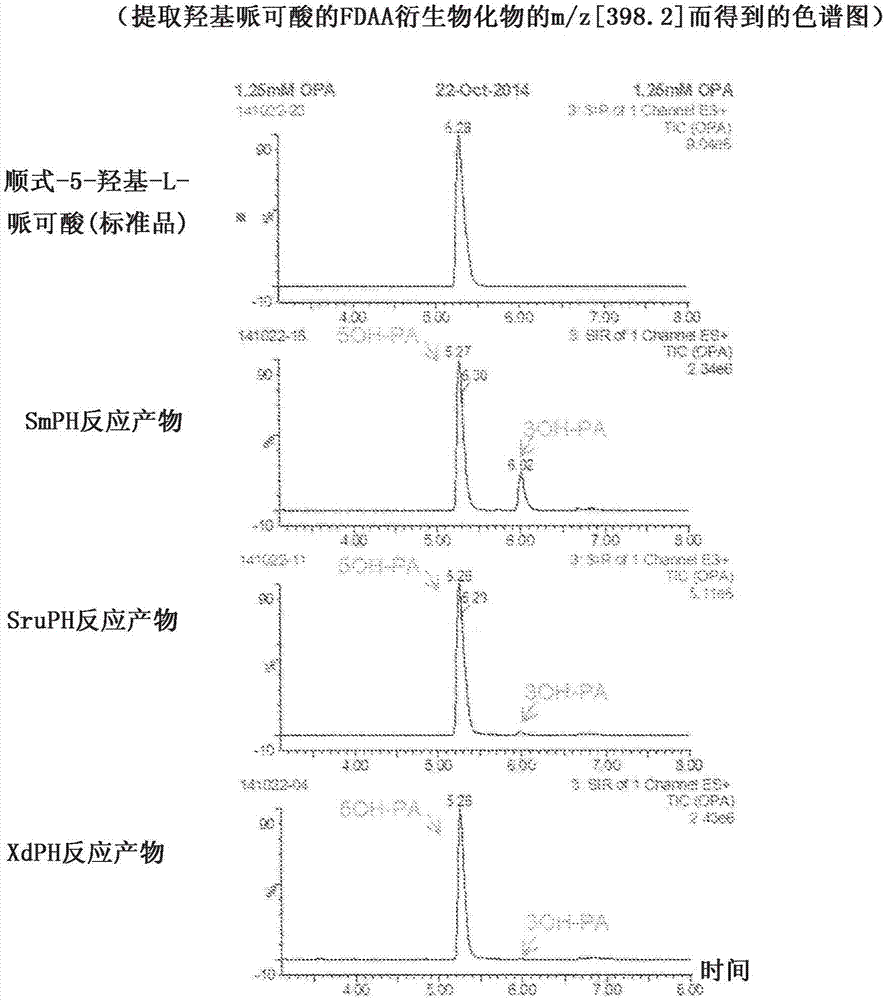
利用1-氟-2,4-二硝基苯基-5-l-丙氨酰胺(fdaa)使反應產物形成衍生物,然后利用uplc‐ms(waters制)進行分析。結果如圖2所示,確認到由使用各粗制酶液進行了反應后的液體生成了與5-羥基哌可酸標準品的保留時間5.3分鐘一致的化合物。各酶液顯示出的5-羥基哌可酸生成活性(u/g)以單位蛋白質量(g)計分別算出為4.1u/g、8.9u/g、3.1u/g。此處的單位(u)表示在1分鐘內生成1微摩爾的底物的能力。
另外,確認到由smph反應產物生成了認為是順式-3-羥基哌可酸的保留時間為6.0分鐘的化合物。將利用色譜圖上的羥基哌可酸峰的面積值對順式-3-羥基哌可酸在總羥基哌可酸量中所占的比例進行比較的結果示于表2。可知xdph反應產物中僅生成了0.17%的順式-3-羥基哌可酸。
需要說明的是,利用hplc的羥基哌可酸的分析條件如下述表1所示。
實施例4
羥化反應的溫度依賴性的確認
為了確認各酶反應的溫度依賴性,將各羥化酶的粗制酶液在各種溫度下溫育1小時30分鐘后,測定在30℃下反應1小時后的5-羥基哌可酸生成量。將所得到的結果以將各酶的最高活性值設為100(%)時的相對活性的形式示于圖3。
表1
a)lc的設定
b)洗脫條件
c)ms的條件
表2
實施例5
α-酮戊二酸依賴型哌可酸羥化酶基因的克隆
以xenorhabdusdoucetiaefrm16株及其近緣種xenorhabdusromaniistr.pr06-a株來源的染色體dna作為模板,使用擴增推測的l-脯氨酸順式-4-羥化酶xdph基因(xdphori:genbank登錄號fo704550-xdd1_0936、序列編號9)的引物xdphfus-f(序列編號12)和xdphfus-r(序列編號13),利用聚合酶鏈式反應(pcr)分別擴增出約1kbp的dna片段。使用in-fusionhd克隆試劑盒(寶生物株式會社制)按照常規方法將所得到的兩種dna片段插入到利用限制性內切酶ncoi、hindiii消化后的pqe60(qiagen)中,分別得到pqexdphori、pqexrphori。
接著,使用所得到的各質粒,按照常規方法對大腸桿菌(escherichiacoli)jm109進行轉化,得到重組大腸桿菌jm109/pqexdphori和jm109/pqexrphori。按照常規方法對插入到pqe60中的基因序列進行確認,決定了xenorhabdusromaniistr.pr06-a株來源的氨基酸羥化酶的基因序列(xrph_ori、序列編號10)和氨基酸序列(xrph、序列編號11)。xrph相對于xdph具有99%的同一性。
為了得到表達所導入的基因的菌體,使用含有氨芐青霉素的液體lb培養基對各重組大腸桿菌培養一夜后,收集菌體。
在塑料管內添加20mmol/ll-哌可酸、15mmol/lα-酮戊二酸、10mmol/l三(2-羧乙基)膦鹽酸鹽(tcep)、0.5mmol/l硫酸鐵、10mmol/l檸檬酸鈉、1%nymeen、以及將由所得到的重組大腸桿菌jm109/pqexdphori、jm109/pqexrphori的培養液0.4ml得到的菌體用ph6.5的50mmol/l雙(2-羥乙基)亞氨基三(羥甲基)甲烷緩沖液懸浮而得到的液體0.2ml,制成總容量為1ml的反應液,使其在20℃下振蕩20小時。
使用accq·tag(waters公司制)使反應后的液體形成衍生物后,以下述條件的hplc條件進行分析,測定所生成的5-羥基-l-哌可酸。
柱:xbridgec185μm(2.1×150mm)(waters公司制)
洗脫液a:10mmol/l乙酸銨(ph5)
洗脫液b:甲醇(0~0.5分鐘
流速:0.3ml/分鐘
檢測:熒光檢測器
溫度:30℃
將測定結果示于表3。確認到不僅xenorhabdusdoucetiaefrm16株來源的羥化酶xdph保持了哌可酸5位羥化活性,xenorhabdusromaniistr.pr06-a株來源的羥化酶也保持了哌可酸5位羥化活性。
表3
序列表
<110>株式會社api(apicorporation)
<120>順式-5-羥基-l-哌可酸的制造方法(methodofproducingcis-5-hydroxy-l-pipecolicacid)
<130>ap351-15399
<150>jp2014-229685
<151>2014-11-12
<160>13
<170>patentinversion3.5
<210>1
<211>882
<212>dna
<213>artificalsequence
<220>
<223>xdph_ecodon
<400>1
atgatgagcgcaaaactgctggcaagcattgaattgaaccaagaacagatcgagcatgat60
ctgaatattgttggtagcgagatcctggacgtggcgtacagcgagtatgcgtgcggcaat120
tggggtaccattaccctgtggaaccacagcggcgatgctggcgaccagacgagccgcgaa180
tacgttggtcaggcccgtccgactgagctgggccagcaattagactgcattaatcagctg240
atccgtaacaatttcaacatcagcctgatcaagagcgtgcgcatcttccgtagctataac300
ggtggtgcgatctatccgcacattgactacttggaattcaaccaaggttttaagcgcgtg360
cacctggttctgaaatccgaccgttcatgtctgaatagcgaagagaacacggtttatcac420
atgctgcctggtgaagtgtggtttgtcgatggtcatagcgcgcactcggcgatgagcctg480
agccgtgtcggcaagtactcgctggtcctggactttgattctggcgccaaattcgaagat540
ctgtattctgagagccacaccctgtgtgttgataacctggagccggacattatccatgac600
cgccagccactgccgaccagcctgcgtgatagcctggcacacattgctgagcatgcggat660
gaattcaatatccaatccattctgttcctggccacccgttttcactttagctacgcagtg720
agcattcgtgagtacttccaactcctggacgagtgcttttctcgcaacccgtacaagtcc780
gttcgcgagcgttacgaagcgctgaaagacattttggtgcgtagcggttataccagccac840
aatgtcaatcatttcaacagcttgtccggtgtcacgatcggc882
<210>2
<211>843
<212>dna
<213>artificalsequence
<220>
<223>smph_ecodon
<400>2
atgagcacccattttctcggcaaggtaaagtttgatgaagcgcgtttggcagaggacttg60
agcaccctggaagtggctgagtttagcagcgcctacagcgactttgcatgcggcaaatgg120
gaagcgtgcgtcctgcgcaaccgcaccggtatgcaagaagaagatatcgttgttagccac180
aatgctccggcgctggcgacgccgctgagcaagtccctgccgtatctgaacgagctggtg240
gaaacccatttcgactgtagcgccgtgcgttacacgcgcattgttcgtgtctctgagaac300
gcatgtatcattccgcattctgactatctggagttggatgaaacgttcacgcgcctgcac360
ctggttctggacacgaatagcggttgtgcgaataccgaagaggataagattttccacatg420
ggtctgggcgagatctggttcctggatgctatgctgccgcacagcgccgcatgctttagc480
aaaactccgcgtctgcacttgatgattgatttcgaggcgaccgcgttcccagagagcttt540
ttgcgtaacgttgagcagccggtcaccacccgtgatatggtggacccgcgtaaagagctg600
accgacgaggtgatcgaaggtatcctgggcttttccatcatcattagcgaggcaaattac660
cgtgagattgtctccattctggcgaaactgcacttcttttacaaggcggattgccgcagc720
atgtacgactggctgaaagaaatctgcaaacgtcgtggtgaccctgccctgattgaaaag780
accgcgtcgctggagcgcttcttcctgggtcatcgtgaacgcggtgaggttatgacgtat840
taa843
<210>3
<211>897
<212>dna
<213>artificalsequence
<220>
<223>sruph_ecodon
<400>3
atgaaaagctattccttgggcaagtttgaggaccgttctattgacagcctgatcgaagaa60
gcgtccggtctgccggacagcgcgtattcttctgcgtaccaagagtacagcatcggtctg120
tgggacaccgccacgctgtggaacgaacgtggcaacgagagcggcgaggttagcgagcac180
gcggcagccgctgcgccgaccgcaattggtcgtagcaccccgcgtctgaacgaatttgta240
cgtgccaagttcaatgtcgatgtcctgcgtgcggttcgcttgttcagagcgcgtcagggt300
gcaattatcatcccgcaccgcgattacctggagcatagcaatggtttttgccgtattcac360
cttccgctggttacgaccccgggtgcgcgtaatagcgagaataacgaagtgtatcgcatg420
ctgcctggcgagctgtggttcctggattcaaacgaagtgcacagcggtggcgttctggac480
agcggtacgcgcattcacctcgttttagatttcacccacgagcataatgagaatccggct540
gcagtgctgaagaacgccgaccgtttgcgtccgatcgcgcgtgacccgcgcatctcgcgc600
tccaaactggatcacgaagcgctggagagcctgatccgcggtggccgtgtcgtcactctg660
gcgatgtggccagctctggtgcagatgctggcacgcattcatctgaccagcgacgcacat720
ccggccgagctgtacgactggttggatgacctggcggaccgcagcggcaacgatgagctg780
gttgcagaagcgcgtcgtatgcgtcgttactttctgaccgatggtattagccgtacgccg840
agcttcgaacgcttttggcgcgaactggatgccgctcgtaaaggtgagttggtgagc897
<210>4
<211>294
<212>prt
<213>xenorhabdusdoucetiaefrm16
<400>4
metmetseralalysleuleualaserilegluleuasnglnglugln
151015
ilegluhisaspleuasnilevalglysergluileleuaspvalala
202530
tyrserglutyralacysglyasntrpglythrilethrleutrpasn
354045
hisserglyaspalaglyaspglnthrserargglutyrvalglygln
505560
alaargprothrgluleuglyglnglnleuaspcysileasnglnleu
65707580
ileargasnasnpheasnileserleuilelysservalargilephe
859095
argsertyrasnglyglyalailetyrprohisileasptyrleuglu
100105110
pheasnglnglyphelysargvalhisleuvalleulysserasparg
115120125
sercysleuasnserglugluasnthrvaltyrhismetleuprogly
130135140
gluvaltrpphevalaspglyhisseralahisseralametserleu
145150155160
serargvalglylystyrserleuvalleuasppheaspserglyala
165170175
lysphegluaspleutyrsergluserhisthrleucysvalaspasn
180185190
leugluproaspileilehisaspargglnproleuprothrserleu
195200205
argaspserleualahisilealagluhisalaaspglupheasnile
210215220
glnserileleupheleualathrargphehisphesertyralaval
225230235240
serileargglutyrpheglnleuleuaspglucyspheserargasn
245250255
protyrlysservalarggluargtyrglualaleulysaspileleu
260265270
valargserglytyrthrserhisasnvalasnhispheasnserleu
275280285
serglyvalthrilegly
290
<210>5
<211>280
<212>prt
<213>sinorhizobiummeliloti1021
<400>5
metserthrhispheleuglylysvallyspheaspglualaargleu
151015
alagluaspleuserthrleugluvalalaglupheserseralatyr
202530
seraspphealacysglylystrpglualacysvalleuargasnarg
354045
thrglymetglnglugluaspilevalvalserhisasnalaproala
505560
leualathrproleuserlysserleuprotyrleuasngluleuval
65707580
gluthrhispheaspcysseralavalargtyrthrargilevalarg
859095
valsergluasnalacysileileprohisserasptyrleugluleu
100105110
aspgluthrphethrargleuhisleuvalleuaspthrasnsergly
115120125
cysalaasnthrglugluasplysilephehismetglyleuglyglu
130135140
iletrppheleuaspalametleuprohisseralaalacyspheser
145150155160
lysthrproargleuhisleumetileasppheglualathralaphe
165170175
progluserpheleuargasnvalgluglnprovalthrthrargasp
180185190
metvalaspproarglysgluleuthraspgluvalilegluglyile
195200205
leuglypheserileileileserglualaasntyrarggluileval
210215220
serileleualalysleuhisphephetyrlysalaaspcysargser
225230235240
mettyrasptrpleulysgluilecyslysargargglyaspproala
245250255
leuileglulysthralaserleugluargphepheleuglyhisarg
260265270
gluargglygluvalmetthrtyr
275280
<210>6
<211>299
<212>prt
<213>segniliparusrugosusatccbaa-974
<400>6
metlyssertyrserleuglylysphegluaspargserileaspser
151015
leuilegluglualaserglyleuproaspseralatyrserserala
202530
tyrglnglutyrserileglyleutrpaspthralathrleutrpasn
354045
gluargglyasngluserglygluvalsergluhisalaalaalaala
505560
alaprothralaileglyargserthrproargleuasnglupheval
65707580
argalalyspheasnvalaspvalleuargalavalargleuphearg
859095
alaargglnglyalaileileileprohisargasptyrleugluhis
100105110
serasnglyphecysargilehisleuproleuvalthrthrprogly
115120125
alaargasnsergluasnasngluvaltyrargmetleuproglyglu
130135140
leutrppheleuaspserasngluvalhisserglyglyvalleuasp
145150155160
serglythrargilehisleuvalleuaspphethrhisgluhisasn
165170175
gluasnproalaalavalleulysasnalaaspargleuargproile
180185190
alaargaspproargileserargserlysleuasphisglualaleu
195200205
gluserleuileargglyglyargvalvalthrleualamettrppro
210215220
alaleuvalglnmetleualaargilehisleuthrseraspalahis
225230235240
proalagluleutyrasptrpleuaspaspleualaaspargsergly
245250255
asnaspgluleuvalalaglualaargargmetargargtyrpheleu
260265270
thraspglyileserargthrproserphegluargphetrpargglu
275280285
leuaspalaalaarglysglygluleuvalser
290295
<210>7
<211>36
<212>dna
<213>artificalsequence
<220>
<223>primer
<400>7
tttcatatgagcacccattttctcggcaaggtaaag36
<210>8
<211>33
<212>dna
<213>artificalsequence
<220>
<223>primer
<400>8
tttaagcttttaatacgtcataacctcaccgcg33
<210>9
<211>858
<212>dna
<213>xenorhabdusdoucetiaefrm16
<400>9
atgagaacgcattttgtgggtgaagttgcattggacctggcacgtctggaggctgatctg60
gcaacctgtcgtagcctggagtggagcgaagcgtattcggattacgtctttggtggtagc120
tggaaaagctgcatgctgtgggcgcctggtggcgatgccggcgatggtgttgtgacggac180
tacgcgtacgaccgtagcgcgggttttacgccgcacgcagagcgcctgccgtatctggca240
gagcttattcgtgagactgcagatctggatcgcctgaatttcgcgcgcttggcgctggta300
accaactctgtgattatcccgcaccgtgacctgttggaattaagcgacttgccggacgaa360
gctcgcaatgaacaccgtatgcacattccgctggcgaccaatgataactgcttcttcaac420
gaggacaacgttgtgtatcgtatgcgtcgtggtgaagtttggtttctggatgccagccgt480
attcatagcgtcgctgtcctgacggcgcagccacgtatccatctgatgctggatttcgtg540
gacaccccgggtgcgggcagcttcacgcgcgttgcgggtggcggcgttgaggccggcatc600
ccggtcgatcgcatcgtgacccgtccgccgctgggcgacgacgagcgtgcggacctgttc660
ggtgtcgccccactcctgtccatggataccttcgacgaagtgttttccattgttatcaag720
aaacacttccgtcgtgatggtggcagcgactttgtgtgggacaccatgctggaactggcg780
gcgaagtctccggatccggccgtcctgccgcataccgaagaactgcgcaagcactacacc840
ctggaccgcagcgcataa858
<210>10
<211>885
<212>dna
<213>xenorhabdusromaniipr06-a
<400>10
atgatgagtgcaaaattgttggccagcattgaactgaatcaggaacaaattgaacacgac60
ttaaatattgtaggtagtgaaattctggatgttgcatatagcgaatatgcttgtggcaac120
tggggcaccatcacgttatggaatcacagtggagacgctggtgaccaaacatcaagagag180
tatgttgggcaggctcgtcctacagagcttggacagcagttggattgtattaaccaactt240
attcgaaacaattttaatatatctctgatcaaatcagtgcgtatattccgttcttataac300
ggtggagccatttatccacatattgattatttggaattcaatcaagggttcaaacgtgtg360
catctggtgctaaaatcagaccgaagttgcctcaattccgaagagaatacagtttatcac420
atgctccctggtgaagtatggtttgttgacggtcatagtgcacacagtgcaatgtcacta480
agtcgtgtagggaaatacagtttagtgcttgattttgactctggtgctaaattcgaggat540
ttgtattccgaaagccacacgttatgtgtagacaacctagaaccagatattattcatgat600
cggcaaccattgccaacatcactgcgggattcactcgctcatatagctgagcacgccgat660
gaattcaacattcagtctatcctgtttcttgctacccgctttcactttagttatgcagtg720
tcaatcagagaatattttcaattgttagatgaatgttttagtcgtaatccgtacaaatca780
gttagagaaagatacgaggcattgaaggatattctagtccgaagtggctatacatcccat840
aagtttaaccactttaactcactgtctggagtcactattggttaa885
<210>11
<211>294
<212>prt
<213>xenorhabdusromaniipr06-a
<400>11
metmetseralalysleuleualaserilegluleuasnglnglugln
151015
ilegluhisaspleuasnilevalglysergluileleuaspvalala
202530
tyrserglutyralacysglyasntrpglythrilethrleutrpasn
354045
hisserglyaspalaglyaspglnthrserargglutyrvalglygln
505560
alaargprothrgluleuglyglnglnleuaspcysileasnglnleu
65707580
ileargasnasnpheasnileserleuilelysservalargilephe
859095
argsertyrasnglyglyalailetyrprohisileasptyrleuglu
100105110
pheasnglnglyphelysargvalhisleuvalleulysserasparg
115120125
sercysleuasnserglugluasnthrvaltyrhismetleuprogly
130135140
gluvaltrpphevalaspglyhisseralahisseralametserleu
145150155160
serargvalglylystyrserleuvalleuasppheaspserglyala
165170175
lysphegluaspleutyrsergluserhisthrleucysvalaspasn
180185190
leugluproaspileilehisaspargglnproleuprothrserleu
195200205
argaspserleualahisilealagluhisalaaspglupheasnile
210215220
glnserileleupheleualathrargphehisphesertyralaval
225230235240
serileargglutyrpheglnleuleuaspglucyspheserargasn
245250255
protyrlysservalarggluargtyrglualaleulysaspileleu
260265270
valargserglytyrthrserhislyspheasnhispheasnserleu
275280285
serglyvalthrilegly
290
<210>12
<211>47
<212>dna
<213>artificialsequence
<220>
<223>primer
<400>12
gaattcattaaagaggagaaattaaccatgatgagtgcaaaattgtt47
<210>13
<211>47
<212>dna
<213>artificialsequence
<220>
<223>primer
<400>13
caacaggagtccaagctcagctaattattaaccaatagtgactccag47
- 還沒有人留言評論。精彩留言會獲得點贊!