通過氧化切割從長鏈脂肪酸生產6?碳化學物的方法與流程
本申請要求2014年5月13日提交的美國臨時申請系列號61/992,794的優先權,其公開通過引用以其整體并入本文。
技術領域
本文公開了從十二酰-[acp]生物合成己二酰-[acp]和己酸的方法和從辛酰-[acp]生物合成己二酰-[acp]的方法。使用具有庚二酰-[acp]合酶活性的多肽和例如具有醛脫氫酶活性的多肽,如在表達一種或多種此類多肽的重組宿主細胞中生物合成該產物。本文還公開了使用具有脫氫酶,還原酶,單加氧酶,轉氨酶,N-乙酰轉移酶,脫酰基酶或硫酯酶活性的一種或多種分離的多肽,或使用表達一種或多種此類多肽的重組宿主細胞將己二酰-[acp]和/或己酸轉化為C6單體,如己二酸,6-氨基己酸,六亞甲基二胺,6-羥基己酸,己內酰胺或1,6-己二醇(下文稱為“C6結構單元”(buiding blocks))的方法。
發明背景
尼龍是聚酰胺,其有時通過二胺與二羧酸的縮聚(condensation polymerization)合成。類似地,尼龍可通過內酰胺的縮聚生成。一種普遍存在的尼龍是尼龍6,6,其通過六亞甲基二胺(HMD)和己二酸的反應生成。尼龍6可通過己內酰胺的開環聚合生成。因此,己二酸,六亞甲基二胺和己內酰胺是尼龍的生產中的重要中間體(Anton&Baird,Polyamides Fibers,Encyclopedia of Polymer Science and Technology,2001)。
工業上,己二酸和己內酰胺通過環己烷的空氣氧化生產。環己烷的空氣氧化在一系列的步驟中產生環己酮(K)和環己醇(A)的混合物,命名為KA油。KA油的硝酸氧化產生己二酸(Musser,Adipic acid,Ullmann’s Encyclopedia of Industrial Chemistry,2000)。己內酰胺由環己酮經它的肟重排和隨后的酸重排制備(Fuchs,Kieczka and Moran,Caprolactam,Ullmann’s Encyclopedia of Industrial Chemistry,2000)。
工業上,通過將C6結構單元氫氰化成己二腈,然后氫化成HMD來制備六亞甲基二胺(HMD)(Herzog and Smiley,Hexamethylenediamine,Ullmann’s Encyclopedia of Industrial Chemistry,2012)。
鑒于對石油化學原料的依賴;生物技術提供了通過生物催化的替代方法。生物催化是使用生物催化劑如酶來進行有機化合物的生物化學轉化。
生物衍生的原料和石油化工原料兩者都是生物催化工藝的可行起始材料。
因而,針對此背景,明顯的是需要用于生產己二酸、己內酰胺、6-氨基己酸、6-羥基己酸、六亞甲基二胺和1,6-己二醇(下文稱為“C6結構單元”)的可持續的方法,其中所述方法是基于生物催化的(Jang等,Biotechnol.Bioeng.,2012,109(10),2437–2459)。
然而,無野生型的原核生物或真核生物天然地過度生產或分泌C6結構單元到細胞外環境。但是,己二酸和己內酰胺的代謝已有報道(Ramsay等,Appl.Environ.Microbiol.,1986,52(1),152–156;以及Kulkarni和Kanekar,Current Microbiology,1998,37,191–194)。
一些細菌和酵母將二羧酸,己二酸作為碳源通過β-氧化高效轉化為中心代謝產物。CoA己二酸至3-氧代己二酸的β-氧化促進經由與例如芳香族底物降解相關的鄰位裂解途徑的。由幾種細菌和真菌將3-氧代己二酰-CoA轉化為乙酰-CoA和琥珀酰-CoA的分解代謝已經全面表征(Harwood和Parales,Annual Review of Microbiology,1996,50,553–590)。己二酸和6-氨基己酸都是己內酰胺的分解代謝中的中間物,最終經3-氧代己二酰-CoA降解為中心代謝產物。
已經提出了由生物質-糖生產己二酸的潛在的代謝途徑:(1)以生物化學的方式通過鄰位裂解芳香族降解途徑從葡萄糖轉化成順式,順式-粘康酸,然后化學催化成己二酸;(2)通過琥珀酰-CoA和乙酰-CoA的縮合的可逆己二酸降解途徑和(3)組合β-氧化、脂肪酸合成酶和ω-氧化。然而,沒有報道過使用這些策略的信息(Jang等,Biotechnology&Bioengineering,2012,109(10),2437–2459)。
最優性原理指出,微生物調節它們的生物化學網絡來支持最大生物質(biomass)生長。超出在宿主生物體中表達異源性途徑的需要,將碳通量引導至充當碳源的C6結構單元而非生物質生長組分與最優性原理矛盾。例如,將1-丁醇途徑從梭菌屬物種轉移至其它生產菌株中,其與天然生產者(native producer)的生產性能相比,經常缺乏一個數量級(Shen等,Appl.Environ.Microbiol.,2011,77(9),2905–2915)。
在C6脂族主鏈上形成末端官能團如羧基、胺或者羥基之前,作為中心前體的六碳脂族主鏈的高效合成是合成C6結構單元中的關鍵考慮因素。
發明簡述
本申請至少部分地基于以下發現:可構建用于生產六碳鏈式脂族主鏈前體的生物化學途徑,在其中可形成一個或兩個官能團,即羧基、胺或者羥基,導致以下一種或者多種物質的合成:己二酸、6-氨基己酸、六亞甲基二胺、6-羥基己酸、己內酰胺或1,6-己二醇(下文稱為“C6結構單元”)。己二酸和己二酸鹽(酯),乙酸和乙酸鹽(酯),6-羥基己酸和6-羥基己酸鹽(酯),6-氨基己酸和6-氨基己酸鹽(酯)在本文中可替換使用,指代以其任意中性或離子化形式的化合物,包括其任意的鹽形式。本領域的技術人員應當理解具體的形式將依賴于pH。本文描述的這些途徑、代謝工程和培養策略依賴于脂肪酸合成酶或類似酶或和多肽,其具有接受C8或C12酰基-[acp]底物并氧化切割所述底物的C6和C7碳之間的C-C鍵的能力,以在當辛酰基-[acp]是底物時產生6-氧己酰-[acp]和乙醛或當十二酰基-[acp]是底物時產生6-氧己酰-[acp]和己醛。具有接受C8或C12酰基-[acp]底物并氧化切割所述底物的C6和C7碳之間的C-C鍵的能力的多肽(本文稱為庚二酰-[acp]合酶)可以與由來自枯草芽孢桿菌(bacillus subtilis)的bioI編碼的野生型庚二酰-[acp]合酶具有至少70%的序列同一性。野生型庚二酰-[acp]合酶通常氧化切割酰基-[acp]底物的C7和C8碳之間的C-C鍵。
面對最優性原理,令人驚訝地發現,可將適當的非天然途徑、原料、宿主微生物、對宿主的生物化學網絡的弱化策略和培養策略組合起來,從而高效地生產一種或多種C6結構單元。
在一些實施方案中,用于轉化為C6結構單元的C6脂族主鏈可以從脂肪酸合成中產生的十二酰基-[acp]或辛酰基-[acp]形成。見圖1。
在一些實施方案中,可以使用具有硫酯酶活性的多肽或具有醛脫氫酶活性的多肽酶促形成末端羧基。見圖2。
在一些實施方案中,可以使用具有ω-轉氨酶活性的多肽或具有二胺轉氨酶活性的多肽酶促形成末端胺基。見圖3,圖4,圖5和圖6。與己內酰胺相關的酰胺鍵是首先在直鏈碳鏈上具有末端羧基和末端胺基以形成鍵的結果。
在一些實施方案中,可以使用具有烷烴1-單加氧酶活性或具有醇脫氫酶活性的多肽酶促形成末端羥基。見圖7和圖8。
在一個方面,本申請特征在于在重組宿主中生物合成己二酰-[acp]的方法。所述方法包括使用具有庚二酰-[acp]合酶活性的多肽在宿主中將十二酰基-[acp]酶轉化為己二酰-[acp]和己酸,其中具有庚二酰-[acp]合酶活性的多肽接受十二酰基-[acp]作為底物并氧化切割底物的C6和C7碳之間的C-C鍵;或使用具有庚二酰-[acp]合酶活性的多肽在宿主中將辛酰基-[acp]酶促轉化成己二酰-[acp]和乙酸,其中具有庚二酰-[acp]合酶活性的多肽接受辛酰基-[acp]并氧化切割底物的C6和C7碳之間的C-C鍵。具有庚二酰-[acp]合酶活性的多肽可以與SEQ ID NO:23所示的氨基酸序列具有至少70%,至少80%或至少90%的序列同一性。該方法可以包括使用具有醛脫氫酶活性的多肽以將具有庚二酰-[acp]合酶活性的多肽的切割產物轉化為(i)己二酰-[acp]和己酸或(ii)己二酰-[acp]和乙酸酯。具有醛脫氫酶活性的多肽可以分類在EC 1.2.1.4或EC 1.2.1.3下。
本文公開的方法還可以包括使用至少一種具有選自醛脫氫酶,烷烴1-單加氧酶,硫酯酶,ω-轉氨酶,羧酸還原酶,N-酰基轉移酶,脫酰基酶,和醇脫氫酶的活性的多肽,酶促轉化己二酰-[acp]或己酸為選自下組的產物:己酸,己內酰胺,6-羥基己酸,6-氨基己酸,六亞甲基二胺和1,6-己二醇。
例如,該方法進一步可以包括使用具有硫酯酶活性的多肽將己二酰-[acp]酶促轉化為己二酸。具有硫酯酶活性的多肽可以與SEQ ID NO:1或SEQ ID NO:2中列出的氨基酸序列具有至少70%的序列同一性。
例如,本文所述的任何方法還可以包括使用至少一種具有選自(i)烷烴1-單加氧酶;(ii)醇脫氫酶;和(iii)醛脫氫酶的活性的多肽,將己酸酶促轉化為己二酸。具有醛脫氫酶活性的多肽可以分類在EC 1.2.1.3,EC 1.2.1.16,EC 1.2.1.20,EC 1.2.1.63或EC 1.2.1.79下和/或具有醇脫氫酶活性的多肽可以分類在EC 1.1.1.2或EC 1.1.1.258下。
例如,本文所述的任何方法還可以包括使用至少一種具有選自下組活性的多肽將己酸酶促轉化成6-氨基己酸:(i)烷烴1-單加氧酶;(ii)醇脫氫酶;和(iii)ω-轉氨酶。
例如,本文所述的任何方法還可以包括使用至少一種具有選自以下的活性的多肽將己二酸酶促轉化為6-氨基己酸:(i)羧酸還原酶;和(ii)ω-轉氨酶。
例如,本文所述的任何方法還可以包括使用至少一種具有選自以下的活性的多肽將己二酸或6-氨基己酸酶促轉化為六亞甲基二胺:(i)羧酸還原酶;和(ii)ω-轉氨酶。
例如,本文所述的任何方法還可以包括使用至少一種具有選自以下的活性的多肽將6-氨基己酸酶促轉化為六亞甲基二胺:(i)N-乙酰轉移酶;(ii)羧酸還原酶;(iii)ω-轉氨酶;和(iv)脫酰基酶。
例如,本文所述的任何方法還可以包括使用具有烷烴1-單加氧酶活性的多肽將己酸酶促轉化成6-羥基己酸。具有烷烴1-單加氧酶活性的多肽可以與SEQ ID NO:16-18中任一項所示的氨基酸序列具有至少70%的序列同一性。
例如,本文所述的任何方法還可以包括使用至少一種具有選自下組的活性的多肽將己二酸酶促轉化為6-羥基己酸:(i)羧酸還原酶;和(ii)醇脫氫酶。
例如,本文所述的任何方法還可以包括使用至少一種具有選自下組的活性的多肽將6-羥基己酸酶促轉化為六亞甲基二胺:(i)羧酸還原酶;(ii)ω-轉氨酶;和(iii)醇脫氫酶。
例如,本文所述的任何方法還可以包括使用至少一種具有選自下組的活性的多肽將6-羥基己酸酶促轉化為1,6-己二醇:(i)羧酸還原酶和(ii)醇脫氫酶。
例如,本文所述的任何方法還可以包括使用具有羧酸還原酶活性的多肽將己二酸酶促轉化為己二酸半醛。
例如,本文所述的任何方法還可以包括使用具有醇脫氫酶活性的多肽將6-羥基己酸酶促轉化為己二酸半醛。
例如,本文所述的任何方法還可包括使用至少一種具有選自(i)羧酸還原酶和(ii)轉氨酶的活性的多肽將己二酸半醛酶促轉化為六亞甲基二胺。
在任何所述方法中,具有羧酸還原酶活性的多肽可以與SEQ ID NO:3-7中中任一項所示的氨基酸序列具有至少70%的序列同一性,或者具有ω-轉氨酶活性的多肽可以具有與SEQ ID NO:8-13中中任一項所示的氨基酸序列具有至少70%的序列同一性。
在一些是實施方案中,生物原料可以是或源自單糖,二糖,木質纖維素、半纖維素、纖維素、木質素、乙酰丙酸和甲酸、甘油三酯、甘油、脂肪酸、農業廢物、濃縮酒糟(condensed distillers'solubles)或城市廢物。
在一些實施方案中,非生物原料可以是或可以源自天然氣、合成氣、CO2/H2、甲醇、乙醇、苯甲酸鹽(酯)、非揮發性殘留物(NVR)或來自環己烷氧化過程的堿洗液(caustic wash)廢物流、或對苯二甲酸/異酞酸混合物廢物流。
本文所述途徑的反應可以在一種或多種細胞(例如宿主細胞)株中進行,所述細胞株(a)天然表達一種或多種相關酶,(b)經遺傳工程化而表達一種或多種相關酶,或(c)天然表達一種或多種相關酶并經遺傳工程化而表達一種或多種相關酶。或者,可以從以上任意類型的宿主細胞中提取相關酶,并以純化或半純化形式使用。提取的酶可選地固定在固體基質上,例如適合的反應容器的底和/或壁。此外,這些提取物包括可以作為相關酶來源使用的裂解物(如細胞裂解物)。在本申請提供的方法中,所有步驟可以在細胞(例如宿主細胞)中進行,所有步驟可以使用提取的酶進行,或一些步驟可以在細胞中進行,而其它步驟可以使用提取的酶進行。
本文所述的多種酶催化可逆反應,并且感興趣的反應可以是所述反應的反向。圖1-8所示的示意性途徑闡明了對于每種中間物感興趣的反應。
在一些實施方案中,經由在選擇性環境中的連續培養改善所述宿主對高濃度一種或多種C6結構單元的耐受性。
在一些實施方案中,弱化或增強所述宿主的內源性生物化學網絡,來(1)保證乙酰-CoA和丙二酰-CoA的細胞內可用度,(2)創造NADH不平衡,其僅可通過脂肪酸合成和一種或多種C6結構單元的形成平衡,(3)阻止通向并包括C6結構單元的中心代謝物和中心前體的降解和(4)保證從細胞的高效流出。
在一些實施方案中,培養策略需要達到需氧或微氧的培養條件。
在一些實施方案中,培養策略需要經由氮,磷酸鹽或氧限制的營養限制。
在一些實施方案中,培養策略需要防止脂肪酸摻入脂質體或其它碳儲存單元中。
在一些實施方案中,使用例如發酵策略,通過單一類型的微生物,例如含有一種或多種外源核酸的重組宿主,產生一種或多種C6結構單元。
本申請的特征還在于包含至少一種編碼具有庚二酰-[acp]合酶活性的多肽的外源核酸的重組宿主,所述宿主產生:(a)己二酰-[acp]和己酸,其中所述具有庚二酰-[acp]合酶活性的多肽接受十二酰基-[acp]作為底物并氧化切割底物的C6和C7碳之間的CC鍵;或[b]己二酰-[acp],其中具有庚二酰-[acp]合酶活性的多肽接受辛酰基-[acp]作為底物,并氧化切割底物的C6和C7碳之間的C-C鍵。具有庚二酰-[acp]合酶活性的多肽可以與SEQ ID NO:23所示的氨基酸序列具有至少70%,至少80%或至少90%的序列同一性。
宿主還可以包括具有醛脫氫酶活性的外源多肽。
宿主還可以包括一種或多種具有選自烷烴1-單加氧酶,硫酯酶,醇脫氫酶和醛脫氫酶的活性的外源多肽,所述宿主產生己二酸。
宿主還可以包括具有羧酸還原酶活性的外源多肽和具有ω-轉氨酶活性的外源多肽,所述宿主產生6-氨基己酸。
產生6-氨基己酸的重組宿主還可以包括具有水解酶活性的外源多肽,所述宿主產生己內酰胺。
宿主還可以包括一種或多種具有選自烷烴1-單加氧酶,硫酯酶,羧酸還原酶和醇脫氫酶的活性的外源多肽,所述宿主產生6-羥基己酸。
宿主還可以包括至少一種具有選自烷烴1-單加氧酶,硫酯酶,羧酸還原酶和醇脫氫酶的活性的外源多肽,所述宿主產生己二酸半醛。宿主還可以包括至少一種具有ω-轉氨酶活性的外源多肽,所述宿主產生六亞甲基二胺。
宿主還可以包括至少一種具有選自N-乙酰轉移酶和脫酰基酶的活性的外源多肽,所述宿主產生六亞甲基二胺。
宿主可以包括(i)至少一種具有烷烴1-單加氧酶活性的外源多肽,至少一種具有醇脫氫酶活性的外源多肽,至少一種具有ω-轉氨酶活性的外源多肽和至少一種具有羧酸還原酶活性的多肽或(ii)至少一種具有硫酯酶活性的外源多肽,至少一種具有羧酸還原酶活性的多肽和至少一種具有ω-轉氨酶活性的外源多肽,宿主產生六亞甲基二胺。
宿主可以包括(i)至少一種具有羧酸還原酶活性的外源多肽,至少一種具有醇脫氫酶活性的外源多肽和至少一種具有烷烴1-單加氧酶活性的多肽,或(ii)至少一種具有羧酸還原酶活性的多肽,至少一種具有醇脫氫酶活性的外源多肽和至少一種具有硫酯酶活性的外源多肽,所述宿主產生1,6己二醇。
在任何方法或宿主中,具有硫酯酶活性的多肽可以與SEQ ID NO:1或SEQ ID NO:2所示的氨基酸序列具有至少70%的序列同一性。
在任何方法或宿主中,具有烷烴1-單加氧酶活性的多肽可以與SEQ ID NO:16-18中中任一項所示的氨基酸序列具有至少70%的序列同一性。
在任何方法或宿主中,具有羧酸還原酶活性的多肽與SEQ ID NO:3-7中中任一項所示的氨基酸序列具有至少70%的序列同一性。
在任何方法或宿主中,具有ω-轉氨酶活性的多肽可以與SEQ ID NO:8-13中中任一項所示的氨基酸序列具有至少70%的序列同一性。
任何重組宿主可以是原核生物,如來自選自下組的屬的原核生物:埃希氏菌屬(Escherichia);梭菌屬(Clostridia);棒狀桿菌屬(Corynebacteria);貪銅菌屬(Cupriavidus);假單胞菌屬(Pseudomonas);代爾夫特菌屬(Delftia);芽孢桿菌屬(Bacilluss);乳桿菌屬(Lactobacillus);乳球菌屬(Lactococcus);和紅球菌屬(Rhodococcus)。例如,所述原核生物可以選自下組:大腸桿菌(Escherichia coli)、楊氏梭菌(Clostridium ljungdahlii)、自產乙醇梭菌(Clostridium autoethanogenum)、克魯佛梭菌(Clostridium kluyveri)、谷氨酸棒狀桿菌(Corynebacterium glutamicum)、鉤蟲貪銅菌(Cupriavidus necator)、耐金屬貪銅菌(Cupriavidus metallidurans)、熒光假單胞菌(Pseudomonas fluorescens)、惡臭假單胞菌(Pseudomonas putida)、食油假單胞菌(Pseudomonas oleavorans)、食酸代爾夫特菌(Delftia acidovorans)、枯草芽胞桿菌(Bacillus subtillis)、德氏乳桿菌(Lactobacillus delbrueckii)、乳酸乳球菌(Lactococcus lactis)、和馬紅球菌(Rhodococcus equi)。此類原核生物也可以是構建能夠生成C6結構單元的本文中描述的重組宿主細胞的基因來源。
任何宿主可以是真核生物,例如來自選自下組的屬的真核生物:曲霉屬(Aspergillus)、酵母屬(Saccharomyces)、畢赤氏酵屬(Pichia)、耶羅維亞酵母屬(Yarrowia)、伊薩酵母屬(Issatchenkia)、德巴利酵母屬(Debaryomyces)、Arxula、和克魯維酵母菌屬(Kluyveromyces)。例如,所述真核生物可以選自下組:黑曲霉(Aspergillus niger)、釀酒酵母(Saccharomyces cerevisiae)、巴斯德畢赤酵母(Pichia pastoris)、解脂耶羅維亞酵母(Yarrowia lipolytica)、東方伊薩酵母(Issathenkia orientalis)、漢遜德巴利酵母(Debaryomyces hansenii)、Arxula adenoinivorans、和乳酸克魯維酵母菌(Kluyveromyces lactis)。此類真核生物也可以是構建能夠生成C6結構單元的本文中描述的重組宿主細胞的基因來源。
本文所述任何宿主還可以包含一種或多種以下酶的弱化:聚羥基烷酸酯(polyhydroxyalkanoate)合酶,形成乙酸的磷酸乙酸轉移酶,乙酸激酶,乳酸脫氫酶,形成乙醇的醇脫氫酶,丙糖磷酸異構酶,NADH消耗型轉氫酶,NADH特異性谷氨酸脫氫酶,和NADH/NADPH利用型谷氨酸脫氫酶。
本文所述任何宿主還可以過表達編碼以下的一種或多種基因:乙酰-CoA合成酶,6-磷酸葡萄糖酸脫氫酶;轉酮醇酶;吡啶(puridine)核苷酸轉氫酶;甘油醛-3P-脫氫酶;蘋果酸酶;葡萄糖-6-磷酸脫氫酶;葡萄糖脫氫酶;果糖1,6二磷酸酶;L-丙氨酸脫氫酶;L-谷氨酸脫氫酶;甲酸脫氫酶;L-谷氨酰胺合成酶;二胺轉運體;二羧酸轉運體;和/或多藥物轉運體。
此外,本文描述了包含具有庚二酰-[acp]合酶活性的多肽和十二烷酰基-[acp]的生物化學網絡,其中具有庚二酰-[acp]合酶活性的多肽將十二酰基-[acp]酶促轉化為己醛或6-氧代己酰-[acp]。生物化學網絡可以進一步包括具有醛脫氫酶活性的多肽,其中具有醛脫氫酶活性的多肽進一步將己醛和6-氧代己酰-[acp]分別轉化成己酸和己二酰-[acp]。
此外,本文描述了包含具有庚二酰基-[acp]合酶活性的多肽和辛酰基-[acp]的生物化學網絡,其中具有庚二酰基-[acp]合酶活性的多肽將辛酰基-[acp]酶促轉化為乙醛和6-氧代己酰基-[acp]。生物化學網絡可以進一步包括具有醛脫氫酶活性的多肽,其中具有醛脫氫酶活性的多肽進一步分別將乙醛和6-氧代己酰基-[acp]轉化為乙酸和己二酰基-[acp]。
任何生物化學網絡可進一步包括具有醛脫氫酶活性的多肽,具有單加氧化酶活性的多肽,具有硫酯酶活性的多肽,具有ω-轉氨酶活性的多肽,具有羧酸還原酶活性的多肽,具有二胺轉氨酶活性的多肽,具有N-乙酰轉移酶活性的多肽,具有賴氨酸N-乙酰轉移酶活性,具有脫酰酶活性的多肽或具有醇脫氫酶多肽活性的多肽,其中所述具有醛脫氫酶活性的多肽,所述具有單加氧酶活性的多肽,所述具有硫酯酶活性德多肽,所述具有ω-轉氨酶活性的多肽,所述具有羧酸還原酶活性的多肽,具有二胺轉氨酶活性的多肽,具有N-乙酰轉移酶活性的多肽,具有賴氨酸N-乙酰轉移酶活性的多肽,具有脫酰基酶活性的多肽或具有醇脫氫酶活性的多肽將己二酰-[acp]和/或己酸酶轉化為己二酸,6-氨基己酸,己內酰胺,六亞甲基二胺,6-羥基己酸和1,6-己二醇中的至少一種。
此外,本文描述了使用具有庚二酰基-[acp]合酶的多肽獲得己醛和6-氧己酰基-[acp]的手段(means)。該手段還可以包括將己醛和6-氧己酰基-[acp]分別轉化為己酸和己二酰-[acp]的手段。
此外,本文描述了使用具有庚二酰-[acp]合酶活性的多肽獲得乙醛和6-氧己酰基-[acp]的手段。該手段還可以包括將乙醛和6-氧代己酰基-[acp]分別轉化成乙酸和己二酰基-[acp]的手段。
所述方法可以包括使用具有醛脫氫酶活性的多肽。該方法還可以包括將己二酰-[acp]或己酸轉化成己二酸,6-氨基己酸,己內酰胺,六亞甲基二胺,6-羥基己酸和1,6-己二醇中的至少一種的手段。所述手段可以包括具有醛脫氫酶活性的多肽,具有單硫氧化酶活性的多肽,具有硫酯酶活性的多肽,具有ω-轉氨酶活性的多肽,具有羧酸還原酶活性的多肽,具有二胺轉氨酶活性的多肽,具有N-乙酰轉移酶活性的多肽,具有賴氨酸N-乙酰轉移酶活性的多肽,具有脫酰基酶活性的多肽或具有醇脫氫酶活性的多肽
此外,本文描述的是使用具有庚二酰基-[acp]合酶活性的多肽獲得己二酰-[acp]或己酸的步驟。
另一方面,本申請的特征在于包含己醛和6-氧代己酰基-[acp]和具有庚二酰基-[acp]合酶活性的多肽的組合物。另一方面,本申請的特征在于包含乙醛和6-氧代己酰基-[acp]和具有庚二酰基-[acp]合酶活性的多肽的組合物。
組合物可以是無細胞的或細胞的。所述組合物還可包括具有醛脫氫酶活性的多肽,具有單加氧酶活性的多肽,具有硫酯酶活性的多肽,具有ω-轉氨酶活性的多肽,具有羧酸還原酶活性的多肽,具有二胺轉氨酶活性的多肽,具有N-乙酰轉移酶活性的多肽,具有賴氨酸N-乙酰轉移酶活性的多肽,具有脫酰基酶活性的多肽或具有醇脫氫酶活性的多肽和己二酸,6-氨基己酸,己內酰胺,六亞甲基二胺,6-羥基己酸和1,6-己二醇中的至少一種。
本申請還描述了用于生產生物衍生的己二酰-[acp],己酸,己二酸,己內酰胺,6-羥基己酸,6-氨基己酸,六亞甲基二胺或1,6-己二醇的方法,其包括在條件下培養或生長本文所述的任何重組宿主足夠的時間段以產生生物衍生的己二酰-[acp],己酸,己二酸,己內酰胺,6-羥基己酸,6-氨基己酸,六亞甲基二胺或1,6-己二醇。
在另一方面,本文特征為包含生物衍生的己二酰-[acp],己二酸,己內酰胺,6-羥基己酸,6-氨基己酸,六亞甲基二胺或1,6-己二醇的培養基,其中生物衍生的己二酰-[acp],己二酸,己內酰胺,6-羥基己酸,6-氨基己酸,六亞甲基二胺或1,6-己二醇具有反映大氣二氧化碳攝取來源的碳-12,碳-13和碳-14同位素比率。可以從重組宿主分離培養基。
本申請的特征還在于生物衍生的己二酰-[acp],己二酸,己內酰胺,6-羥基己酸,6-氨基己酸,六亞甲基二胺或1,6-己二醇,其具有反映大氣二氧化碳攝取源的碳-12,碳-13和碳-14同位素比率,優選通過生長本文所述的重組宿主生產。生物衍生的己二酰-[acp],己二酸,己內酰胺,6-羥基己酸,6-氨基己酸,六亞甲基二胺或1,6-己二醇可具有至少80%,至少85%,至少90%,至少95%或至少98%的Fm值。
在另一方面,本申請特征在于組合物,其包含生物衍生的己二酰-[acp],己二酸,己內酰胺,6-羥基己酸,6-氨基己酸,六亞甲基二胺或1,6-己二醇和不同于生物來源的己二酰-[acp],己二酸,己內酰胺,6-羥基己酸,6-氨基己酸,六亞甲基二胺或1,6-己二醇的化合物。不同于生物衍生的己二酰-[acp],己二酸,己內酰胺,6-羥基己酸,6-氨基己酸,六亞甲基二胺或1,6-己二醇的化合物可以是痕量的本文所述重組宿主的細胞部分。
本申請特征還在于包含本文所述的生物衍生的己二酸,己內酰胺,6-羥基己酸,6-氨基己酸,六亞甲基二胺或1,6-己二醇的生物基聚合物(biobased polymer)以及通過模制(molding)生物基聚合物獲得的模制產品。
本申請特征還在于了包含本文所述的生物衍生的己二酸,己內酰胺,6-羥基己酸,6-氨基己酸,六亞甲基二胺或1,6-己二醇的生物基樹脂以及通過模制生物基樹脂獲得的模制產品。
在另一方面,本申請的特征在于生產生物基聚合物的方法,其包括使生物衍生的己二酸,己內酰胺,6-羥基己酸,6-氨基己酸,六亞甲基二胺或1,6-己二醇與其自身或另一種化合物在聚合物生成反應中起化學反應。
在另一方面,該文獻的特征在于生產生物基樹脂的方法,其包括使生物衍生的己二酸,己內酰胺,6-羥基己酸,6-氨基己酸,六亞甲基二胺或1,6-己二醇與其自身或另一種化合物在樹脂生成反應中起化學反應。
除非另有定義,否則本文使用的所有技術和科學術語具有與本發明所屬領域的普通技術人員通常理解的相同的含義。盡管與本文所述的方法和材料類似或等同的方法和材料可用于實施本發明,但下文描述了合適的方法和材料。本文提及的所有出版物,專利申請,專利和其它參考文獻通過引用以其整體并入。在沖突的情況下,以本說明書包括定義為準。此外,材料,方法和實施例僅僅是說明性的,而不是限制性的。
在附圖和下面的描述中闡述了本發明的一個或多個實施例的細節。本發明的其它特征,目的和優點將從說明書和附圖以及權利要求書中顯而易見。根據專利法中的標準實踐,權利要求中的詞語“包括”可以被“基本上由...組成”或由“由...組成”替代。
附圖簡述
圖1是使用十二酰基-[acp]作為示例性長鏈脂肪酸中心代謝物導致己二酰-[acp]和己酸的示例性生物化學途徑的示意圖,以及使用辛酰基-[acp]作為示例性長鏈脂肪酸中心代謝物導致己二酰-[acp]和乙酸的示例性生物化學途徑的示意圖。
圖2是使用己二酰-[acp]或己酸作為中心前體導致己二酸的示例性生物化學途徑的示意圖。
圖3是使用己二酰-[acp]或己酸作為中心前體導致6-氨基己酸的示例性生物化學途徑的示意圖。圖3還包含從6-氨基己酸產生己內酰胺的示意圖。
圖4是使用6-氨基己酸或己二酸半醛(也稱為6-氧代己酸)作為中心前體導致六亞甲基二胺的示例性生物化學途徑的示意圖。
圖5是使用6-氨基己酸作為中心前體導致六亞甲基二胺的示例性生物化學途徑的示意圖。
圖6是使用6-羥基己酸作為中心前體的導致六亞甲基二胺的示例性生物化學途徑的示意圖。
圖7是使用己二酰-[acp]或己酸作為中心前體導致6-羥基己酸的示例性生物化學途徑的示意圖。
圖8是使用6-羥基己酸作為中心前體導致1,6-己二醇的示例性生物化學途徑的示意圖。
圖9是總結20分鐘后在340nm處的吸光度變化的柱狀圖,其是相對于僅酶對照(無底物)的NADPH的消耗和羧酸還原酶的活性的量度。
圖10是20分鐘后340nm處吸光度變化的柱狀圖,其是相對于空載體對照的NADPH的消耗和羧酸鹽還原酶將己二酸轉化為己二酸半醛的活性的量度。
圖11是20分鐘后340nm處吸光度變化的柱狀圖,其是相對于空載體對照的NADPH的消耗和將6-羥基己酸轉化為6-羥基己醛的羧酸還原酶的活性的量度。
圖12是20分鐘后在340nm處的吸光度變化的柱狀圖,其是相對于空載體對照的NADPH的消耗和將N6-乙酰基-6-氨基己酸轉化為N6-乙酰基-6-氨基己醛的羧酸還原酶的活性的量度。
圖13是20分鐘后340nm處吸光度變化的柱狀圖,其是相對于空載體對照的NADPH的消耗和將己二酸半醛轉化為己二醛的羧酸還原酶的活性的量度。
圖14是總結了4小時后丙酮酸轉化為L-丙氨酸的轉化百分比(mol/mol)的柱狀圖,作為僅酶對照(無底物)的ω-轉氨酶活性的量度。
圖15是24小時后丙酮酸鹽轉化為L-丙氨酸后的轉化百分比(mol/mol)的柱狀圖,作為相對于空載體對照的將6-氨基己酸轉化為己二酸半醛的ω-轉氨酶活性的量度。
圖16是4小時后L-丙氨酸轉化為丙酮酸的轉化百分比(mol/mol)的柱狀圖,作為相對于空載體對照,將己二酸半醛轉化為6-氨基己酸的ω-轉氨酶活性的量度。
圖17是4小時后丙酮酸轉化為L-丙氨酸的轉化百分比(mol/mol)的柱狀圖,作為相對于空載體對照,將六亞甲基二胺轉化為6-氨基己醛的ω-轉氨酶活性的量度。
圖18是4小時后丙酮酸轉化為L-丙氨酸的轉化百分比(mol/mol)的柱狀圖,作為相對于空載體對照,將N6-乙酰基-1,6-二氨基己烷轉化為N6-乙酰基-6-氨基己醛的ω-轉氨酶活性的量度。
圖19是4小時后丙酮酸轉化為L-丙氨酸的轉化百分比(mol/mol)的柱狀圖,作為相對于空載體對照,將6-氨基己醇轉化為6-氧代己醇的ω-轉氨酶活性的量度。
圖20是如通過LC-MS測定的6-羥基己酸的峰面積變化的柱狀圖,作為相對于空載體對照,將己酸轉化為6-羥基己酸的單加氧酶活性的量度。
圖21含有短乳桿菌硫酯酶(參見GenBank登錄號ABJ63754.1,SEQ ID NO:1),植物乳桿菌硫酯酶(參見GenBank登錄號CCC78182.1,SEQ ID NO:2),海分枝桿菌羧酸還原酶(參見Genbank登錄號ACC40567.1,SEQ ID NO:3),恥垢分枝桿菌羧酸還原酶(參見Genbank登錄號ABK71854.1,SEQ ID NO:4),Segniliparus rugosus羧酸還原酶(參見Genbank登錄號EFV11917.1,SEQ ID NO:5),馬賽分枝桿菌(Mycobacterium massiliense)羧酸還原酶(參見Genbank登錄號EIV11143.1,SEQ ID NO:6),Segniliparus rotundus羧酸還原酶(參見Genbank登錄號ADG98140.1,SEQ ID NO:7),紫色色桿菌ω-轉氨酶(參見Genbank登錄號AAQ59697.1,SEQ ID NO:8),銅綠假單胞菌ω-轉氨酶(參見Genbank登錄號AAG08191.1,SEQ ID NO:9),丁香假單胞菌ω-轉移酶(參見Genbank登錄號AAY39893.1,SEQ ID NO:10),球形紅細菌ω-轉移酶(參見Genbank登錄號ABA81135.1,SEQ ID NO:11),大腸桿菌ω-轉移酶(參見Genbank登錄號AAA57874.1,SEQ ID NO:12),河流孤菌ω-轉移酶(參見Genbank登錄號AEA39183.1,SEQ ID NO:13),枯草芽孢桿菌磷酸泛酰巰基乙胺基轉移酶(參見Genbank登錄號CAA44858.1,SEQ ID NO:14),諾卡氏菌屬物種NRRL 5646磷酸泛酰巰基乙胺基轉移酶(參見Genbank登錄號ABI83656.1,SEQ ID NO:15),單胞菌屬物種JS666單加氧酶(參見Genbank登錄號ABE47160.1,SEQ ID NO:16),分枝桿菌屬物種HXN-1500單加氧酶(參見Genbank登錄號CAH04396.1,SEQ ID NO:17),南非分支桿菌(Mycobacterium austroafricanum)單加氧酶(參見Genbank登錄號ACJ06772.1,SEQ ID NO:18),單胞菌屬物種JS666氧化還原酶(參見Genbank登錄號ABE47159.1,SEQ ID NO:19),分枝桿菌屬物種HXN-1500氧化還原酶(參見Genbank登錄號CAH04397.1,SEQ ID NO:20),單胞菌屬物種JS666鐵氧還原蛋白(參見Genbank登錄號ABE47158.1,SEQ ID NO:21),分枝桿菌屬物種HXN-1500鐵氧還蛋白(參見Genbank登錄號CAH04398.1,SEQ ID NO:22)和由bioI編碼的枯草芽孢桿菌庚二酰-[acp]合酶(參見Genbank登錄號AAB17462.1,SEQ ID NO:23)。
發明詳述
本文描述了酶、非天然途徑、培養策略、原料、宿主微生物和對宿主的生物化學網絡的弱化,從中心代謝產物生成六碳鏈式脂族主鏈,在所述六碳鏈式脂族主鏈中可形成一個或兩個末端官能團,導致己二酸、6-氨基己酸、6-羥基乙酸、六亞甲基二胺、己內酰胺或1,6-己二醇(本文稱為“C6結構單元”)的合成。如本文所使用的,術語“中心前體”用于表示本文所示的導致C6結構單元合成的任意代謝途徑中的任意代謝產物。術語“中心代謝產物”用于本文表示在所有微生物中生產以支持生長的代謝產物。
本文所述的宿主微生物可以包括能夠操作,使得能生產一種或多種C6結構單元的內源性途徑。在內源性途徑中,所述宿主微生物天然表達催化所述途徑中反應的所有酶。含有工程化途徑的宿主微生物不天然表達催化所述途徑中反應的所有酶,但已經過工程化使得所述途徑中的所有的酶在所述宿主中表達。
如本文中提及核酸(或蛋白質)和宿主使用的,術語“外源”指不像其在自然界中被發現一樣存在于特定類型細胞中(并且不能從特定類型細胞獲得)的核酸或由所述核酸編碼的蛋白質。如此,非天然存在的核酸一旦在宿主中即視為對于宿主而言外源的。重要的是注意非天然存在的核酸可含有在自然界中發現的核酸序列的核酸亞序列或片段,只要該核酸作為整體不存在于自然界中。例如,表達載體內含有基因組DNA序列的核酸分子是非天然存在的核酸,如此一旦導入宿主中對于宿主細胞而言是外源的,因為該核酸分子作為整體(基因組DNA加載體DNA)不存在于自然界中。如此,作為整體不存在于自然界中的任何載體、自主復制的質粒或病毒(例如逆轉錄病毒、腺病毒、或皰疹病毒)視為非天然存在的核酸。由此得出結論通過PCR或限制內切性核酸酶處理產生的基因組DNA片段以及cDNA也視為非天然存在的核酸,因為它們作為未見于自然界的分開的分子存在。由此還得出結論任何以未見于自然界中的排布含有啟動子序列和多肽編碼序列(例如cDNA或基因組DNA)的任何核酸也是非天然存在的核酸。天然存在的核酸可以是對于特定宿主微生物而言外源的。例如,從酵母x的細胞分離的完整染色體一旦將該染色體導入酵母y的細胞中就酵母y細胞而言是外源核酸。
比較而言,如本文中提及核酸(例如基因)(或蛋白質)和宿主使用的,術語“內源的”指就像其在自然界中被發現一樣的確存在于特定宿主中(并且可以從特定宿主獲得)的核酸(或蛋白質)。此外,“內源表達”核酸(或蛋白質)的細胞就像其在自然界中被發現時相同特定類型的宿主那樣表達所述核酸(或蛋白質)。此外,“內源生成”核酸、蛋白質、或其它化合物的宿主就像其在自然界中被發現時相同特定類型的宿主那樣生成所述核酸、蛋白質、或化合物。
例如,根據宿主和由宿主產生的化合物,除了具有庚二酰-[acp]合酶活性的多肽之外,還可以在宿主中表達一種或多種以下多肽:具有醛脫氫酶活性的多肽,具有烷烴1-單加氧酶活性的多肽,具有硫酯酶活性的多肽,具有ω-轉氨酶活性的多肽,具有羧酸還原酶活性的多肽,具有水解酶活性的多肽,具有二胺轉氨酶活性的多肽,具有N-乙酰轉移酶活性的多肽,具有賴氨酸N-乙酰轉移酶活性的多肽,具有脫酰基酶活性的多肽,或具有醇脫氫酶活性的多肽。在表達具有羧酸還原酶活性的多肽的重組宿主中,也可以具有磷酸泛酰巰基乙胺基轉移酶活性的多肽,因為它增強羧酸還原酶活性的活性。在表達具有單加氧酶活性的多肽的重組宿主中,還可以表達電子轉移鏈蛋白,如具有氧化還原酶活性的多肽或鐵氧還蛋白多肽。
例如,重組宿主可以包括具有庚二酰-[acp]合酶活性的外源多肽并產生6-氧代己酰-[acp]和己醛或乙醛,這取決于十二酰基-[acp]或辛酰基-[acp]是否是具有庚二酰-[acp]合酶活性的多肽的底物。
例如,重組宿主可以包括具有庚二酰-[acp]合酶活性的外源多肽和具有醛脫氫酶活性的外源多肽,并且產生(i)己二酰-[acp]和己酸或(ii)己二酰-[acp]和乙酸,這取決于十二酰基-[acp]或辛酰基-[acp]是否是具有庚二酰-[acp]合酶活性的多肽的底物。在其中十二酰基-[acp]是重組宿主中具有庚二酰-[acp]合酶活性的多肽的底物的實施方案中,可以產生己二酰基-[acp]和己酸。在其中辛酰基-[acp]是重組宿主中具有庚二酰-[acp]合酶活性的多肽的底物的實施方案中,可以產生己二酰-[acp]和乙酸。
例如,重組宿主可以包括具有庚二酰-[acp]合酶活性的外源多肽,具有醛脫氫酶活性的外源多肽和具有硫酯酶活性的外源多肽,并產生己二酸。參見例如圖2。
例如,重組宿主可以包括具有庚二酰-[acp]合酶活性的外源多肽,具有醛脫氫酶活性的外源多肽,以及至少一種選自下組的多肽:(i)具有烷烴1-單加氧酶活性的外源多肽,(ii)具有醇脫氫酶活性的外源多肽和(iii)具有醛脫氫酶活性的多肽并產生己二酸。在一些實施方案中,宿主包括具有不同醛脫氫酶活性的兩種或更多種外源多肽。參見例如圖2。
例如,重組宿主可以包括具有庚二酰-[acp]合酶活性的外源多肽,具有醛脫氫酶活性的外源多肽,以及至少一種選自下組的多肽(i)具有烷烴1-單加氧酶活性的外源多肽,(ii)具有醇脫氫酶活性的外源多肽,(iii)具有硫酯酶活性的外源多肽,并產生己二酸。在一些實施方案中,宿主包括具有不同醛脫氫酶活性的兩種或更多種外源多肽。參見例如圖2。
例如,重組宿主可以包括具有庚二酰-[acp]合酶活性的外源多肽,具有醛脫氫酶活性的外源多肽,具有烷烴1-單加氧酶活性的外源多肽和具有醇脫氫酶活性的外源多肽,并產生己二酸半醛。參見例如圖2。
例如,重組體可以包括具有庚二酰-[acp]合酶活性的外源多肽,具有醛脫氫酶活性的外源多肽,具有硫酯酶活性的外源多肽和具有羧酸還原酶活性的外源多肽,并產生己二酸半醛。參見例如圖3。
例如,重組宿主可以包括具有庚二酰-[acp]合酶活性的外源多肽,具有醛脫氫酶活性的外源多肽,以及至少一種選自下組的外源多肽:具有硫酯酶活性的外源多肽,具有羧酸還原酶活性的外源多肽,和具有ω-轉氨酶活性的多肽,并產生6-氨基己酸。例如,重組宿主可以包括具有庚二酰-[acp]合酶活性的外源多肽,具有醛脫氫酶活性的外源多肽,具有硫酯酶活性的外源多肽,具有羧酸還原酶活性的外源多肽和具有ω-轉氨酶活性的外源多肽,并產生6-氨基己酸。參見圖3。
例如,重組宿主可以包括具有庚二酰-[acp]合酶活性的外源多肽,具有醛脫氫酶活性的外源多肽和至少一種選自下組的外源多肽:具有烷烴1-單加氧酶活性的多肽,具有醇脫氫酶活性的多肽,具有ω-轉氨酶活性的多肽,并產生6-氨基己酸。參見圖3。
例如,重組宿主可以包括具有庚二酰-[acp]合酶活性的外源多肽,具有醛脫氫酶活性的外源多肽,以及至少一種選自下組的外源多肽:具有烷烴1-單加氧酶活性的多肽,具有醇脫氫酶活性的多肽,具有ω-轉氨酶活性的多肽,具有硫酯酶活性的外源多肽和具有羧酸還原酶活性的外源多肽,并產生6-氨基己酸。例如,重組宿主可以包括具有庚二酰-[acp]合酶活性的外源多肽,具有醛脫氫酶活性的外源多肽,具有烷烴1-單加氧酶活性的外源多肽,具有醇脫氫酶活性的外源多肽,具有硫酯酶活性的外源多肽,具有羧酸還原酶活性的多肽,以及具有ω-轉氨酶活性的外源多肽,并產生6-氨基己酸。參見圖3。
例如,產生6-氨基己酸的重組宿主還可以包括具有酰胺水解酶(amidohydrolase)活性的外源多肽并產生己內酰胺。參見圖3。
例如,產生6-氨基己酸的重組宿主可包括具有羧酸還原酶活性的外源多肽和具有ω轉氨酶活性的外源多肽,并產生六亞甲基二胺。具有羧酸還原酶活性的外源多肽可以是具有羧酸還原酶活性的第二外源多肽。第二外源羧酸還原酶可以與第一外源羧酸還原酶相同或不同。具有ω轉氨酶活性的外源多肽可以是具有ω轉氨酶活性的第二外源多肽。具有ω轉氨酶活性的第二外源多肽可以與具有ω轉氨酶活性的第一外源多肽相同或不同。參見圖4。
例如,產生己二酸半醛的重組宿主還可以包括具有羧酸還原酶活性的外源多肽和具有轉氨酶活性的外源多肽,并產生六亞甲基二胺。具有羧酸還原酶活性的外源多肽可以是具有羧酸還原酶活性的第二外源多肽。第二外源羧酸還原酶可以與具有羧酸還原酶活性的第一外源多肽相同或不同。具有ω轉氨酶活性的外源多肽可以是具有ω轉氨酶活性的第二外源多肽。第二外源ω轉氨酶可以與第一外源ω轉氨酶相同或不同。參見圖4。
例如,產生6-氨基己酸的重組宿主還可以包括具有N-乙酰轉移酶活性的外源多肽,具有羧酸還原酶活性的外源多肽,具有轉氨酶活性的外源多肽,具有脫酰基酶活性的外源多肽,并產生六亞甲基二胺。具有羧酸還原酶活性的多肽可以是具有羧酸還原酶活性的第二外源多肽。第二外源羧酸還原酶可以與具有羧酸還原酶活性的第一外源多肽相同或不同。具有ω轉氨酶活性的外源多肽可以是具有ω轉氨酶活性的第二外源多肽。第二外源ω轉氨酶可以與第一外源ω轉氨酶相同或不同。參見圖5。
例如,重組宿主可以包括具有庚二酰-[acp]合酶活性的外源多肽,具有醛脫氫酶活性的外源多肽和至少一種選自下組的外源多肽:具有烷烴1-單加氧酶活性的外源多肽,具有硫酯酶活性的外源多肽,具有羧酸還原酶活性的外源多肽和具有醇脫氫酶活性的外源多肽,并產生6-羥基己酸。參見圖7。
例如,產生己酸的重組宿主還可以包括具有烷烴1-單加氧酶活性的外源多肽,并產生6-羥基己酸。參見圖7。
例如,產生己二酰-[acp]的重組宿主還可以包括具有硫酯酶活性的外源多肽,具有羧酸還原酶活性的外源多肽和具有醇脫氫酶活性的外源多肽,并產生6-羥基己酸。參見圖7。
例如,產生6-羥基己酸的重組宿主可以進一步包括至少一種選自下組的外源酶:具有羧酸還原酶活性的外源多肽,具有轉氨酶活性的外源多肽和具有醛脫氫酶活性的外源多肽,并產生六亞甲基二胺。具有羧酸還原酶活性的外源多肽可以是具有羧酸還原酶活性的第二外源多肽。第二外源羧酸還原酶可以與具有羧酸還原酶活性的第一外源多肽相同或不同。具有醇脫氫酶活性的外源多肽可以是具有醇脫氫酶活性的第二外源多肽。第二外源醇脫氫酶可以與第一外源醇脫氫酶相同或不同。參見圖6。
在一些實施方案中,產生6-羥基己酸的重組宿主可以進一步包括具有羧酸還原酶活性的外源多肽和具有醇脫氫酶活性的外源多肽,并產生1,6己二醇。具有羧酸還原酶活性的外源多肽可以是具有羧酸還原酶活性的第二外源多肽。第二外源羧酸還原酶可以與具有羧酸還原酶活性的第一外源多肽相同或不同。具有醇脫氫酶活性的外源多肽可以是具有醇脫氫酶活性的第二外源多肽。第二外源醇脫氫酶可以與第一外源醇脫氫酶相同或不同。參見圖8。
本文所述的可用于生產一種或多種C6結構單元的任何酶可以與對應的野生型酶具有至少70%的序列同一性(同源性)(例如至少75%,80%,85%,90%,91%,92%,93%,94%,95%,96%,97%,98%,99%或100%)。應當理解,可以基于成熟酶(例如,除去任何信號序列)或基于未成熟酶(例如,包括任何信號序列)確定序列同一性。還應當理解,起始甲硫氨酸殘基可以存在于或可以不存在于本文所述的任何酶序列上。
例如,本文描述的具有硫酯酶活性的多肽可以與短乳桿菌硫酯酶的氨基酸序列(參見GenBank登錄號ABJ63754.1,SEQ ID NO:1)或植物乳桿菌硫酯酶的氨基酸序列(參見GenBank登錄號CCC78182.1,SEQ ID NO:NO:2)具有至少70%的序列同一性(同源性)(例如至少75%,80%,85%,90%,91%,92%,93%,94%,95%,96%,97%,98%,99%或100%)。參見圖21。
例如,本文所述的具有羧酸還原酶活性的多肽可以與海分枝桿菌羧酸還原酶(參見Genbank登錄號ACC40567.1,SEQ ID NO:3),恥垢分枝桿菌羧酸還原酶(參見Genbank登錄號ABK71854.1,SEQ ID NO:4),Segniliparus rugosus羧酸還原酶(參見Genbank登錄號EFV11917.1,SEQ ID NO:5),馬賽分枝桿菌羧酸還原酶(參見Genbank登錄號EIV11143.1,SEQ ID NO:6)或Segniliparus rotundus羧酸還原酶(參見Genbank登錄號ADG98140.1,SEQ ID NO:7)的氨基酸序列具有至少70%的序列同一性(同源性)(例如至少75%,80%,85%,90%,91%,92%,93%,94%,95%,96%,97%,98%,99%或100%)。參見圖21。
例如,本文描述的具有ω-轉氨酶活性的多肽可以與紫色色桿菌ω-轉氨酶(參見Genbank登錄號AAQ59697.1,SEQ ID NO:8),銅綠假單胞菌ω-轉氨酶(參見Genbank登錄號AAG08191.1,SEQ ID NO:9),丁香假單胞菌ω- 轉移酶(參見Genbank登錄號AAY39893.1,SEQ ID NO:10),球形紅細菌ω-轉移酶(參見Genbank登錄號ABA81135.1,SEQ ID NO:11),大腸桿菌ω-轉移酶(參見Genbank登錄號AAA57874.1,SEQ ID NO:12),或河流孤菌ω-轉移酶(參見Genbank登錄號AEA39183.1,SEQ ID NO:13)的氨基酸序列具有至少70%的序列同一性(同源性)(例如至少75%,80%,85%,90%,91%,92%,93%,94%,95%,96%,97%,98%,99%或100%)。參見圖21。
例如,本文所述的具有磷酸泛酰巰基乙胺基轉移酶活性的多肽可以與枯草芽孢桿菌磷酸泛酰巰基乙胺基轉移酶(參見Genbank登錄號CAA44858.1,SEQ ID NO:14)或諾卡氏菌屬物種NRRL 5646磷酸泛酰巰基乙胺基轉移酶(參見Genbank登錄號ABI83656.1,SEQ ID NO:15)的氨基酸序列具有至少70%的序列同一性(同源性)(例如至少75%,80%,85%,90%,91%,92%,93%,94%,95%,96%,97%,98%,99%或100%)。參見圖21。
例如,本文所述的具有烷烴1-單加氧酶活性的多肽可以與單胞菌屬物種JS666單加氧酶(參見Genbank登錄號ABE47160.1,SEQ ID NO:16),分枝桿菌屬物種HXN-1500單加氧酶(參見Genbank登錄號CAH04396.1,SEQ ID NO:17)或南非分枝桿菌單加氧酶(參見Genbank登錄號ACJ06772.1,SEQ ID NO:18)的氨基酸序列具有至少70%的序列同一性(同源性)(例如至少75%,80%,85%,90%,91%,92%,93%,94%,95%,96%,97%,98%,99%或100%)。參見圖21。
例如,本文所述的具有氧化還原酶活性的多肽可以與單胞菌屬物種JS666氧化還原酶(參見Genbank登錄號ABE47159.1,SEQ ID NO:19)或分枝桿菌屬物種HXN-1500氧化還原酶(參見Genbank登錄號CAH04397.1,SEQ ID NO:20)的氨基酸序列具有至少70%的序列同一性(同源性)(例如至少75%,80%,85%,90%,91%,92%,93%,94%,95%,96%,97%,98%,99%或100%)。參見圖21。
例如,本文所述的鐵氧還蛋白多肽可以與單胞菌屬物種JS666鐵氧還原蛋白(參見Genbank登錄號ABE47158.1,SEQ ID NO:21)或分枝桿菌屬物種HXN-1500鐵氧還蛋白(參見Genbank登錄號CAH04398.1,SEQ ID NO:22)的氨基酸序列具有至少70%的序列同一性(同源性)(例如至少75%,80%,85%,90%,91%,92%,93%,94%,95%,96%,97%,98%,99%或100%)。參見圖21。
例如,本文所述的具有庚二酰-[acp]合酶活性的多肽可以與枯草芽孢桿菌庚二酰-[acp]合酶(參見Genbank登錄號AAB17462.1,SEQ ID NO:23)的氨基酸序列具有至少70%的序列同一性(同源性)(例如至少75%,80%,85%,90%,91%,92%,93%,94%,95%,96%,97%,98%,99%或100%)。參見圖21。
可以如下測定兩種氨基酸序列間的百分比同一性(同源性)。首先,使用來自含有BLASTP第2.0.14版的單機版BLASTZ的BLAST 2Sequences(Bl2seq)程序比對氨基酸序列。此單機版BLASTZ可以獲自Fish&Richardson的網站(例如www.fr.com/blast/)或美國政府國立生物技術信息中心網站(www.ncbi.nlm.nih.gov)。解釋如何使用Bl2seq程序的用法說明可以參見伴隨BLASTZ的自述文件。Bl2seq使用BLASTP算法實施兩種氨基酸序列之間的比較。為了比較兩種氨基酸序列,如下設置Bl2seq的選項:-i設置為含有要比較的第一氨基酸序列的文件(例如C:\seq1.txt);-j設置為含有要比較的第二氨基酸序列的文件(例如C:\seq2.txt);-p設置為blastp;-o設置為任何期望的文件名稱(例如C:\output.txt);并且所有其它選項保持為其缺省設置。例如,可以使用以下命令來產生含有兩種氨基酸序列間的比較的輸出文件:C:\Bl2seq–i c:\seq1.txt–j c:\seq2.txt–p blastp–o c:\output.txt。如果兩種比較序列共享同源性(同一性),那么指定的輸出文件會呈現那些同源性區作為比對序列。如果兩種比較序列不共享同源性(同一性),那么指定的輸出文件不會呈現比對序列。可以對核酸序列遵循相似的規程,只是使用blastn。
一旦比對,通過計算相同氨基酸殘基在這兩種序列中呈現的位置的數目確定匹配數目。通過用匹配數目除以全長多肽氨基酸序列的長度,接著將所得的數值乘以100來確定百分比同一性(同源性)。注意到百分比同一性(同源性)值被四舍五入到最近的十分位。例如,78.11、78.12、78.13、和78.14被向下四舍五入到78.1,而78.15、78.16、78.17、78.18、和78.19被向上四舍五入到78.2。還注意到長度值會總是整數。
應當領會,許多核酸可以編碼具有特定氨基酸序列的多肽。遺傳密碼的簡并性是本領域中公知的;即對于許多氨基酸,存在有超過一種充當氨基酸密碼子的核苷酸三聯體。例如,可以修飾給定酶的編碼序列中的密碼子,從而獲得特定物種(例如細菌或真菌)中的最佳表達,這使用適合于所述物種的密碼子偏愛表進行。
也可以在本文件的方法中使用本文中描述的任何酶的功能性片段。如本文中使用的,術語“功能性片段”指具有至少25%(例如至少30%;40%;50%;60%;70%;75%;80%;85%;90%;95%;98%;99%;100%;或甚至大于100%)的相應的成熟、全長、野生型蛋白質活性的蛋白質的肽片段。功能性片段一般但不總是可以由蛋白質的連續區構成,其中該區具有功能性活性。
此文件還提供了(i)本文件的方法中使用的酶的功能性變體和(ii)上文描述的功能性片段的功能性變體。相對于相應的野生型序列,酶和功能性片段的功能性變體可以含有添加、缺失、或取代。具有取代的酶一般會具有不超過50(例如不超過1、2、3、4、5、6、7、8、9、10、12、15、20、25、30、35、40、或50)處氨基酸取代(例如保守取代)。這適用于本文中描述的任何酶和功能性片段。保守取代是用一種氨基酸取代具有相似特征的另一種。保守取代包括下列組內的取代:纈氨酸、丙氨酸和甘氨酸;亮氨酸、纈氨酸、和異亮氨酸;天冬氨酸和谷氨酸;天冬酰胺和谷氨酰胺;絲氨酸、半胱氨酸、和蘇氨酸;賴氨酸和精氨酸;和苯丙氨酸和酪氨酸。非極性疏水性氨基酸包括丙氨酸、亮氨酸、異亮氨酸、纈氨酸、脯氨酸、苯丙氨酸、色氨酸和甲硫氨酸。極性中性氨基酸包括甘氨酸、絲氨酸、蘇氨酸、半胱氨酸、酪氨酸、天冬酰胺和谷氨酰胺。帶正電荷的(堿性)氨基酸包括精氨酸、賴氨酸和組氨酸。帶負電荷的(酸性)氨基酸包括天冬氨酸和谷氨酸。上文提及的極性、堿性或酸性組的一種成員被相同組的另一種成員的任何取代可以視為保守取代。比較而言,非保守取代是用一種氨基酸取代具有不同特征的另一種。
缺失變體可以缺乏1、2、3、4、5、6、7、8、9、10、11、12、13、14、15、16、17、18、19、或20個氨基酸的區段(具有兩種或更多種氨基酸)或非連續的單一氨基酸。添加(添加變體)包括融合蛋白,其含有:(a)本文中描述的任何酶或其片段;和(b)內部或末端(C或N)無關或異源氨基酸序列。在此類融合蛋白的背景中,術語“異源氨基酸序列”指與(a)不同的氨基酸序列。異源序列可以是例如用于純化重組蛋白的序列(例如FLAG、多組氨酸(例如六組氨酸)、凝集素(HA)、谷胱甘肽-S-轉移酶(GST)、或麥芽糖結合蛋白(MBP))。異源序列也可以是可用作可檢測標志物的蛋白質,例如螢光素酶、綠色熒光蛋白(GFP)、或氯霉素乙酰基轉移酶(CAT)。在一些實施方案中,融合蛋白含有來自另一種蛋白質的信號序列。在某些宿主細胞(例如酵母宿主細胞)中,可以經由使用異源信號序列提高靶蛋白的表達和/或分泌。在一些實施方案中,融合蛋白可以含有可用于例如引發免疫應答以生成抗體的載體(例如KLH)或ER或高爾基體保留信號。異源序列可以是不同長度的,并且在一些情況中可以是比與異源序列附接的全長靶蛋白質更長的序列。
在一些實施方案中,如下實現十二酰基-[acp]或辛酰基-[acp]氧化切割成一種或多種C6脂族主鏈:通過在特定宿主中酰基載體蛋白的蛋白質工程化,建立由BioI編碼的庚二酰-[acp]合酶內的碳-6和碳-7位置的底物排列(alignment)。
在一些實施方案中,如下實現十二酰基-[acp]或辛酰基-[acp]氧化切割成一種或多種C6脂族主鏈:通過在特定宿主中合成酰基載體蛋白和脂肪酸之間的修飾的磷酸泛酰巰基乙胺接頭,建立由BioI編碼的庚二酰-[acp]合酶內的碳-6和碳-7位置的底物排列。
在一些實施方案中,如下實現十二酰基-[acp]或辛酰基-[acp]氧化切割成一種或多種C6脂肪族主鏈:通過由BioI編碼的庚二酰-[acp]合酶(SEQ ID NO:23)的酶工程化,建立用于氧化切割的酶中的碳-6和碳-7位置的底物排列。
本文所述途徑的反應可以在一種或多種細胞(例如宿主細胞)株中進行,所述細胞株(a)天然表達一種或多種相關酶,(b)經遺傳工程化而表達一種或多種相關酶,或(c)天然表達一種或多種相關酶并經遺傳工程化而表達一種或多種相關酶。或者,可以從以上任意類型的宿主細胞中提取相關酶,并以純化或半純化形式使用。此外,這些提取物包括可以作為相關酶來源使用的裂解物(如細胞裂解物)。在本申請提供的方法中,所有步驟可以在宿主細胞中進行,所有步驟可以使用提取的酶進行,或者一些步驟可以在細胞中進行,其它步驟可以使用提取的酶進行。如本文所述,重組宿主可包括核酸,該核酸編碼如下文更詳細描述的具有合酶活性的多肽,具有脫氫酶活性的多肽,具有還原酶活性的多肽,具有單加氧酶活性的多肽,具有硫酯酶活性的多肽,具有脫酰基酶活性的多肽,具有轉移酶活性的多肽,或具有轉氨酶活性的多肽的一種或多種。
另外,可以使用本文中描述的分離的酶,使用來自宿主微生物的裂解物(例如細胞裂解物)作為酶來源,或者使用來自不同宿主微生物的多種裂解物作為酶來源體外進行一種或多種C6結構單元的產生。
產生用于轉化為C6結構單元的C6脂族主鏈的酶
用于轉化為一種或多種C6結構單元的C6脂肪族主鏈可以自使用乙酰CoA和丙二酰CoA作為中心代謝物的脂肪酸生物合成,和具有用于十二酰基-[acp]或辛酰基-[acp]前體的氧化切割的庚二酰-[acp]合酶活性的多肽形成。適合的庚二酰-[acp]合酶具有接受C8或C12酰基-[acp]底物,并氧化切割底物的C6和C7碳之間的C-C鍵的能力,以產生6-氧代己酰-[acp]和乙醛(當辛酰基-[acp]是底物時)或6-氧代己酰-[acp]和己醛(當十二酰基-[acp]是底物時),并且可以與由來自枯草芽孢桿菌的bioI編碼的野生型庚二酰-[acp]合酶具有至少70%的序列同一性。野生型庚二酰-[acp]合酶分類在EC 1.14.15.12下,并且通常氧化切割酰基-[acp]底物的C7和C8碳之間的C-C鍵。參見Green等,J.Biol.Inorg.Chem.,2001,6,523-533;Cryle and De Voss,Chem.Commun.(Camb.),2004,7,86-87;Cryle and Schlichting,Proc.Natl.Acad.Sci.USA,2008,105,15696–15701。
在一些實施方案中,由具有庚二酰-[acp]合酶活性(例如,與SEQ ID NO:23所示的氨基酸序列具有70%的序列同一性)的多肽產生的半醛產物通過具有醛脫氫酶活性的多肽轉化為它們相應的羧酸。參見圖1。
在一些實施方案中,醛脫氫酶歸類在EC 1.2.1.3或EC 1.2.1.4下。參見圖1。
在C6結構單元的生物合成中產生末端羧基的酶
如圖1和圖2中描繪,可以使用具有硫酯酶活性的多肽或具有醛脫氫酶活性的多肽來酶促形成末端羧基。
在一些實施方案中,導致C6結構單元合成的第一或第二末端羧基通過歸類在EC 1.2.1.-(例如,EC 1.2.1.3,EC 1.2.1.4,EC 1.2.1.16,EC 1.2.1.20,EC 1.2.1.63或EC 1.2.1.79)下的醛脫氫酶酶促形成。例如,第一末端羧基可以由分類在EC 1.2.1.4(Ho&Weiner,Journal of Bacteriology,2005,187(3),1067–1073)或分類為EC 1.2.1.3(Guerrillot&Vandecasteele,Eur.J.Biochem.,1977,81,185–192)下的醛脫氫酶酶促形成。例如,導致己二酸合成的第二末端羧基可以通過分類在EC 1.2.1.-(例如EC 1.2.1.3,EC 1.2.1.16,EC 1.2.1.20,EC 1.2.1.63或EC 1.2.1.79)下的醛脫氫酶,如CpnE,ChnE或ThnG的基因產物酶促形成(參見例如Iwaki等,Appl.Environ.Microbiol.,1999,65(11),5158–5162;或López-Sánchez等,Appl.Environ.Microbiol.,2010,76(1),110-118)。ThnG的基因產物是7-氧代庚酸脫氫酶。ChnE的基因產物是6-氧代己酸脫氫酶。
在一些實施方案中,導致己二酸合成的第二末端羧基由歸類在EC 3.1.2.-下的硫酯酶,如fatB,tesA的基因產物,或具有SEQ ID NO:1或SEQ ID NO:2中所示的氨基酸序列的硫酯酶酶促形成(參見例如Jing等,BMC Biochemistry,2011,12,44;Cantu等,Protein Science,2010,19,1281–1295;Zhuang等,Biochemistry,2008,47(9),2789–2796;或Naggert等,J.Biol.Chem.,1991,266(17),11044–11050)。
在C6結構單元的生物合成中產生末端胺基的酶
如圖3、圖4、圖5和圖6中描繪,可以使用具有ω-轉氨酶活性的多肽或具有脫酰基酶活性的多肽酶促形成末端胺基。
在一些實施方案中,末端氨基可以由歸類于例如EC 2.6.1.-,例如EC 2.6.1.18、EC 2.6.1.19、EC 2.6.1.29、EC 2.6.1.48或EC 2.6.1.82下的ω-轉氨酶(如獲得自紫色色桿菌(GenBank登錄號AAQ59697.1,SEQ ID NO:8))、銅綠假單胞菌(GenBank登錄號AAG08191.1,SEQ ID NO:9)、丁香假單胞菌(GenBank登錄號AAY39893.1,SEQ ID NO:10)、球形紅細菌(GenBank登錄號ABA81135.1,SEQ ID NO:11)、河流弧菌(GenBank登錄號AEA39183.1,AEA39183.1,SEQ ID NO:13)、灰色鏈霉菌(Streptomyces griseus)或Clostridium viride)酶促形成。參見圖3。
可用于本文所述的方法和宿主中的另外的ω-轉氨酶來自大腸桿菌(GenBank登錄號AAA57874.1,SEQ ID NO:12)。一些歸類于例如EC 2.6.1.29或EC 2.6.1.82下的ω-轉氨酶是二胺ω-轉氨酶。
在一些實施方案中,導致6-氨基己酸合成的第一末端胺基由歸類于2.6.1.18下的ω-轉氨酶(如獲得自河流弧菌(SEQ ID NO:13)或紫色色桿菌(SEQ ID NO:8)),歸類于EC 2.6.1.19下(如獲得自灰色鏈霉菌),或分類于EC 2.6.1.48下(如獲得自梭菌屬)酶促形成。還可以使用具有SEQ ID NO:9,10,和11中所示的氨基酸序列的ω-轉氨酶。
來自紫色色桿菌的可逆ω-轉氨酶已經表明接受6-氨基己酸作為氨基供體,從而在己二酸半醛中形成第一末端胺基的類似活性(Kaulmann等,Enzyme and Microbial Technology,2007,41,628-637)。
來自灰色鏈霉菌的可逆的4-氨基丁酸:2-氧代戊二酸轉氨酶已經表明用于將6-氨基己酸轉化為己二酸半醛的類似活性(Yonaha等,Eur.J.Biochem.,1985,146,101-106)。
來自Clostridium viride的可逆的5-氨基戊酸轉氨酶已經表明用于將6-氨基己酸轉化為己二酸半醛的類似活性(Barker等,The Journal of Biological Chemistry,1987,262(19),8994-9003)。
在一些實施方案中,導致六亞甲基二胺合成的第二末端胺基由歸類于EC 2.6.1.29或歸類于EC 2.6.1.82下的二胺轉氨酶,如YgjG的基因產物酶促形成。可以使用具有SEQ ID NO:8-13所示的氨基酸序列的ω-轉氨酶可以用于生物合成六亞甲基二胺。
YgjG的基因產物接受寬范圍的二胺碳鏈長度底物,諸如腐胺,尸胺和亞精胺(Samsonova等,BMC Microbiology,2003,3:2)。
來自大腸桿菌菌株B的二胺轉氨酶已經表明對1,6-二氨基己烷的活性(Kim,The Journal of Chemistry,1963,239(3),783-786)。
在一些實施方案中,通過分類于例如EC 3.5.1.17下的脫酰基酶,如酰基賴氨酸脫酰基酶,在N 6-乙酰基-1,6-二氨基己烷中酶促形成第二末端胺基,其導致庚二胺的形成。
在C6結構單元的生物合成中產生末端羥基的酶
如圖7和圖8中描繪,可以使用具有烷烴1-單加氧酶活性的多肽或具有醇脫氫酶活性的多肽來酶促形成末端羥基。
在一些實施方案中,導致合成C6結構單元的第一末端羥基通過烷烴1-單加氧酶(如由alkBGT編碼)或細胞色素P450(如來自CYP153家族(如CYP153A)酶促形成(參見,例如Van Beilen&Funhoff,Current Opinion in Biotechnology,2005,16,308–314;Koch et al.,Appl.Environ.Microbiol.,2009,75(2),337-344;or Nieder and Shapiro,Journal of Bacteriology,1975,122(1),93-98)。見例如SEQ ID NO.16-18。
CYP153A家族的末端烷烴1-單加氧酶和alkB單加氧酶的底物特異性已經成功地擴大(Koch等,2009,同上)。盡管在體外觀察到CYP153A6的非末端羥基化,但在體內僅發生1-羥基化(Funhoff等,Journal of Bacteriology,2006,188(14),5220–5227)。
在一些實施方案中,導致6-羥基己酸合成的末端羥基通過歸類在EC 1.1.1.-(例如EC 1.1.1.2)下的醇脫氫酶,如YMR318C,cpnD或gabD的基因產物,或分類在EC 1.1.1.258下,如ChnD的基因產物酶促形成。分類在EC 1.1.1.258下的醇脫氫酶是6-羥基己酸脫氫酶。
在一些實施方案中,導致1,6己二醇合成的第二末端羥基通過分類在EC 1.1.1.-(例如EC 1.1.1.1,EC 1.1.1.2,EC 1.1.1.21,或EC 1.1.1.184)下的醇脫氫酶,如YMR318C或YqhD的基因產物或具有GenBank登錄號CAA81612.1的蛋白酶促合成。
生物化學途徑
使用長鏈酰基-[acp]脂肪酸合成中間物作為前體,導致C6脂族主鏈,己二酰-[acp]和己酸的途徑
在一些實施方案中,如下從十二酰基-[acp]合成己二酰-[acp]和己酸:通過具有庚二酰-[acp]合酶活性的多肽(例如,與BioI的基因產物具有至少70%的序列同一性,參見Genbank登錄號AAB17462.1,SEQ ID NO:23),將十二酰基-[acp]轉化成蘇型-6,7-二羥基十二酰基-[acp];然后通過具有庚二酰-[acp]合酶活性的多肽(例如,與BioI的基因產物具有至少70%的序列同一性,參見Genbank登錄號AAB17462.1,SEQ ID NO:23)轉化為6-氧代己酰-[acp]和己醛;然后通過分類在例如EC 1.2.1.4或EC 1.2.1.3下的醛脫氫酶轉化為己二酰-[acp]和己酸。參見例如圖1。
在一些實施方案中,如下從辛酰基-[acp]合成己二酰-[acp]和乙酸:通過具有庚二酰基-[acp]合酶活性的多肽(例如,與BioI的基因產物具有至少70%的序列同一性,參見Genbank登錄號AAB17462.1,SEQ ID NO:23)將辛酰基-[acp]轉化成蘇型-6,7-二羥基辛酰基-[acp];隨后通過具有庚二酰基-[acp]合酶活性的多肽(例如,與BioI的基因產物具有至少70%的序列同一性,參見Genbank登錄號AAB17462.1,SEQ ID NO:23)轉化為6-氧代己酰-[acp]和乙醛;然后通過歸類在例如EC 1.2.1.4或EC 1.2.1.3下的醛脫氫酶轉化為己二酰-[acp]和乙酸。參見例如圖1。
使用己酸或己二酰-[acp]中心前體到己二酸的途徑
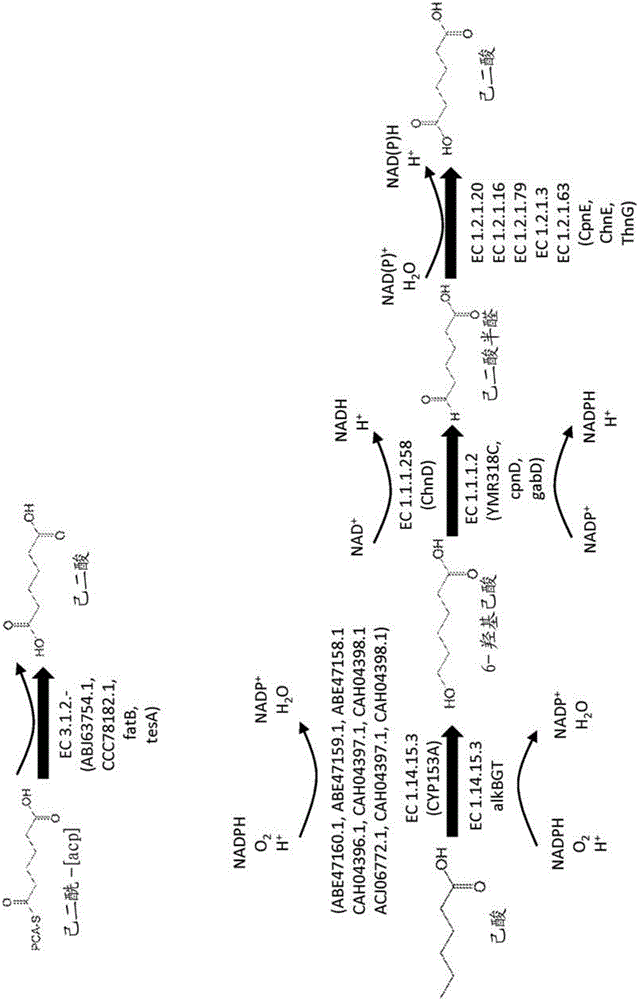
在一些實施方案中,如下從中心前體己酸合成己二酸:通過烷烴1-單加氧酶如alkBor或來自CYP153A家族,如單胞菌屬物種JS666單加氧酶(參見Genbank登錄號ABE47160.1,SEQ ID NO:16),分枝桿菌屬物種HXN-1500單加氧酶(參見Genbank登錄號CAH04396.1,SEQ ID NO:17),或南非分枝桿菌單加氧酶(參見Genbank登錄號ACJ06772.1,SEQ ID NO:18)將己酸轉化為6-羥基己酸;隨后通過醇脫氫酶(例如,分類在EC 1.1.1.2或EC 1.1.1.258下)如YMR318C,cpnD,gabD或ChnD的基因產物將6-羥基己酸轉化為己二酸半醛;隨后通過醛脫氫酶(例如,分類在EC 1.2.1.-,EC 1.2.1.3,EC 1.2.1.16,EC 1.2.1.20,EC 1.2.1.63或EC 1.2.1.79下)如ThnG,ChnE或CpnE的基因產物將己二酸半醛轉化為己二酸。參見圖2。
由YMR318C編碼的醇脫氫酶具有廣泛的底物特異性,包括C6醇的氧化。
在一些實施方案中,如下從中心前體己二酰-[acp]合成己二酸:通過硫酯酶(例如,分類在EC 3.1.2.-下),如來自短乳桿菌(參見GenBank登錄號ABJ63754.1,SEQ ID NO:1),植物乳桿菌(參見GenBank登錄號CCC78182.1,SEQ ID NO:2),或fatB或tesA的基因產物將己二酰-[acp]轉化為己二酸。參見圖2。
使用己二酰-[acp]或己酸作為中心前體到6-氨基己酸的途徑
在一些實施方案中,如下從中心前體己酸合成6-氨基己酸:通過烷烴1-單加氧酶,如alkB或來自CYP153A家族,如單胞菌屬物種JS666單加氧酶(參見Genbank登錄號ABE47160.1,SEQ ID NO:16),分枝桿菌屬物種HXN-1500單加氧酶(參見Genbank登錄號CAH04396.1,SEQ ID NO:17),或南非分枝桿菌單加氧酶(參見Genbank登錄號ACJ06772.1,SEQ ID NO:18)將己酸轉化為6-羥基己酸;隨后通過醇脫氫酶(例如,分類在EC 1.1.1.2或EC 1.1.1.258下),如YMR318C,cpnD,gabD或ChnD的基因產物將6-羥基己酸轉化為己二酸半醛;隨后通過ω-轉氨酶(例如,分類在EC 2.6.1.-下),如來自紫色色桿菌(Genbank登錄號AAQ59697.1,SEQ ID NO:8),銅綠假單胞菌(Genbank登錄號AAG08191.1,SEQ ID NO:9),丁香假單胞菌(Genbank 登錄號AAY39893.1,SEQ ID NO:10),球形紅細菌酶(Genbank登錄號ABA81135.1,SEQ ID NO:11),或河流孤菌(Genbank登錄號AEA39183.1,SEQ ID NO:13)將己二酸半醛轉化為6-氨基己酸。參見圖3。
在一些實施方案中,如下從中心前體己二酰-[acp]合成6-氨基己酸:通過硫酯酶(例如分類在EC 3.1.2.-分類下),如來自短乳桿菌(參見GenBank登錄號ABJ63754.1,SEQ ID NO:1),植物乳桿菌(參見GenBank登錄號CCC78182.1,SEQ ID NO:2),或fatB或tesA的基因產物將己二酰-[acp]轉化為己二酸;隨后通過例如分類在例如EC 1.2.99.6下的羧酸還原酶例如car的基因產物(例如來自Segniliparus rugosus,Genbank登錄號EFV11917.1,SEQ ID NO:5或來自Segniliparus rotundus,Genbank登錄號ADG98140.1,SEQ ID NO:7,與磷酸泛酰巰基乙胺基轉移酶增強劑(例如,由來自枯草芽孢桿菌的sfp基因(SEQ ID NO:14)或來自諾卡氏菌屬的npt基因(SEQ ID NO:15)編碼)組合或來自灰色鏈霉菌的GriC和GriD的基因產物將己二酸轉化為己二酸半醛(Suzuki等人,J.Antibiot.,2007,60(6),380-387);隨后通過ω-轉氨酶,如來自紫色色桿菌(Genbank登錄號AAQ59697.1,SEQ ID NO:8),銅綠假單胞菌(Genbank登錄號AAG08191.1,SEQ ID NO:9),丁香假單胞菌(Genbank登錄號AAY39893.1,SEQ ID NO:10),球形紅細菌(Genbank登錄號ABA81135.1,SEQ ID NO:11),或河流孤菌(Genbank登錄號AEA39183.1,SEQ ID NO:13)轉化為6-氨基己酸。見圖3。
在一些實施方案中,通過分類在EC 3.5.2.-下的酰胺水解酶將6-氨基己酸轉化為己內酰胺。
使用6-氨基己酸作為中心前體到六亞甲基二胺的途徑
在一些實施方案中,如下從中心前體6-氨基己酸合成六亞甲基二胺:通過羧酸還原酶(例如,分類在EC 1.2.99.6下),如car的基因產物(例如,SEQ ID NO:3-7)與npt(SEQ ID NO:15)或sfp(SEQ ID NO:14)的基因產物的組合將6-氨基己酸轉化為6-氨基己醛,或者備選可以使用GriC&GriD的基因產物(Suzuki等人,2007,同上)代替car的基因產物;隨后通過ω-轉氨酶(例如,分類在EC 2.6.1.18,EC 2.6.1.19,EC 2.6.1.48,EC 2.6.1.29或EC 2.6.1.82下),如來自紫色色桿菌(Genbank登錄號AAQ59697.1,SEQ ID NO:8),銅綠假單胞菌(Genbank登錄號AAG08191.1,SEQ ID NO:9),丁香假單胞菌(Genbank登錄號AAY39893.1,SEQ ID NO:10),球形紅細菌酶(Genbank登錄號ABA81135.1,SEQ ID NO:11),大腸桿菌(參見Genbank登錄號AAA57874.1,SEQ ID NO:12),或河流孤菌(Genbank登錄號AEA39183.1,SEQ ID NO:13)將6-氨基己醛轉化為六亞甲基二胺。見圖4。
由car和增強劑npt的基因產物編碼的羧酸還原酶具有廣泛的底物特異性,包括末端雙官能的C4和C5羧酸(Venkitasubramanian等,Enzyme and Microbial Technology,2008,42,130–137)。
在一些實施方案中,如下從中心前體6-氨基己酸合成六亞甲基二胺:通過N-乙酰基轉移酶,如賴氨酸N-乙酰轉移酶(其分類在例如EC 2.3.1.32下),將6-氨基己酸轉化為N6-乙酰基-6-氨基己酸;隨后通過分類在例如EC 1.2.99.6下的羧酸還原酶,如來自Segniliparus rugosus(參見Genbank登錄號EFV11917.1,SEQ ID NO:5),馬賽分枝桿菌(參見Genbank登錄號EIV11143.1,SEQ ID NO:6)或Segniliparus rotundus(Genbank登錄號ADG98140.1,SEQ ID NO:7)的羧酸還原酶與磷酸泛酰巰基乙胺基轉移酶增強劑(例如,由來自枯草芽孢桿菌的sfp基因或來自諾卡氏菌屬的npt基因編碼)組合將N6-乙酰基-6-氨基己酸轉化為N6-乙酰基-氨基己醛,或者備選可以使用來自灰色鏈霉菌的GriC和GriD的基因產物(Suzuki等人,2007,同上)代替car的基因產物;隨后通過分類在例如EC 2.6.1.-下的ω-轉氨酶,如來自紫色色桿菌(Genbank登錄號AAQ59697.1,SEQ ID NO:8),銅綠假單胞菌(Genbank登錄號AAG08191.1,SEQ ID NO:9),丁香假單胞菌(Genbank登錄號AAY39893.1,SEQ ID NO:10),球形紅細菌酶(Genbank登錄號ABA81135.1,SEQ ID NO:11),大腸桿菌(參見Genbank登錄號AAA57874.1,SEQ ID NO:12),或河流孤菌(Genbank登錄號AEA39183.1,SEQ ID NO:13)將N6-乙酰基-氨基己醛轉化為N6-乙酰基-1,6-二氨基己烷;隨后通過例如分類在EC 3.5.1.17下的脫酰基酶將N6-乙酰基-1,6-二氨基己烷轉化為六亞甲基二胺。見圖5。
使用己二酸半醛作為中心前體到六亞甲基二胺的途徑
在一些實施方案中,如下從中心前體己二酸半醛合成六亞甲基二胺:通過羧酸還原酶(例如分類在EC 1.2.99.6下),如來自Segniliparus rotundus(參見Genbank登錄號ADG98140.1,SEQ ID NO:7)與磷酸泛酰巰基乙胺轉移酶增強劑(例如由來自枯草芽孢桿菌的sfp基因或來自諾卡氏菌屬的npt基因編碼)的組合將己二酸半醛轉化為1,6-己二醛,或者來自灰色鏈霉菌的GriC和GriD的基因產物(Suzuki等人,2007,同上)可以用于替換car的基因產物;隨后通過分類在例如EC 2.6.1.18,EC 2.6.1.19,EC 2.3.1.29,EC 2.6.1.48或EC 2.3.1.82下的轉氨酶將1,6-己二醛轉化為6-氨基己醛;隨后通過例如分類在EC 2.6.1.18,EC 2.6.1.19,EC 2.6.1.48,EC 2.6.1.29或EC 2.6.1.82下的ω-轉氨酶,如來自紫色色桿菌(Genbank登錄號AAQ59697.1,SEQ ID NO:8),銅綠假單胞菌(Genbank登錄號AAG08191.1,SEQ ID NO:9),丁香假單胞菌(Genbank登錄號AAY39893.1,SEQ ID NO:10),球形紅細菌酶(Genbank登錄號ABA81135.1,SEQ ID NO:11),大腸桿菌(參見Genbank登錄號AAA57874.1,SEQ ID NO:12),或河流孤菌(Genbank登錄號AEA39183.1,SEQ ID NO:13)將6-氨基己醛轉化為六亞甲基二胺。見圖4。
使用6-羥基己酸作為中心前體到六亞甲基二胺的途徑
在一些實施方案中,如下從中心前體6-羥基己酸合成六亞甲基二胺:通過羧酸還原酶(分類在例如EC 1.2.99.6下),如來自海分枝桿菌(參見Genbank登錄號ACC40567.1,SEQ ID NO:3),恥垢分枝桿菌(參見Genbank登錄號ABK71854.1,SEQ ID NO:4),Segniliparus rugosus(參見Genbank登錄號EFV11917.1,SEQ ID NO:5),馬賽分枝桿菌(參見Genbank登錄號EIV11143.1,SEQ ID NO:6),或Segniliparus rotundus(參見Genbank登錄號ADG98140.1,SEQ ID NO:7)(例如與磷酸泛酰巰基乙胺基轉移酶增強劑(如,由來自枯草芽孢桿菌的sfp基因或來自諾卡氏菌屬的npt基因編碼)組合),或者來自灰色鏈霉菌的GriC和GriD的基因產物(Suzuki等人,2007,同上)將6-羥基己酸轉化為6-羥基己醛;隨后通過分類在例如EC 2.6.1.-下的ω-轉氨酶,如來自紫色色桿菌(Genbank登錄號AAQ59697.1,SEQ ID NO:8),銅綠假單胞菌(Genbank登錄號AAG08191.1,SEQ ID NO:9),丁香假單胞菌(Genbank登錄號AAY39893.1,SEQ ID NO:10),球形紅細菌酶(Genbank登錄號ABA81135.1,SEQ ID NO:11),大腸桿菌(參見Genbank登錄號AAA57874.1,SEQ ID NO:12),或河流孤菌(Genbank登錄號AEA39183.1,SEQ ID NO:13)將6-羥基己醛轉化為6-氨基己醇;隨后通過分類在例如EC 1.1.1.1下的醇脫氫酶(例如來自嗜熱脂肪地芽孢桿菌的具有GenBank登錄號CAA81612.1或由YMR318C或YqhD編碼的蛋白)將6-氨基己醇轉化為6-氨基己醛;隨后通過分類在例如EC 2.6.1.-下的ω-轉氨酶,如來自紫色色桿菌(Genbank登錄號AAQ59697.1,SEQ ID NO:8),銅綠假單胞菌(Genbank登錄號AAG08191.1,SEQ ID NO:9),丁香假單胞菌(Genbank登錄號AAY39893.1,SEQ ID NO:10),球形紅細菌酶(Genbank登錄號ABA81135.1,SEQ ID NO:11),大腸桿菌(參見Genbank登錄號AAA57874.1,SEQ ID NO:12),或河流孤菌(Genbank登錄號AEA39183.1,SEQ ID NO:13)將6-氨基己醛轉化為六亞甲基二胺。見圖6。
使用己二酰-[acp]或己酸作為中心前體到1,6-己二醇的途徑
在一些實施方案中,如下從中心前體己酸合成6-羥基己酸:通過烷烴1-單加氧酶,如alkB或來自CYP153A家族,如單胞菌屬物種JS666單加氧酶(參見Genbank登錄號ABE47160.1,SEQ ID NO:16),分枝桿菌屬物種HXN-1500單加氧酶(參見Genbank登錄號CAH04396.1,SEQ ID NO:17),南非分枝桿菌單加氧酶(參見Genbank登錄號ACJ06772.1,SEQ ID NO:18)將己酸轉化為6-羥基己酸。見圖7。
在一些實施方案中,如下從中心前體己二酰-[acp]合成6-羥基己酸:通過硫酯酶(例如,分類在EC 3.1.2.-下),如來自短乳桿菌(參見GenBank登錄號ABJ63754.1,SEQ ID NO:1)或植物乳桿菌(參見GenBank登錄號CCC78182.1,SEQ ID NO:2),或fatB或tesA的基因產物將己二酰-[acp]轉化為己二酸;隨后通過羧酸還原酶(例如,分類在EC 1.2.99.6下),如來自Segniliparus rugosus(參見Genbank登錄號EFV11917.1,SEQ ID NO:5)或來自Segniliparus rotundus(參見Genbank登錄號ADG98140.1,SEQ ID NO:7)與磷酸泛酰巰基乙胺基轉移酶增強劑(例如,由來自枯草芽孢桿菌的sfp基因編碼)或GriC&GriD的基因產物轉化為己二酸半醛;隨后通過醇脫氫酶(例如,分類在EC 1.1.1.2或EC 1.1.1.258下),如YMR318C,ChnD,cpnD或gabD的基因產物轉化為6-羥基己酸。見圖7。
在一些實施方案中,如下從中心前體6-羥基己酸合成1,6-己二醇:通過羧酸還原酶(例如分類在EC 1.2.99.6下),如來自海分枝桿菌(參見Genbank登錄號ACC40567.1,SEQ ID NO:3),恥垢分枝桿菌(參見Genbank登錄號ABK71854.1,SEQ ID NO:4),Segniliparus rugosus(參見Genbank登錄號EFV11917.1,SEQ ID NO:5),馬賽分枝桿菌(參見Genbank登錄號EIV11143.1,SEQ ID NO:6),或Segniliparus rotundus(參見Genbank登錄號ADG98140.1,SEQ ID NO:7)(例如與例如由來自枯草芽孢桿菌的sfp基因或來自諾卡氏菌屬的npt基因編碼的磷酸泛酰巰基乙胺基轉移酶增強劑組合),或者GriC&GriD的基因產物將6-羥基己酸轉化為6-羥基己醛;隨后通過醇脫氫酶(例如分類在EC 1.1.1.-下如EC 1.1.1.1,EC 1.1.1.2,EC 1.1.1.21或EC 1.1.1.184),如由YMR318C或YqhD編碼或具有GenBank登錄號CAA81612.1的蛋白將6-羥基己醛轉化為1,6己二醇(Liu等,Microbiology,2009,155,2078-2085)。見圖8。
培養策略
在一些實施方案中,使用厭氧,需氧或微需氧培養條件在重組宿主中生物合成一種或多種C6結構單元。在一些實施方案中,培養策略需要營養限制,例如氮,磷酸鹽或氧限制。
在一些實施方案中,可以采用使用例如陶瓷膜的細胞保留策略來實現并維持補料分批或連續發酵期間的高細胞密度。
在一些實施方案中,在一種或多種C6結構單元的合成中對發酵補料的主要碳源可以源自生物或非生物原料。
在一些實施方案中,生物原料可以是或可以源自單糖,二糖,木質纖維素,半纖維素,纖維素,木質素,乙酰丙酸和甲酸,甘油三酯,甘油,脂肪酸,農業廢物,濃縮的酒糟,或城市廢物。
已經在幾種微生物,如大腸桿菌、鉤蟲貪銅菌、食油假單胞菌、惡臭假單胞菌和解脂耶羅維亞酵母中證明了對來自生物柴油生產的粗制甘油的有效分解代謝(Lee等,Appl.Biochem.Biotechnol.,2012,166:1801-1813;Yang等,Biotechnology for Biofuels,2012,5:13;Meijnen等,Appl.Microbiol.Biotechnol.,2011,90:885-893)。
已經在幾種生物體,如鉤蟲貪銅菌和惡臭假單胞菌中證明了在經由前體丙酰基-CoA合成3-羥基戊酸中對木質纖維素衍生的乙酰丙酸的有效分解代謝(Jaremko and Yu,Journal of Biotechnology,2011,155,2011,293-298;Martin and Prather,Journal of Biotechnology,2009,139,61-67)。
已經在幾種微生物,如惡臭假單胞菌、鉤蟲貪銅菌中證明了對木質素衍生的芳香族化合物諸如苯甲酸類似物的有效分解代謝(Bugg等,Current Opinion in Biotechnology,2011,22,394-400;Pérez-Pantoja等,FEMS Microbiol.Rev.,2008,32,736-794)。
已經在幾種微生物,包括解脂耶羅維亞酵母中證明了對農業廢物,如橄欖磨坊廢水的有效利用(Papanikolaou等,Bioresour.Technol.,2008,99(7):2419–2428)。
已經對幾種微生物,如大腸桿菌、谷氨酸棒狀桿菌和德氏乳桿菌和乳酸乳球菌證明了可發酵糖類,如源自纖維素、半纖維素、甘蔗和甜菜糖蜜、木薯、玉米和其它農業來源的單糖和二糖的有效利用(參見例如Hermann等,Journal of Biotechnology,2003,104,155-172;Wee等,Food Technol.Biotechnol.,2006,44(2),163-172;Ohashi等,Journal of Bioscience and Bioengineering,1999,87(5),647–654)。
已經對鉤蟲貪銅菌證明了源自多種農業木質纖維素來源的糠醛的有效利用(Li等,Biodegradation,2011,22:1215-1225)。
在一些實施方案中,非生物原料可以是或者可以源自天然氣、合成氣、CO2/H2、甲醇,乙醇,苯甲酸甲酯,非揮發性殘留物(NVR)或來自環己烷氧化過程的堿洗液(caustic wash)廢物流,或對苯二甲酸/間苯二甲酸混合物廢物流。
已經對甲基營養型酵母巴斯德畢赤酵母證明了甲醇的有效分解代謝。
已經對克氏梭菌證明了乙醇的有效分解代謝(Seedorf等,Proc.Natl.Acad.Sci.USA,2008,105(6)2128-2133)。
已經對鉤蟲貪銅菌證明了CO2和H2(其可以源自天然氣和其它化學和石油化學來源)的有效分解代謝(Prybylski等,Energy,Sustainability and Society,2012,2:11)。
已經對多種微生物,如楊氏梭菌和自產乙醇梭菌(Clostridium autoethanogenum)證明了合成氣的有效分解代謝(等,Applied and Environmental Microbiology,2011,77(15):5467-5475)。
已經對多種微生物,如食酸代爾夫特菌和鉤蟲貪銅菌證明了來自環己烷過程的非揮發性殘留物廢物流的有效分解代謝(Ramsay等,Applied and Environmental Microbiology,1986,52(1):152-156)。
在一些實施方案中,宿主微生物可以是原核生物。例如,原核生物可以是來自以下的細菌:埃希氏菌屬如大腸桿菌;梭菌屬如楊氏梭菌、自產乙醇梭菌或者克魯佛梭菌;棒狀桿菌屬如谷氨酸棒狀桿菌;貪銅菌屬如鉤蟲貪銅菌或者耐金屬貪銅菌;假單胞菌屬如熒光假單胞菌、惡臭假單胞菌或者食油假單胞菌;代爾夫特菌屬如食酸代爾夫特菌;芽孢桿菌屬如枯草芽胞桿菌;乳桿菌屬如德氏乳桿菌;或者乳球菌屬如乳酸乳球菌。此類原核生物也可以是構建能夠生成一種或多種C6結構單元的本文中描述的重組宿主細胞的基因來源。
在一些實施方案中,宿主微生物可以是真核生物。例如,真核生物可以是絲狀真菌,例如來自曲霉屬如黑曲霉的。或者,真核生物可以是酵母,例如來自酵母屬如釀酒酵母;來自畢赤氏酵屬如巴斯德畢赤酵母;來自耶羅維亞酵母屬如解脂耶羅維亞酵母;來自伊薩酵母屬如東方伊薩酵母;來自德巴利酵母屬如漢遜德巴利酵母;來自Arxula屬如Arxula adenoinivorans;或者來自克魯維酵母菌屬如乳酸克魯維酵母菌。此類真核生物也可以是構建能夠生成一種或多種C6結構單元的本文中描述的重組宿主細胞的基因來源。
代謝工程
本申請提供方法,其涉及少于對上述所有途徑描述的所有的步驟。此類方法可以涉及例如此類步驟中的一,二,三,四,五,六,七,八,九,十,十一,十二或者更多個。在此類方法包括少于所有步驟的情況下,第一個,并且在一些實施方案中唯一的,步驟可為所列步驟中的任何步驟。
此外,本文中描述的重組宿主可以包括上述酶中的任何組合,使得所述步驟中的一個或者多個,例如此類步驟中的一,二,三,四,五,六,七,八,九,十或者更多個可以在重組宿主內實施。本申請提供了所列出的任何屬和種的宿主細胞,并且經遺傳工程化以表達一種或多種(例如,二,三,四,五,六,七,八,九,10,11,12或更多種)本文所述的任何酶的重組形式。因此,例如,宿主細胞可以含有編碼酶的外源核酸,所述酶催化本文所述的任何途徑的一個或多個步驟。
另外,本申請認識到,在酶已經被描述為接受CoA活化的底物的情況下,存在與[acp]結合底物相關的類似酶活性,其不一定為相同的酶種類。
此外,本文件認識到,在酶已經被描述為接受底物的(R)-對映異構體的情況下,存在與底物的(S)-對映異構體相關的類似酶活性,其不一定為相同的酶種類。
本申請還認識到,在已經顯示酶接受特定輔因子如NADPH或者共底物如乙酰基-CoA的情況下,許多酶在催化特定酶活性中在接受大量不同輔因子或者共底物方面是泛宿主性的(promiscuous)。此外,本申請認識到,在酶對例如特定輔助因子如NADH具有高特異性的情況下,對輔助因子NADPH具有高特異性的具有相似或者相同活性的酶可為不同的酶種類。
在一些實施方案中,本文所述的途徑中的酶是經由非直接的酶工程或者合理的酶設計方法的結果,目的在于改善活性、改善特異性、降低反饋抑制、降低阻抑(repression)、改善酶溶解度、改變立體特異性,或者改變輔助因子特異性。
在一些實施方案中,可以將本文概述的途徑中的酶經由附加型或者染色體整合方法基因給予(即,通過在宿主生物體中具有多個拷貝的基因來過表達)至所得的遺傳修飾的生物體中。
在一些實施方案中,使用基因組級別(genome-scale)系統生物學技術如通量平衡分析來設計用于將碳流量引導至C6結構單元的基因組級別的弱化或者敲除策略。
弱化策略包括但不限于使用轉座子,同源重組(雙交叉方法),誘變,酶抑制劑和RNAi干擾。
在一些實施方案中,使用通量組(fluxomic)、代謝物組(metabolomic)和轉錄物組(transcriptomal)數據來告知或者支持基因組級別的系統生物學技術,由此在將碳流量導向C6結構單元中設計基因組級別的弱化或者敲除策略中。
在一些實施方案中,通過在選擇性環境中連續培養,改善了宿主微生物對高濃度C6結構單元的耐受性。
在一些實施方案中,宿主微生物的生物化學網絡被弱化或增強以(1)保證乙酰-CoA和丙二酰-CoA的細胞內可用度,(2)創造NADPH不平衡,其僅可以通過C6結構單元的形成來平衡,(3)阻止通向和包括一種或多種C6結構單元的中心代謝物或中心前體的降解,和/或(4)保證從細胞的高效流出。
在一些要求乙酰-CoA的細胞內可用度用于C6結構單元的合成的實施方案中,可以弱化產生乙酸的內源性磷酸轉乙酰酶,例如pta(Shen等,Appl.Environ.Microbiol.,2011,77(9):2905–2915)。
在一些要求乙酰-CoA的細胞內可用度用于C6結構單元的合成的實施方案中,可以弱化乙酸合成途徑中編碼乙酸激酶的內源性基因,例如ack。
在一些要求乙酰-CoA和NADH的細胞內可用度用于C6結構單元的合成的實施方案中,可以弱化編碼催化丙酮酸降解為乳酸的酶的內源性基因,例如由ldhA編碼的乳酸脫氫酶(Shen等,Appl.Environ.Microbiol.,2011,77(9),2905-2915)。
在一些要求乙酰-CoA的細胞內可用度用于C6結構單元的合成的實施方案中,可以弱化編碼催化磷酸烯醇丙酮酸降解為琥珀酸的酶的內源基因,例如frdBC(見Shen等,2011,見上文)。
在一些要求乙酰-CoA的細胞內可用度用于C6結構單元的合成的實施方案中,可以弱化編碼催化乙酰-CoA降解為乙醇的酶的內源性基因,例如由adhE編碼的醇脫氫酶(Shen等,2011,見上文)。
在一些要求乙酰-CoA的細胞內可用度用于C6結構單元的合成的實施方案中,可以在微生物中過表達重組乙酰-CoA合成酶,如acs的基因產物(Satoh等,J.Bioscience and Bioengineering,2003,95(4):335–341)。
在一些實施方案中,通過弱化內源性葡萄糖-6-磷酸異構酶(EC 5.3.1.9),可以將碳通量導入戊糖磷酸循環中。
在一些實施方案中,在C6結構單元合成中途徑需要過量的NADPH輔因子的情況下,可以在宿主生物體中過表達編碼吡啶核苷酸轉氫酶的基因例如UdhA(Brigham等,Advanced Biofuels and Bioproducts,2012,Chapter 39,1065-1090)。
在一些實施方案中,在C6結構單元合成中途徑需要過量的NADPH輔因子的情況下,可以在宿主生物體中過表達重組甘油醛-3-磷酸-脫氫酶基因,例如GapN(Brigham等,2012,見上文)。
在一些實施方案中,在C6結構單元合成中途徑需要過量的NADPH輔因子的情況下,可以在宿主生物體中過表達重組蘋果酸酶基因,如maeA或maeB(Brigham等,2012,見上文)。
在一些實施方案中,在C6結構單元合成中途徑需要過量的NADPH輔因子的情況下,可以在宿主生物體中過表達重組葡萄糖-6-磷酸脫氫酶基因,例如zwf(Lim等,Journal of Bioscience and Bioengineering,2002,93(6),543-549)。
在一些實施方案中,在C6結構單元合成中途徑需要過量的NADPH輔因子的情況下,可以在宿主生物體中過表達重組果糖1,6-二磷酸酶基因,例如fbp(Becker et al.,Journal of Biotechnology,2007,132,99-109)。
在一些實施方案中,在C6結構單元合成中途徑需要過量的NADPH輔因子的情況下,可以弱化編碼磷酸丙糖異構酶(EC 5.3.1.1)的內源基因。
在一些實施方案中,在C6結構單元合成中途徑需要過量的NADPH輔因子的情況下,可以在宿主生物體中過表達重組葡萄糖脫氫酶,如gdh的基因產物(Satoh等,2003,見上文)。
在一些實施方案中,弱化編碼促進NADPH轉化為NADH的酶的內源基因,如可以歸類于EC 1.4.1.2(NADH特異性)和EC 1.4.1.4(NADPH特異性)下的谷氨酸脫氫酶的相互轉化產生的NADH生成循環;或轉氫酶。
在一些實施方案中,可以弱化編碼使用NADH和NADPH兩者作為輔因子的谷氨酸脫氫酶(EC 1.4.1.3)的內源基因。
在一些實施方案中,膜結合的烷烴1-單加氧酶通過將P450錨定到內質網的N末端區域的截短而增溶(Scheller等,J.Biol.Chem.,1994,269(17),12779-12783)。
在一些使用天然積累聚羥基鏈烷酸酯的宿主的實施方案中,可以在宿主菌株中弱化編碼聚合物合酶的內源基因。
在使用天然積累脂質體的宿主的一些實施方案中,可以在宿主菌株中弱化編碼合酶的相關基因。
在一些實施方案中,弱化降解導致并包括C6結構單元的中心代謝物和中心前體的β-氧化酶。
在一些實施方案中,弱化編碼通過CoA酯化活化C6結構單元的酶的內源基因,如CoA連接酶。
在一些實施方案中,通過遺傳工程化改造對細胞膜的結構修飾或者提高C6結構單元的任何關聯轉運蛋白活性增強或放大C6結構單元穿過細胞膜到胞外介質的流出。
六亞甲基二胺的流出可以通過過表達廣泛底物范圍多藥物轉運體來提高或放大,如來自枯草芽孢桿菌的Blt(Woolridge等,1997,J.Biol.Chem.,272(14):8864–8866);來自大腸桿菌的AcrB和AcrD(Elkins&Nikaido,2002,J.Bacteriol.,184(23),6490–6499)或來自金黃色葡萄球菌的NorA(Ng等,1994,Antimicrob Agents Chemother,38(6),1345–1355)或來自枯草芽孢桿菌的Bmr(Neyfakh,1992,Antimicrob Agents Chemother,36(2),484–485)。
6-氨基己酸和六亞甲基二胺的流出可以通過過表達溶質轉運體來提高或放大,如來自于谷氨酸棒狀桿菌的lysE轉運體(Bellmann等,2001,Microbiology,147,1765–1774)。
己二酸的流出可以通過過表達二羧酸轉運體來提高或放大,如來自于谷氨酸棒狀桿菌的SucE轉運體(Huhn等,Appl.Microbiol.&Biotech.,89(2),327–335)。
使用重組宿主生產C6結構單元
通常,可以通過提供宿主微生物并且用含有如上文描述的合適碳源的培養基培養提供的微生物生成一種或多種C6結構單元。一般地,培養基和/或培養條件可以使得微生物生長至足夠的密度,并且有效生成C6結構單元。對于大規模生產工藝,可以使用任何方法,諸如那些在別處描述的(Manual of Industrial Microbiology and Biotechnology,2nd Edition,Editors:A.L.Demain and J.E.Davies,ASM Press;and Principles of Fermentation Technology,P.F.Stanbury and A.Whitaker,Pergamon)。簡言之,用特定的微生物接種含有合適培養基的大罐(例如,100加侖,200加侖,500加侖,或更大的罐)。在接種后,溫育微生物以容許生成生物量。一旦達到期望的生物量,可以將含有微生物的培養液轉移到第二個罐。此第二個罐可以是任何大小。例如,第二個罐可以比/與第一個罐大、小或相同大小。通常,第二個罐大于第一個,從而可以對來自第一個罐的培養液添加額外的培養基。另外,此第二個罐內的培養基可以與第一個罐中使用的培養基相同或不同。
一旦轉移,可以溫育微生物以容許生成C6結構單元。一旦生成,可以使用任何方法來分離C6結構單元。例如,可以經由吸附方法從發酵液選擇性回收C6結構單元。在己二酸和6-氨基己酸的情況中,所得的洗脫液可以經由蒸發進一步濃縮,經由蒸發和/或冷卻結晶來結晶,并且經由離心回收晶體。在六亞甲基二胺和1,6-己二醇的情況中,可以采用蒸餾來實現期望的產物純度。
本發明在以下實施例中進一步描述,所述實施例不限制權利要求書中描述的本發明范圍。
實施例
實施例1
在形成6-羥基己酸中使用己酸作為底物的CYP153單加氧酶的酶活性
將編碼HIS標簽的核苷酸序列添加到單胞菌屬物種JS666,分支桿菌屬物種HXN-1500和南非分支桿菌基因,其分別編碼(1)單加氧酶(SEQ ID NO:16-18),(2)相關的鐵氧還原蛋白還原酶配偶體(SEQ ID NO:19-20)和物種的鐵氧還原蛋白(SEQ ID NO:21-22)。對于南非分枝桿菌單加氧酶,使用分支桿菌屬物種HXN-1500氧化還原酶和鐵氧還原蛋白配偶體。將編碼蛋白配偶體的三種經修飾的核酸序列克隆到pgBlue表達載體中,在雜交體pTac啟動子下。將每種表達載體轉化到BL21[DE3]大腸桿菌宿主中。將每種所得重組大腸桿菌菌株在含有50mL LB培養基和抗生素選擇壓力的500mL搖瓶培養物中在37℃下培養。使用1mM IPTG在28℃下誘導每種培養物24小時。
通過離心收集來自每個誘導的搖瓶培養物的團粒。將每個團粒重懸浮,并使用Y-perTM溶液(ThermoScientific,Rockford,IL)在室溫下通透細胞20分鐘。將經通透的細胞在Y-perTM溶液中保持在0℃。
在由最終濃度為25mM磷酸鉀緩沖液(pH=7.8),1.7mM MgSO4,,2.5mM NADPH和30mM己酸鹽組成的緩沖液中進行酶活性測定。通過將懸浮在Y-perTM溶液中的固定質量的濕細胞重量的通透細胞加入到含有庚酸鹽的測定緩沖液中起始每個酶活性測定反應,然后在加熱塊振蕩器中在28℃下以1400rpm振蕩溫育24小時。通過LC-MS定量7-羥基庚酸的形成。
如針對空載體對照所確認的,SEQ ID NO 16-18的單加氧酶基因產物連同還原酶和鐵氧還原蛋白配偶體,接受己酸作為底物(見圖20)并合成6-羥基己酸作為反應產物。
實施例2
使用己二酸半醛作為底物并且形成6-氨基己酸的ω-轉氨酶的酶活性
將編碼His標簽的核苷酸序列添加至分別編碼SEQ ID NO:8、9、10、11和13的ω-轉氨酶的來自紫色色桿菌、銅綠假單胞菌、丁香假單胞菌、球形紅桿菌、和河流弧菌的基因(見圖12),使得可以產生N-末端有HIS標簽的ω-轉氨酶。將每種所得的經修飾基因克隆到pET21a表達載體中在T7啟動子的控制下,并且將每種表達載體轉化入BL21[DE3]大腸桿菌宿主中。將所得重組大腸桿菌菌株在含有50mL LB培養基和抗生素選擇壓力的250mL搖瓶培養物中在37℃下培養,以230rpm振蕩。使用1mM IPTG于16℃誘導每種培養物過夜。
經由離心收獲來自每個經誘導的搖瓶培養物的團粒。重懸每個團粒粒并經由超聲處理裂解。經由離心分開細胞碎片與上清液,并且立即在酶活性測定法中使用無細胞提取物。
在緩沖液中實施逆向(即6-氨基己酸至己二酸半醛)的酶活性測定法,所述緩沖液由終濃度為50mM HEPES緩沖液(pH=7.5)、10mM 6-氨基己酸、10mM丙酮酸和100μM吡哆(pyridoxyl)5’磷酸鹽組成。通過添加ω-轉氨酶基因產物或空載體對照的無細胞提取物到含有6-氨基己酸的測定緩沖液啟動每個酶活性測定反應,并在以250rpm振蕩的情況下于25℃溫育24小時。經由RP-HPLC量化從丙酮酸形成L-丙氨酸。
每種沒有6-氨基己酸的僅酶對照表明丙酮酸至L-丙氨酸的低基線轉化。參見圖14。如相對于空載體對照所確認的,SEQ ID NO 8、SEQ ID NO 10、SEQ ID NO 11和SEQ ID NO 13的基因產物接受6-氨基己酸作為底物。見圖15。
對SEQ ID NO 8、SEQ ID NO 9、SEQ ID NO 10、SEQ ID NO 11和SEQ ID NO 13的轉氨酶確認正向(即己二酸半醛至6-氨基己酸)的酶反應。在緩沖液中實施酶活性測定法,所述緩沖液由終濃度50mM HEPES緩沖液(pH=7.5)、10mM己二酸半醛、10mM L-丙氨酸和100μM吡哆5’磷酸鹽組成。通過添加ω-轉氨酶基因產物或空載體對照的無細胞提取物到含有己二酸半醛的測定緩沖液啟動每個酶活性測定反應,并在以250rpm振蕩的情況下于25℃溫育4小時。經由RP-HPLC量化丙酮酸的形成。
如相對于空載體對照所確認的,SEQ ID NO 8、SEQ ID NO 9、SEQ ID NO 10、SEQ ID NO 11和SEQ ID NO 13的轉氨酶接受己二酸半醛作為底物。參 見圖16。確認了ω-轉氨酶活性的可逆性,證明了SEQ ID NO 8、SEQ ID NO 9、SEQ ID NO 10、SEQ ID NO 11和SEQ ID NO 13的ω-轉氨酶接受己二酸半醛作為底物,并且合成6-氨基己酸作為反應產物。
實施例3
羧酸還原酶使用己二酸作為底物并形成己二酸半醛的酶活性
將編碼HIS標簽的核苷酸序列添加至分別編碼SEQ ID NO:5和7的羧酸還原酶的來自Segniliparus rugosus和Segniliparus rotundus的基因(參見圖21),使得可以生成N-末端有HIS標簽的羧酸還原酶。將每種經修飾的基因與編碼有HIS標簽的來自枯草芽孢桿菌的磷酸泛酰巰基乙胺基轉移酶的sfp基因一起克隆入pET Duet表達載體中,兩者都在T7啟動子下。將每種表達載體轉化入BL21[DE3]大腸桿菌宿主,并且于37℃在含有50mL LB培養基和抗生素選擇壓力的250mL搖瓶培養物中在以230rpm振蕩的情況中培養所得的重組大腸桿菌菌株。使用自誘導培養基于37℃誘導每種培養物過夜。
經由離心收獲來自每個經誘導的搖瓶培養物的團粒。重懸每個團粒和經由超聲處理裂解,并經由離心分離細胞碎片與上清液。使用Ni-親和層析從上清液中純化羧酸還原酶和磷酸泛酰巰基乙胺基轉移酶,以10倍稀釋入50mM HEPES緩沖液(pH=7.5)中,并經由超濾濃縮。
在緩沖液中一式三份實施酶活性測定法(即從己二酸至己二酸半醛),所述緩沖液由終濃度50mM HEPES緩沖液(pH=7.5)、2mM己二酸、10mM MgCl2、1mM ATP和1mM NADPH組成。通過添加純化的羧酸還原酶和磷酸泛酰巰基乙胺基轉移酶基因產物或空載體對照至含有己二酸的測定緩沖液起始每個酶活性測定反應,然后于室溫溫育20分鐘。通過于340nm的吸光度監測NADPH的消耗。每種沒有己二酸的僅酶對照表明NADPH的低基線消耗。見圖9。
如相對于空載體對照所確認的,SEQ ID NO 5和SEQ ID NO 7的基因產物,由sfp的基因產物增強,接受己二酸作為底物(參見圖10),并且合成己二酸半醛。
實施例4
羧酸還原酶使用6-羥基己酸作為底物并形成6-羥基己醛的酶活性
將編碼His標簽的核苷酸序列添加至分別編碼SEQ ID NO:3-7的羧酸還原酶的來自海分枝桿菌、恥垢分枝桿菌、恥垢分枝桿菌、Segniliparus rugosus、馬賽分枝桿菌和Segniliparus rotundus的基因(見圖21),使得可以生成N-末端有HIS標簽的羧酸還原酶。將每種經修飾的基因與編碼有His標簽的來自枯草芽孢桿菌的磷酸泛酰巰基乙胺基轉移酶的sfp基因一起克隆入pET Duet表達載體中,兩者都在T7啟動子下。將每種表達載體轉化入BL21[DE3]大腸桿菌宿主中。于37℃在含有50mL LB培養基和抗生素選擇壓力的250mL搖瓶培養物中在以230rpm振蕩的情況中培養所得的每種重組大腸桿菌菌株。使用自誘導培養基于37℃誘導每種培養物過夜。
經由離心收獲來自每個經誘導的搖瓶培養物的團粒。重懸每個團粒,并經由超聲處理裂解。經由離心分離細胞碎片與上清液。使用Ni-親和層析從上清液中純化羧酸還原酶和磷酸泛酰巰基乙胺基轉移酶,以10倍稀釋入50mM HEPES緩沖液(pH=7.5)中,并經由超濾濃縮。
在緩沖液中一式三份實施酶活性測定法(即從6-羥基己酸至6-羥基己醛),所述緩沖液由終濃度50mM HEPES緩沖液(pH=7.5)、2mM 6-羥基己醛、10mM MgCl2、1mM ATP和1mM NADPH組成。通過添加純化的羧酸還原酶和磷酸泛酰巰基乙胺基轉移酶或空載體對照至含有6-羥基己酸的測定緩沖液啟動每個酶活性測定反應,然后于室溫溫育20分鐘。通過于340nm的吸光度監測NADPH的消耗。每種沒有6-羥基己酸的僅酶對照表明NADPH的低基線消耗。參見圖9。
如相對于空載體對照確認的,SEQ ID NO 3-7的基因產物,由sfp的基因產物增強,接受6-羥基己酸作為底物(參見圖11),并合成6-羥基己醛。
實施例5
ω-轉氨酶對于6-氨基己醇,形成6-氧代己醇的酶活性
將編碼N-末端His標簽的核苷酸序列添加至分別編碼SEQ ID NO:8-13的ω-轉氨酶的來自紫色色桿菌、銅綠假單胞菌、丁香假單胞菌、球形紅桿菌、大腸桿菌和河流弧菌基因(參見圖21),使得可以產生N-末端有HIS標簽的ω-轉氨酶。將經修飾基因克隆至pET21a表達載體中,在T7啟動子下。將每種表達載體轉化入BL21[DE3]大腸桿菌宿主中。于37℃在含有50mL LB培養基和抗生素選擇壓力的250mL搖瓶培養物中在以230rpm振蕩的情況中培養每種所得的重組大腸桿菌菌株。使用1mM IPTG于16℃誘導每種培養物過夜。
經由離心收獲來自每種經誘導的搖瓶培養物的團粒。重懸每個團粒,并經由超聲處理裂解。經由離心分離細胞碎片與上清液,并且立即在酶活性測定法中使用無細胞提取物。
在緩沖液中實施逆向(即6-氨基己醇至6-氧代己醇)的酶活性測定法,所述緩沖液由終濃度50mM HEPES緩沖液(pH=7.5)、10mM 6-氨基己醇、10 mM丙酮酸和100μM吡哆5’磷酸鹽組成。通過添加ω-轉氨酶基因產物或空載體對照的無細胞提取物到含有6-氨基己醇的測定緩沖液啟動每個酶活性測定反應,并在以250rpm振蕩的情況下于25℃溫育4小時。經由RP-HPLC量化L-丙氨酸形成。
每個沒有6-氨基己醇的僅酶對照具有丙酮酸至L-丙氨酸的低基線轉化。參見圖14。
如相對于空載體對照所確認的,SEQ ID NO 8-13的基因產物接受6-氨基己醇作為底物,并合成6-氧代己醇作為反應產物。鑒于ω-轉氨酶活性的可逆性(參見實施例2),可以推斷SEQ ID 8-13的基因產物接受6-氨基己醇作為底物,并形成6-氧代己醇。
實施例6
ω-轉氨酶使用六亞甲基二胺作為底物并形成6-氨基己醛的酶活性
將編碼N-末端His標簽的核苷酸序列添加至分別編碼SEQ ID NO:8-13的ω-轉氨酶的紫色色桿菌、銅綠假單胞菌、丁香假單胞菌、球形紅桿菌、大腸桿菌、和河流弧菌基因(參見圖21),使得可以產生N-末端有HIS標簽的ω-轉氨酶。將經修飾的基因克隆至pET21a表達載體中,在T7啟動子下。將每種表達載體轉化入BL21[DE3]大腸桿菌宿主中。于37℃在含有50mL LB培養基和抗生素選擇壓力的250mL搖瓶培養物中在以230rpm振蕩的情況中培養每種所得的重組大腸桿菌菌株。使用1mM IPTG于16℃誘導每種培養物過夜。
經由離心收獲來自每個經誘導的搖瓶培養物的團粒。重懸每個團粒,并經由超聲處理裂解。經由離心分離細胞碎片與上清液,并且立即在酶活性測定法中使用無細胞提取物。
在緩沖液中實施逆向(即六亞甲基二胺至6-氨基己醛)的酶活性測定法,所述緩沖液由終濃度50mM HEPES緩沖液(pH=7.5)、10mM六亞甲基二胺、10mM丙酮酸和100μM吡哆5’磷酸鹽組成。通過添加ω-轉氨酶基因產物或空載體對照的無細胞提取物到含有六亞甲基二胺的測定緩沖液起始每個酶活性測定反應,并在以250rpm振蕩的情況下于25℃溫育4小時。經由RP-HPLC量化L-丙氨酸的形成。
每個沒有六亞甲基二胺的僅酶對照具有丙酮酸至L-丙氨酸的低基線轉化。參見圖14。
如相對于空載體對照所確認的,SEQ ID NO 8-13的基因產物接受六亞甲基二胺作為底物(參見圖17),并合成6-氨基己醛作為反應產物。鑒于ω-轉氨酶活性的可逆性(參見實施例2),可以推斷SEQ ID 8–13的基因產物接受6-氨基己醛作為底物,并形成六亞甲基二胺。
實施例7
羧酸還原酶對于N6-乙酰-6-氨基己酸,形成N6-乙酰-6-氨基己醛的酶活性
一式三份在緩沖液中測定用于將N6-乙酰-6-氨基己酸轉化成N6-乙酰-6-氨基己醛的N末端有His標簽的SEQ ID NO:5-7的羧酸還原酶的活性(參見實施例4,和圖21),所述緩沖液由終濃度50mM HEPES緩沖液(pH=7.5)、2mM N6-乙酰-6-氨基己酸、10mM MgCl2、1mM ATP、和1mM NADPH組成。通過添加純化的羧酸還原酶和磷酸泛酰巰基乙胺基轉移酶或空載體對照至含有N6-乙酰-6-氨基己酸的測定緩沖液起始測定,然后于室溫溫育20分鐘。通過于340nm的吸光度監測NADPH的消耗。每種沒有N6-乙酰-6-氨基己酸的僅酶對照表明NADPH的低基線消耗。參見圖9。
如相對于空載體對照所確認的,SEQ ID NO 5-7的基因產物,由sfp的基因產物增強,接受N6-乙酰-6-氨基己酸作為底物(參見圖12),并合成N6-乙酰-6-氨基己醛。
實施例8
ω-轉氨酶使用N6-乙酰-1,6-二氨基己烷并形成N6-乙酰-6-氨基己醛的酶活性
在緩沖液中測定用于將N6-乙酰-1,6-二氨基己烷轉化成N6-乙酰-6-氨基己醛的N末端有His標簽的SEQ ID NO:8-13的ω-轉氨酶的活性(參見實施例6,及圖21),所述緩沖液由終濃度50mM HEPES緩沖液(pH=7.5)、10mM N6-乙酰-1,6-二氨基己烷、10mM丙酮酸和100μM吡哆5’磷酸鹽組成。通過添加ω-轉氨酶或空載體對照的無細胞提取物至含有N6-乙酰-1,6-二氨基己烷的測定緩沖液起始每個酶活性測定反應,然后在250rpm振蕩的情況下于25℃溫育4h。經由RP-HPLC量化L-丙氨酸的形成。
每個沒有N6-乙酰-1,6-二氨基己烷的僅酶對照表明丙酮酸至L-丙氨酸的低基線轉化。參見圖14。
如相對于空載體對照所確認的,SEQ ID NO:8–13的基因產物接受N6-乙酰-1,6-二氨基己烷作為底物(參見圖18),并合成N6-乙酰-6-氨基己醛作為反應產物。
鑒于ω-轉氨酶活性的可逆性(參見實施例2),SEQ ID NO:8-13的基因產物接受N6-乙酰-6-氨基己醛作為底物,形成N6-乙酰-1,6-二氨基己烷。
實施例9:羧酸還原酶使用己二酸半醛作為底物并形成己二醛的酶活性
使用己二酸半醛作為底物測定N-末端有His標簽的SEQ ID NO 7的羧酸還原酶(參見實施例4和圖21)。一式三份在緩沖液中實施酶活性測定法,所述緩沖液由終濃度50mM HEPES緩沖液(pH=7.5)、2mM己二酸半醛、10mM MgCl2、1mM ATP和1mM NADPH組成。通過添加純化的羧酸還原酶和磷酸泛酰巰基乙胺基轉移酶或空載體對照至含有己二酸半醛的測定緩沖液起始測定,然后于室溫溫育20分鐘。通過340nm的吸光度監測NADPH的消耗。每種沒有己二酸半醛的僅酶對照表明NADPH的低基線消耗。參見圖9。
如相對于空載體對照所確認的,SEQ ID NO 7的基因產物,由sfp的基因產物增強,接受己二酸半醛作為底物(參見圖13),并合成己二醛。
其它實施例
應當理解,雖然已經結合其詳細描述描述了本發明,但是前面的描述旨在說明而不是限制由所附權利要求的范圍限定的本發明的范圍。其他方面,優點和修改在所附權利要求的范圍內。
序列表
<110> 英威達技術有限責任公司.
<120> 通過氧化切割從長鏈脂肪酸生產6-碳化學物的方法
<130> 35643-0030WO1
<150> US 61/992,794
<151> 2014-05-13
<160> 23
<170> FastSEQ for Windows Version 4.0
<210> 1
<211> 246
<212> PRT
<213> 短乳桿菌
<400> 1
Met Ala Ala Asn Glu Phe Ser Glu Thr His Arg Val Val Tyr Tyr Glu
1 5 10 15
Ala Asp Asp Thr Gly Gln Leu Thr Leu Ala Met Leu Ile Asn Leu Phe
20 25 30
Val Leu Val Ser Glu Asp Gln Asn Asp Ala Leu Gly Leu Ser Thr Ala
35 40 45
Phe Val Gln Ser His Gly Val Gly Trp Val Val Thr Gln Tyr His Leu
50 55 60
His Ile Asp Glu Leu Pro Arg Thr Gly Ala Gln Val Thr Ile Lys Thr
65 70 75 80
Arg Ala Thr Ala Tyr Asn Arg Tyr Phe Ala Tyr Arg Glu Tyr Trp Leu
85 90 95
Leu Asp Asp Ala Gly Gln Val Leu Ala Tyr Gly Glu Gly Ile Trp Val
100 105 110
Thr Met Ser Tyr Ala Thr Arg Lys Ile Thr Thr Ile Pro Ala Glu Val
115 120 125
Met Ala Pro Tyr His Ser Glu Glu Gln Thr Arg Leu Pro Arg Leu Pro
130 135 140
Arg Pro Asp His Phe Asp Glu Ala Val Asn Gln Thr Leu Lys Pro Tyr
145 150 155 160
Thr Val Arg Tyr Phe Asp Ile Asp Gly Asn Gly His Val Asn Asn Ala
165 170 175
His Tyr Phe Asp Trp Met Leu Asp Val Leu Pro Ala Thr Phe Leu Arg
180 185 190
Ala His His Pro Thr Asp Val Lys Ile Arg Phe Glu Asn Glu Val Gln
195 200 205
Tyr Gly His Gln Val Thr Ser Glu Leu Ser Gln Ala Ala Ala Leu Thr
210 215 220
Thr Gln His Met Ile Lys Val Gly Asp Leu Thr Ala Val Lys Ala Thr
225 230 235 240
Ile Gln Trp Asp Asn Arg
245
<210> 2
<211> 261
<212> PRT
<213> 植物乳桿菌
<400> 2
Met Ala Thr Leu Gly Ala Asn Ala Ser Leu Tyr Ser Glu Gln His Arg
1 5 10 15
Ile Thr Tyr Tyr Glu Cys Asp Arg Thr Gly Arg Ala Thr Leu Thr Thr
20 25 30
Leu Ile Asp Ile Ala Val Leu Ala Ser Glu Asp Gln Ser Asp Ala Leu
35 40 45
Gly Leu Thr Thr Glu Met Val Gln Ser His Gly Val Gly Trp Val Val
50 55 60
Thr Gln Tyr Ala Ile Asp Ile Thr Arg Met Pro Arg Gln Asp Glu Val
65 70 75 80
Val Thr Ile Ala Val Arg Gly Ser Ala Tyr Asn Pro Tyr Phe Ala Tyr
85 90 95
Arg Glu Phe Trp Ile Arg Asp Ala Asp Gly Gln Gln Leu Ala Tyr Ile
100 105 110
Thr Ser Ile Trp Val Met Met Ser Gln Thr Thr Arg Arg Ile Val Lys
115 120 125
Ile Leu Pro Glu Leu Val Ala Pro Tyr Gln Ser Glu Val Val Lys Arg
130 135 140
Ile Pro Arg Leu Pro Arg Pro Ile Ser Phe Glu Ala Thr Asp Thr Thr
145 150 155 160
Ile Thr Lys Pro Tyr His Val Arg Phe Phe Asp Ile Asp Pro Asn Arg
165 170 175
His Val Asn Asn Ala His Tyr Phe Asp Trp Leu Val Asp Thr Leu Pro
180 185 190
Ala Thr Phe Leu Leu Gln His Asp Leu Val His Val Asp Val Arg Tyr
195 200 205
Glu Asn Glu Val Lys Tyr Gly Gln Thr Val Thr Ala His Ala Asn Ile
210 215 220
Leu Pro Ser Glu Val Ala Asp Gln Val Thr Thr Ser His Leu Ile Glu
225 230 235 240
Val Asp Asp Glu Lys Cys Cys Glu Val Thr Ile Gln Trp Arg Thr Leu
245 250 255
Pro Glu Pro Ile Gln
260
<210> 3
<211> 1174
<212> PRT
<213> 海魚分枝桿菌
<400> 3
Met Ser Pro Ile Thr Arg Glu Glu Arg Leu Glu Arg Arg Ile Gln Asp
1 5 10 15
Leu Tyr Ala Asn Asp Pro Gln Phe Ala Ala Ala Lys Pro Ala Thr Ala
20 25 30
Ile Thr Ala Ala Ile Glu Arg Pro Gly Leu Pro Leu Pro Gln Ile Ile
35 40 45
Glu Thr Val Met Thr Gly Tyr Ala Asp Arg Pro Ala Leu Ala Gln Arg
50 55 60
Ser Val Glu Phe Val Thr Asp Ala Gly Thr Gly His Thr Thr Leu Arg
65 70 75 80
Leu Leu Pro His Phe Glu Thr Ile Ser Tyr Gly Glu Leu Trp Asp Arg
85 90 95
Ile Ser Ala Leu Ala Asp Val Leu Ser Thr Glu Gln Thr Val Lys Pro
100 105 110
Gly Asp Arg Val Cys Leu Leu Gly Phe Asn Ser Val Asp Tyr Ala Thr
115 120 125
Ile Asp Met Thr Leu Ala Arg Leu Gly Ala Val Ala Val Pro Leu Gln
130 135 140
Thr Ser Ala Ala Ile Thr Gln Leu Gln Pro Ile Val Ala Glu Thr Gln
145 150 155 160
Pro Thr Met Ile Ala Ala Ser Val Asp Ala Leu Ala Asp Ala Thr Glu
165 170 175
Leu Ala Leu Ser Gly Gln Thr Ala Thr Arg Val Leu Val Phe Asp His
180 185 190
His Arg Gln Val Asp Ala His Arg Ala Ala Val Glu Ser Ala Arg Glu
195 200 205
Arg Leu Ala Gly Ser Ala Val Val Glu Thr Leu Ala Glu Ala Ile Ala
210 215 220
Arg Gly Asp Val Pro Arg Gly Ala Ser Ala Gly Ser Ala Pro Gly Thr
225 230 235 240
Asp Val Ser Asp Asp Ser Leu Ala Leu Leu Ile Tyr Thr Ser Gly Ser
245 250 255
Thr Gly Ala Pro Lys Gly Ala Met Tyr Pro Arg Arg Asn Val Ala Thr
260 265 270
Phe Trp Arg Lys Arg Thr Trp Phe Glu Gly Gly Tyr Glu Pro Ser Ile
275 280 285
Thr Leu Asn Phe Met Pro Met Ser His Val Met Gly Arg Gln Ile Leu
290 295 300
Tyr Gly Thr Leu Cys Asn Gly Gly Thr Ala Tyr Phe Val Ala Lys Ser
305 310 315 320
Asp Leu Ser Thr Leu Phe Glu Asp Leu Ala Leu Val Arg Pro Thr Glu
325 330 335
Leu Thr Phe Val Pro Arg Val Trp Asp Met Val Phe Asp Glu Phe Gln
340 345 350
Ser Glu Val Asp Arg Arg Leu Val Asp Gly Ala Asp Arg Val Ala Leu
355 360 365
Glu Ala Gln Val Lys Ala Glu Ile Arg Asn Asp Val Leu Gly Gly Arg
370 375 380
Tyr Thr Ser Ala Leu Thr Gly Ser Ala Pro Ile Ser Asp Glu Met Lys
385 390 395 400
Ala Trp Val Glu Glu Leu Leu Asp Met His Leu Val Glu Gly Tyr Gly
405 410 415
Ser Thr Glu Ala Gly Met Ile Leu Ile Asp Gly Ala Ile Arg Arg Pro
420 425 430
Ala Val Leu Asp Tyr Lys Leu Val Asp Val Pro Asp Leu Gly Tyr Phe
435 440 445
Leu Thr Asp Arg Pro His Pro Arg Gly Glu Leu Leu Val Lys Thr Asp
450 455 460
Ser Leu Phe Pro Gly Tyr Tyr Gln Arg Ala Glu Val Thr Ala Asp Val
465 470 475 480
Phe Asp Ala Asp Gly Phe Tyr Arg Thr Gly Asp Ile Met Ala Glu Val
485 490 495
Gly Pro Glu Gln Phe Val Tyr Leu Asp Arg Arg Asn Asn Val Leu Lys
500 505 510
Leu Ser Gln Gly Glu Phe Val Thr Val Ser Lys Leu Glu Ala Val Phe
515 520 525
Gly Asp Ser Pro Leu Val Arg Gln Ile Tyr Ile Tyr Gly Asn Ser Ala
530 535 540
Arg Ala Tyr Leu Leu Ala Val Ile Val Pro Thr Gln Glu Ala Leu Asp
545 550 555 560
Ala Val Pro Val Glu Glu Leu Lys Ala Arg Leu Gly Asp Ser Leu Gln
565 570 575
Glu Val Ala Lys Ala Ala Gly Leu Gln Ser Tyr Glu Ile Pro Arg Asp
580 585 590
Phe Ile Ile Glu Thr Thr Pro Trp Thr Leu Glu Asn Gly Leu Leu Thr
595 600 605
Gly Ile Arg Lys Leu Ala Arg Pro Gln Leu Lys Lys His Tyr Gly Glu
610 615 620
Leu Leu Glu Gln Ile Tyr Thr Asp Leu Ala His Gly Gln Ala Asp Glu
625 630 635 640
Leu Arg Ser Leu Arg Gln Ser Gly Ala Asp Ala Pro Val Leu Val Thr
645 650 655
Val Cys Arg Ala Ala Ala Ala Leu Leu Gly Gly Ser Ala Ser Asp Val
660 665 670
Gln Pro Asp Ala His Phe Thr Asp Leu Gly Gly Asp Ser Leu Ser Ala
675 680 685
Leu Ser Phe Thr Asn Leu Leu His Glu Ile Phe Asp Ile Glu Val Pro
690 695 700
Val Gly Val Ile Val Ser Pro Ala Asn Asp Leu Gln Ala Leu Ala Asp
705 710 715 720
Tyr Val Glu Ala Ala Arg Lys Pro Gly Ser Ser Arg Pro Thr Phe Ala
725 730 735
Ser Val His Gly Ala Ser Asn Gly Gln Val Thr Glu Val His Ala Gly
740 745 750
Asp Leu Ser Leu Asp Lys Phe Ile Asp Ala Ala Thr Leu Ala Glu Ala
755 760 765
Pro Arg Leu Pro Ala Ala Asn Thr Gln Val Arg Thr Val Leu Leu Thr
770 775 780
Gly Ala Thr Gly Phe Leu Gly Arg Tyr Leu Ala Leu Glu Trp Leu Glu
785 790 795 800
Arg Met Asp Leu Val Asp Gly Lys Leu Ile Cys Leu Val Arg Ala Lys
805 810 815
Ser Asp Thr Glu Ala Arg Ala Arg Leu Asp Lys Thr Phe Asp Ser Gly
820 825 830
Asp Pro Glu Leu Leu Ala His Tyr Arg Ala Leu Ala Gly Asp His Leu
835 840 845
Glu Val Leu Ala Gly Asp Lys Gly Glu Ala Asp Leu Gly Leu Asp Arg
850 855 860
Gln Thr Trp Gln Arg Leu Ala Asp Thr Val Asp Leu Ile Val Asp Pro
865 870 875 880
Ala Ala Leu Val Asn His Val Leu Pro Tyr Ser Gln Leu Phe Gly Pro
885 890 895
Asn Ala Leu Gly Thr Ala Glu Leu Leu Arg Leu Ala Leu Thr Ser Lys
900 905 910
Ile Lys Pro Tyr Ser Tyr Thr Ser Thr Ile Gly Val Ala Asp Gln Ile
915 920 925
Pro Pro Ser Ala Phe Thr Glu Asp Ala Asp Ile Arg Val Ile Ser Ala
930 935 940
Thr Arg Ala Val Asp Asp Ser Tyr Ala Asn Gly Tyr Ser Asn Ser Lys
945 950 955 960
Trp Ala Gly Glu Val Leu Leu Arg Glu Ala His Asp Leu Cys Gly Leu
965 970 975
Pro Val Ala Val Phe Arg Cys Asp Met Ile Leu Ala Asp Thr Thr Trp
980 985 990
Ala Gly Gln Leu Asn Val Pro Asp Met Phe Thr Arg Met Ile Leu Ser
995 1000 1005
Leu Ala Ala Thr Gly Ile Ala Pro Gly Ser Phe Tyr Glu Leu Ala Ala
1010 1015 1020
Asp Gly Ala Arg Gln Arg Ala His Tyr Asp Gly Leu Pro Val Glu Phe
1025 1030 1035 1040
Ile Ala Glu Ala Ile Ser Thr Leu Gly Ala Gln Ser Gln Asp Gly Phe
1045 1050 1055
His Thr Tyr His Val Met Asn Pro Tyr Asp Asp Gly Ile Gly Leu Asp
1060 1065 1070
Glu Phe Val Asp Trp Leu Asn Glu Ser Gly Cys Pro Ile Gln Arg Ile
1075 1080 1085
Ala Asp Tyr Gly Asp Trp Leu Gln Arg Phe Glu Thr Ala Leu Arg Ala
1090 1095 1100
Leu Pro Asp Arg Gln Arg His Ser Ser Leu Leu Pro Leu Leu His Asn
1105 1110 1115 1120
Tyr Arg Gln Pro Glu Arg Pro Val Arg Gly Ser Ile Ala Pro Thr Asp
1125 1130 1135
Arg Phe Arg Ala Ala Val Gln Glu Ala Lys Ile Gly Pro Asp Lys Asp
1140 1145 1150
Ile Pro His Val Gly Ala Pro Ile Ile Val Lys Tyr Val Ser Asp Leu
1155 1160 1165
Arg Leu Leu Gly Leu Leu
1170
<210> 4
<211> 1173
<212> PRT
<213> 恥垢分枝桿菌
<400> 4
Met Thr Ser Asp Val His Asp Ala Thr Asp Gly Val Thr Glu Thr Ala
1 5 10 15
Leu Asp Asp Glu Gln Ser Thr Arg Arg Ile Ala Glu Leu Tyr Ala Thr
20 25 30
Asp Pro Glu Phe Ala Ala Ala Ala Pro Leu Pro Ala Val Val Asp Ala
35 40 45
Ala His Lys Pro Gly Leu Arg Leu Ala Glu Ile Leu Gln Thr Leu Phe
50 55 60
Thr Gly Tyr Gly Asp Arg Pro Ala Leu Gly Tyr Arg Ala Arg Glu Leu
65 70 75 80
Ala Thr Asp Glu Gly Gly Arg Thr Val Thr Arg Leu Leu Pro Arg Phe
85 90 95
Asp Thr Leu Thr Tyr Ala Gln Val Trp Ser Arg Val Gln Ala Val Ala
100 105 110
Ala Ala Leu Arg His Asn Phe Ala Gln Pro Ile Tyr Pro Gly Asp Ala
115 120 125
Val Ala Thr Ile Gly Phe Ala Ser Pro Asp Tyr Leu Thr Leu Asp Leu
130 135 140
Val Cys Ala Tyr Leu Gly Leu Val Ser Val Pro Leu Gln His Asn Ala
145 150 155 160
Pro Val Ser Arg Leu Ala Pro Ile Leu Ala Glu Val Glu Pro Arg Ile
165 170 175
Leu Thr Val Ser Ala Glu Tyr Leu Asp Leu Ala Val Glu Ser Val Arg
180 185 190
Asp Val Asn Ser Val Ser Gln Leu Val Val Phe Asp His His Pro Glu
195 200 205
Val Asp Asp His Arg Asp Ala Leu Ala Arg Ala Arg Glu Gln Leu Ala
210 215 220
Gly Lys Gly Ile Ala Val Thr Thr Leu Asp Ala Ile Ala Asp Glu Gly
225 230 235 240
Ala Gly Leu Pro Ala Glu Pro Ile Tyr Thr Ala Asp His Asp Gln Arg
245 250 255
Leu Ala Met Ile Leu Tyr Thr Ser Gly Ser Thr Gly Ala Pro Lys Gly
260 265 270
Ala Met Tyr Thr Glu Ala Met Val Ala Arg Leu Trp Thr Met Ser Phe
275 280 285
Ile Thr Gly Asp Pro Thr Pro Val Ile Asn Val Asn Phe Met Pro Leu
290 295 300
Asn His Leu Gly Gly Arg Ile Pro Ile Ser Thr Ala Val Gln Asn Gly
305 310 315 320
Gly Thr Ser Tyr Phe Val Pro Glu Ser Asp Met Ser Thr Leu Phe Glu
325 330 335
Asp Leu Ala Leu Val Arg Pro Thr Glu Leu Gly Leu Val Pro Arg Val
340 345 350
Ala Asp Met Leu Tyr Gln His His Leu Ala Thr Val Asp Arg Leu Val
355 360 365
Thr Gln Gly Ala Asp Glu Leu Thr Ala Glu Lys Gln Ala Gly Ala Glu
370 375 380
Leu Arg Glu Gln Val Leu Gly Gly Arg Val Ile Thr Gly Phe Val Ser
385 390 395 400
Thr Ala Pro Leu Ala Ala Glu Met Arg Ala Phe Leu Asp Ile Thr Leu
405 410 415
Gly Ala His Ile Val Asp Gly Tyr Gly Leu Thr Glu Thr Gly Ala Val
420 425 430
Thr Arg Asp Gly Val Ile Val Arg Pro Pro Val Ile Asp Tyr Lys Leu
435 440 445
Ile Asp Val Pro Glu Leu Gly Tyr Phe Ser Thr Asp Lys Pro Tyr Pro
450 455 460
Arg Gly Glu Leu Leu Val Arg Ser Gln Thr Leu Thr Pro Gly Tyr Tyr
465 470 475 480
Lys Arg Pro Glu Val Thr Ala Ser Val Phe Asp Arg Asp Gly Tyr Tyr
485 490 495
His Thr Gly Asp Val Met Ala Glu Thr Ala Pro Asp His Leu Val Tyr
500 505 510
Val Asp Arg Arg Asn Asn Val Leu Lys Leu Ala Gln Gly Glu Phe Val
515 520 525
Ala Val Ala Asn Leu Glu Ala Val Phe Ser Gly Ala Ala Leu Val Arg
530 535 540
Gln Ile Phe Val Tyr Gly Asn Ser Glu Arg Ser Phe Leu Leu Ala Val
545 550 555 560
Val Val Pro Thr Pro Glu Ala Leu Glu Gln Tyr Asp Pro Ala Ala Leu
565 570 575
Lys Ala Ala Leu Ala Asp Ser Leu Gln Arg Thr Ala Arg Asp Ala Glu
580 585 590
Leu Gln Ser Tyr Glu Val Pro Ala Asp Phe Ile Val Glu Thr Glu Pro
595 600 605
Phe Ser Ala Ala Asn Gly Leu Leu Ser Gly Val Gly Lys Leu Leu Arg
610 615 620
Pro Asn Leu Lys Asp Arg Tyr Gly Gln Arg Leu Glu Gln Met Tyr Ala
625 630 635 640
Asp Ile Ala Ala Thr Gln Ala Asn Gln Leu Arg Glu Leu Arg Arg Ala
645 650 655
Ala Ala Thr Gln Pro Val Ile Asp Thr Leu Thr Gln Ala Ala Ala Thr
660 665 670
Ile Leu Gly Thr Gly Ser Glu Val Ala Ser Asp Ala His Phe Thr Asp
675 680 685
Leu Gly Gly Asp Ser Leu Ser Ala Leu Thr Leu Ser Asn Leu Leu Ser
690 695 700
Asp Phe Phe Gly Phe Glu Val Pro Val Gly Thr Ile Val Asn Pro Ala
705 710 715 720
Thr Asn Leu Ala Gln Leu Ala Gln His Ile Glu Ala Gln Arg Thr Ala
725 730 735
Gly Asp Arg Arg Pro Ser Phe Thr Thr Val His Gly Ala Asp Ala Thr
740 745 750
Glu Ile Arg Ala Ser Glu Leu Thr Leu Asp Lys Phe Ile Asp Ala Glu
755 760 765
Thr Leu Arg Ala Ala Pro Gly Leu Pro Lys Val Thr Thr Glu Pro Arg
770 775 780
Thr Val Leu Leu Ser Gly Ala Asn Gly Trp Leu Gly Arg Phe Leu Thr
785 790 795 800
Leu Gln Trp Leu Glu Arg Leu Ala Pro Val Gly Gly Thr Leu Ile Thr
805 810 815
Ile Val Arg Gly Arg Asp Asp Ala Ala Ala Arg Ala Arg Leu Thr Gln
820 825 830
Ala Tyr Asp Thr Asp Pro Glu Leu Ser Arg Arg Phe Ala Glu Leu Ala
835 840 845
Asp Arg His Leu Arg Val Val Ala Gly Asp Ile Gly Asp Pro Asn Leu
850 855 860
Gly Leu Thr Pro Glu Ile Trp His Arg Leu Ala Ala Glu Val Asp Leu
865 870 875 880
Val Val His Pro Ala Ala Leu Val Asn His Val Leu Pro Tyr Arg Gln
885 890 895
Leu Phe Gly Pro Asn Val Val Gly Thr Ala Glu Val Ile Lys Leu Ala
900 905 910
Leu Thr Glu Arg Ile Lys Pro Val Thr Tyr Leu Ser Thr Val Ser Val
915 920 925
Ala Met Gly Ile Pro Asp Phe Glu Glu Asp Gly Asp Ile Arg Thr Val
930 935 940
Ser Pro Val Arg Pro Leu Asp Gly Gly Tyr Ala Asn Gly Tyr Gly Asn
945 950 955 960
Ser Lys Trp Ala Gly Glu Val Leu Leu Arg Glu Ala His Asp Leu Cys
965 970 975
Gly Leu Pro Val Ala Thr Phe Arg Ser Asp Met Ile Leu Ala His Pro
980 985 990
Arg Tyr Arg Gly Gln Val Asn Val Pro Asp Met Phe Thr Arg Leu Leu
995 1000 1005
Leu Ser Leu Leu Ile Thr Gly Val Ala Pro Arg Ser Phe Tyr Ile Gly
1010 1015 1020
Asp Gly Glu Arg Pro Arg Ala His Tyr Pro Gly Leu Thr Val Asp Phe
1025 1030 1035 1040
Val Ala Glu Ala Val Thr Thr Leu Gly Ala Gln Gln Arg Glu Gly Tyr
1045 1050 1055
Val Ser Tyr Asp Val Met Asn Pro His Asp Asp Gly Ile Ser Leu Asp
1060 1065 1070
Val Phe Val Asp Trp Leu Ile Arg Ala Gly His Pro Ile Asp Arg Val
1075 1080 1085
Asp Asp Tyr Asp Asp Trp Val Arg Arg Phe Glu Thr Ala Leu Thr Ala
1090 1095 1100
Leu Pro Glu Lys Arg Arg Ala Gln Thr Val Leu Pro Leu Leu His Ala
1105 1110 1115 1120
Phe Arg Ala Pro Gln Ala Pro Leu Arg Gly Ala Pro Glu Pro Thr Glu
1125 1130 1135
Val Phe His Ala Ala Val Arg Thr Ala Lys Val Gly Pro Gly Asp Ile
1140 1145 1150
Pro His Leu Asp Glu Ala Leu Ile Asp Lys Tyr Ile Arg Asp Leu Arg
1155 1160 1165
Glu Phe Gly Leu Ile
1170
<210> 5
<211> 1148
<212> PRT
<213> Segniliparus rugosus
<400> 5
Met Gly Asp Gly Glu Glu Arg Ala Lys Arg Phe Phe Gln Arg Ile Gly
1 5 10 15
Glu Leu Ser Ala Thr Asp Pro Gln Phe Ala Ala Ala Ala Pro Asp Pro
20 25 30
Ala Val Val Glu Ala Val Ser Asp Pro Ser Leu Ser Phe Thr Arg Tyr
35 40 45
Leu Asp Thr Leu Met Arg Gly Tyr Ala Glu Arg Pro Ala Leu Ala His
50 55 60
Arg Val Gly Ala Gly Tyr Glu Thr Ile Ser Tyr Gly Glu Leu Trp Ala
65 70 75 80
Arg Val Gly Ala Ile Ala Ala Ala Trp Gln Ala Asp Gly Leu Ala Pro
85 90 95
Gly Asp Phe Val Ala Thr Val Gly Phe Thr Ser Pro Asp Tyr Val Ala
100 105 110
Val Asp Leu Ala Ala Ala Arg Ser Gly Leu Val Ser Val Pro Leu Gln
115 120 125
Ala Gly Ala Ser Leu Ala Gln Leu Val Gly Ile Leu Glu Glu Thr Glu
130 135 140
Pro Lys Val Leu Ala Ala Ser Ala Ser Ser Leu Glu Gly Ala Val Ala
145 150 155 160
Cys Ala Leu Ala Ala Pro Ser Val Gln Arg Leu Val Val Phe Asp Leu
165 170 175
Arg Gly Pro Asp Ala Ser Glu Ser Ala Ala Asp Glu Arg Arg Gly Ala
180 185 190
Leu Ala Asp Ala Glu Glu Gln Leu Ala Arg Ala Gly Arg Ala Val Val
195 200 205
Val Glu Thr Leu Ala Asp Leu Ala Ala Arg Gly Glu Ala Leu Pro Glu
210 215 220
Ala Pro Leu Phe Glu Pro Ala Glu Gly Glu Asp Pro Leu Ala Leu Leu
225 230 235 240
Ile Tyr Thr Ser Gly Ser Thr Gly Ala Pro Lys Gly Ala Met Tyr Ser
245 250 255
Gln Arg Leu Val Ser Gln Leu Trp Gly Arg Thr Pro Val Val Pro Gly
260 265 270
Met Pro Asn Ile Ser Leu His Tyr Met Pro Leu Ser His Ser Tyr Gly
275 280 285
Arg Ala Val Leu Ala Gly Ala Leu Ser Ala Gly Gly Thr Ala His Phe
290 295 300
Thr Ala Asn Ser Asp Leu Ser Thr Leu Phe Glu Asp Ile Ala Leu Ala
305 310 315 320
Arg Pro Thr Phe Leu Ala Leu Val Pro Arg Val Cys Glu Met Leu Phe
325 330 335
Gln Glu Ser Gln Arg Gly Gln Asp Val Ala Glu Leu Arg Glu Arg Val
340 345 350
Leu Gly Gly Arg Leu Leu Val Ala Val Cys Gly Ser Ala Pro Leu Ser
355 360 365
Pro Glu Met Arg Ala Phe Met Glu Glu Val Leu Gly Phe Pro Leu Leu
370 375 380
Asp Gly Tyr Gly Ser Thr Glu Ala Leu Gly Val Met Arg Asn Gly Ile
385 390 395 400
Ile Gln Arg Pro Pro Val Ile Asp Tyr Lys Leu Val Asp Val Pro Glu
405 410 415
Leu Gly Tyr Arg Thr Thr Asp Lys Pro Tyr Pro Arg Gly Glu Leu Cys
420 425 430
Ile Arg Ser Thr Ser Leu Ile Ser Gly Tyr Tyr Lys Arg Pro Glu Ile
435 440 445
Thr Ala Glu Val Phe Asp Ala Gln Gly Tyr Tyr Lys Thr Gly Asp Val
450 455 460
Met Ala Glu Ile Ala Pro Asp His Leu Val Tyr Val Asp Arg Ser Lys
465 470 475 480
Asn Val Leu Lys Leu Ser Gln Gly Glu Phe Val Ala Val Ala Lys Leu
485 490 495
Glu Ala Ala Tyr Gly Thr Ser Pro Tyr Val Lys Gln Ile Phe Val Tyr
500 505 510
Gly Asn Ser Glu Arg Ser Phe Leu Leu Ala Val Val Val Pro Asn Ala
515 520 525
Glu Val Leu Gly Ala Arg Asp Gln Glu Glu Ala Lys Pro Leu Ile Ala
530 535 540
Ala Ser Leu Gln Lys Ile Ala Lys Glu Ala Gly Leu Gln Ser Tyr Glu
545 550 555 560
Val Pro Arg Asp Phe Leu Ile Glu Thr Glu Pro Phe Thr Thr Gln Asn
565 570 575
Gly Leu Leu Ser Glu Val Gly Lys Leu Leu Arg Pro Lys Leu Lys Ala
580 585 590
Arg Tyr Gly Glu Ala Leu Glu Ala Arg Tyr Asp Glu Ile Ala His Gly
595 600 605
Gln Ala Asp Glu Leu Arg Ala Leu Arg Asp Gly Ala Gly Gln Arg Pro
610 615 620
Val Val Glu Thr Val Val Arg Ala Ala Val Ala Ile Ser Gly Ser Glu
625 630 635 640
Gly Ala Glu Val Gly Pro Glu Ala Asn Phe Ala Asp Leu Gly Gly Asp
645 650 655
Ser Leu Ser Ala Leu Ser Leu Ala Asn Leu Leu His Asp Val Phe Glu
660 665 670
Val Glu Val Pro Val Arg Ile Ile Ile Gly Pro Thr Ala Ser Leu Ala
675 680 685
Gly Ile Ala Lys His Ile Glu Ala Glu Arg Ala Gly Ala Ser Ala Pro
690 695 700
Thr Ala Ala Ser Val His Gly Ala Gly Ala Thr Arg Ile Arg Ala Ser
705 710 715 720
Glu Leu Thr Leu Glu Lys Phe Leu Pro Glu Asp Leu Leu Ala Ala Ala
725 730 735
Lys Gly Leu Pro Ala Ala Asp Gln Val Arg Thr Val Leu Leu Thr Gly
740 745 750
Ala Asn Gly Trp Leu Gly Arg Phe Leu Ala Leu Glu Gln Leu Glu Arg
755 760 765
Leu Ala Arg Ser Gly Gln Asp Gly Gly Lys Leu Ile Cys Leu Val Arg
770 775 780
Gly Lys Asp Ala Ala Ala Ala Arg Arg Arg Ile Glu Glu Thr Leu Gly
785 790 795 800
Thr Asp Pro Ala Leu Ala Ala Arg Phe Ala Glu Leu Ala Glu Gly Arg
805 810 815
Leu Glu Val Val Pro Gly Asp Val Gly Glu Pro Lys Phe Gly Leu Asp
820 825 830
Asp Ala Ala Trp Asp Arg Leu Ala Glu Glu Val Asp Val Ile Val His
835 840 845
Pro Ala Ala Leu Val Asn His Val Leu Pro Tyr His Gln Leu Phe Gly
850 855 860
Pro Asn Val Val Gly Thr Ala Glu Ile Ile Arg Leu Ala Ile Thr Ala
865 870 875 880
Lys Arg Lys Pro Val Thr Tyr Leu Ser Thr Val Ala Val Ala Ala Gly
885 890 895
Val Glu Pro Ser Ser Phe Glu Glu Asp Gly Asp Ile Arg Ala Val Val
900 905 910
Pro Glu Arg Pro Leu Gly Asp Gly Tyr Ala Asn Gly Tyr Gly Asn Ser
915 920 925
Lys Trp Ala Gly Glu Val Leu Leu Arg Glu Ala His Glu Leu Val Gly
930 935 940
Leu Pro Val Ala Val Phe Arg Ser Asp Met Ile Leu Ala His Thr Arg
945 950 955 960
Tyr Thr Gly Gln Leu Asn Val Pro Asp Gln Phe Thr Arg Leu Val Leu
965 970 975
Ser Leu Leu Ala Thr Gly Ile Ala Pro Lys Ser Phe Tyr Gln Gln Gly
980 985 990
Ala Ala Gly Glu Arg Gln Arg Ala His Tyr Asp Gly Ile Pro Val Asp
995 1000 1005
Phe Thr Ala Glu Ala Ile Thr Thr Leu Gly Ala Glu Pro Ser Trp Phe
1010 1015 1020
Asp Gly Gly Ala Gly Phe Arg Ser Phe Asp Val Phe Asn Pro His His
1025 1030 1035 1040
Asp Gly Val Gly Leu Asp Glu Phe Val Asp Trp Leu Ile Glu Ala Gly
1045 1050 1055
His Pro Ile Ser Arg Ile Asp Asp His Lys Glu Trp Phe Ala Arg Phe
1060 1065 1070
Glu Thr Ala Val Arg Gly Leu Pro Glu Ala Gln Arg Gln His Ser Leu
1075 1080 1085
Leu Pro Leu Leu Arg Ala Tyr Ser Phe Pro His Pro Pro Val Asp Gly
1090 1095 1100
Ser Val Tyr Pro Thr Gly Lys Phe Gln Gly Ala Val Lys Ala Ala Gln
1105 1110 1115 1120
Val Gly Ser Asp His Asp Val Pro His Leu Gly Lys Ala Leu Ile Val
1125 1130 1135
Lys Tyr Ala Asp Asp Leu Lys Ala Leu Gly Leu Leu
1140 1145
<210> 6
<211> 1185
<212> PRT
<213> Mycobacterium massiliense
<400> 6
Met Thr Asn Glu Thr Asn Pro Gln Gln Glu Gln Leu Ser Arg Arg Ile
1 5 10 15
Glu Ser Leu Arg Glu Ser Asp Pro Gln Phe Arg Ala Ala Gln Pro Asp
20 25 30
Pro Ala Val Ala Glu Gln Val Leu Arg Pro Gly Leu His Leu Ser Glu
35 40 45
Ala Ile Ala Ala Leu Met Thr Gly Tyr Ala Glu Arg Pro Ala Leu Gly
50 55 60
Glu Arg Ala Arg Glu Leu Val Ile Asp Gln Asp Gly Arg Thr Thr Leu
65 70 75 80
Arg Leu Leu Pro Arg Phe Asp Thr Thr Thr Tyr Gly Glu Leu Trp Ser
85 90 95
Arg Thr Thr Ser Val Ala Ala Ala Trp His His Asp Ala Thr His Pro
100 105 110
Val Lys Ala Gly Asp Leu Val Ala Thr Leu Gly Phe Thr Ser Ile Asp
115 120 125
Tyr Thr Val Leu Asp Leu Ala Ile Met Ile Leu Gly Gly Val Ala Val
130 135 140
Pro Leu Gln Thr Ser Ala Pro Ala Ser Gln Trp Thr Thr Ile Leu Ala
145 150 155 160
Glu Ala Glu Pro Asn Thr Leu Ala Val Ser Ile Glu Leu Ile Gly Ala
165 170 175
Ala Met Glu Ser Val Arg Ala Thr Pro Ser Ile Lys Gln Val Val Val
180 185 190
Phe Asp Tyr Thr Pro Glu Val Asp Asp Gln Arg Glu Ala Phe Glu Ala
195 200 205
Ala Ser Thr Gln Leu Ala Gly Thr Gly Ile Ala Leu Glu Thr Leu Asp
210 215 220
Ala Val Ile Ala Arg Gly Ala Ala Leu Pro Ala Ala Pro Leu Tyr Ala
225 230 235 240
Pro Ser Ala Gly Asp Asp Pro Leu Ala Leu Leu Ile Tyr Thr Ser Gly
245 250 255
Ser Thr Gly Ala Pro Lys Gly Ala Met His Ser Glu Asn Ile Val Arg
260 265 270
Arg Trp Trp Ile Arg Glu Asp Val Met Ala Gly Thr Glu Asn Leu Pro
275 280 285
Met Ile Gly Leu Asn Phe Met Pro Met Ser His Ile Met Gly Arg Gly
290 295 300
Thr Leu Thr Ser Thr Leu Ser Thr Gly Gly Thr Gly Tyr Phe Ala Ala
305 310 315 320
Ser Ser Asp Met Ser Thr Leu Phe Glu Asp Met Glu Leu Ile Arg Pro
325 330 335
Thr Ala Leu Ala Leu Val Pro Arg Val Cys Asp Met Val Phe Gln Arg
340 345 350
Phe Gln Thr Glu Val Asp Arg Arg Leu Ala Ser Gly Asp Thr Ala Ser
355 360 365
Ala Glu Ala Val Ala Ala Glu Val Lys Ala Asp Ile Arg Asp Asn Leu
370 375 380
Phe Gly Gly Arg Val Ser Ala Val Met Val Gly Ser Ala Pro Leu Ser
385 390 395 400
Glu Glu Leu Gly Glu Phe Ile Glu Ser Cys Phe Glu Leu Asn Leu Thr
405 410 415
Asp Gly Tyr Gly Ser Thr Glu Ala Gly Met Val Phe Arg Asp Gly Ile
420 425 430
Val Gln Arg Pro Pro Val Ile Asp Tyr Lys Leu Val Asp Val Pro Glu
435 440 445
Leu Gly Tyr Phe Ser Thr Asp Lys Pro His Pro Arg Gly Glu Leu Leu
450 455 460
Leu Lys Thr Asp Gly Met Phe Leu Gly Tyr Tyr Lys Arg Pro Glu Val
465 470 475 480
Thr Ala Ser Val Phe Asp Ala Asp Gly Phe Tyr Met Thr Gly Asp Ile
485 490 495
Val Ala Glu Leu Ala His Asp Asn Ile Glu Ile Ile Asp Arg Arg Asn
500 505 510
Asn Val Leu Lys Leu Ser Gln Gly Glu Phe Val Ala Val Ala Thr Leu
515 520 525
Glu Ala Glu Tyr Ala Asn Ser Pro Val Val His Gln Ile Tyr Val Tyr
530 535 540
Gly Ser Ser Glu Arg Ser Tyr Leu Leu Ala Val Val Val Pro Thr Pro
545 550 555 560
Glu Ala Val Ala Ala Ala Lys Gly Asp Ala Ala Ala Leu Lys Thr Thr
565 570 575
Ile Ala Asp Ser Leu Gln Asp Ile Ala Lys Glu Ile Gln Leu Gln Ser
580 585 590
Tyr Glu Val Pro Arg Asp Phe Ile Ile Glu Pro Gln Pro Phe Thr Gln
595 600 605
Gly Asn Gly Leu Leu Thr Gly Ile Ala Lys Leu Ala Arg Pro Asn Leu
610 615 620
Lys Ala His Tyr Gly Pro Arg Leu Glu Gln Met Tyr Ala Glu Ile Ala
625 630 635 640
Glu Gln Gln Ala Ala Glu Leu Arg Ala Leu His Gly Val Asp Pro Asp
645 650 655
Lys Pro Ala Leu Glu Thr Val Leu Lys Ala Ala Gln Ala Leu Leu Gly
660 665 670
Val Ser Ser Ala Glu Leu Ala Ala Asp Ala His Phe Thr Asp Leu Gly
675 680 685
Gly Asp Ser Leu Ser Ala Leu Ser Phe Ser Asp Leu Leu Arg Asp Ile
690 695 700
Phe Ala Val Glu Val Pro Val Gly Val Ile Val Ser Ala Ala Asn Asp
705 710 715 720
Leu Gly Gly Val Ala Lys Phe Val Asp Glu Gln Arg His Ser Gly Gly
725 730 735
Thr Arg Pro Thr Ala Glu Thr Val His Gly Ala Gly His Thr Glu Ile
740 745 750
Arg Ala Ala Asp Leu Thr Leu Asp Lys Phe Ile Asp Glu Ala Thr Leu
755 760 765
His Ala Ala Pro Ser Leu Pro Lys Ala Ala Gly Ile Pro His Thr Val
770 775 780
Leu Leu Thr Gly Ser Asn Gly Tyr Leu Gly His Tyr Leu Ala Leu Glu
785 790 795 800
Trp Leu Glu Arg Leu Asp Lys Thr Asp Gly Lys Leu Ile Val Ile Val
805 810 815
Arg Gly Lys Asn Ala Glu Ala Ala Tyr Gly Arg Leu Glu Glu Ala Phe
820 825 830
Asp Thr Gly Asp Thr Glu Leu Leu Ala His Phe Arg Ser Leu Ala Asp
835 840 845
Lys His Leu Glu Val Leu Ala Gly Asp Ile Gly Asp Pro Asn Leu Gly
850 855 860
Leu Asp Ala Asp Thr Trp Gln Arg Leu Ala Asp Thr Val Asp Val Ile
865 870 875 880
Val His Pro Ala Ala Leu Val Asn His Val Leu Pro Tyr Asn Gln Leu
885 890 895
Phe Gly Pro Asn Val Val Gly Thr Ala Glu Ile Ile Lys Leu Ala Ile
900 905 910
Thr Thr Lys Ile Lys Pro Val Thr Tyr Leu Ser Thr Val Ala Val Ala
915 920 925
Ala Tyr Val Asp Pro Thr Thr Phe Asp Glu Glu Ser Asp Ile Arg Leu
930 935 940
Ile Ser Ala Val Arg Pro Ile Asp Asp Gly Tyr Ala Asn Gly Tyr Gly
945 950 955 960
Asn Ala Lys Trp Ala Gly Glu Val Leu Leu Arg Glu Ala His Asp Leu
965 970 975
Cys Gly Leu Pro Val Ala Val Phe Arg Ser Asp Met Ile Leu Ala His
980 985 990
Ser Arg Tyr Thr Gly Gln Leu Asn Val Pro Asp Gln Phe Thr Arg Leu
995 1000 1005
Ile Leu Ser Leu Ile Ala Thr Gly Ile Ala Pro Gly Ser Phe Tyr Gln
1010 1015 1020
Ala Gln Thr Thr Gly Glu Arg Pro Leu Ala His Tyr Asp Gly Leu Pro
1025 1030 1035 1040
Gly Asp Phe Thr Ala Glu Ala Ile Thr Thr Leu Gly Thr Gln Val Pro
1045 1050 1055
Glu Gly Ser Glu Gly Phe Val Thr Tyr Asp Cys Val Asn Pro His Ala
1060 1065 1070
Asp Gly Ile Ser Leu Asp Asn Phe Val Asp Trp Leu Ile Glu Ala Gly
1075 1080 1085
Tyr Pro Ile Ala Arg Ile Asp Asn Tyr Thr Glu Trp Phe Thr Arg Phe
1090 1095 1100
Asp Thr Ala Ile Arg Gly Leu Ser Glu Lys Gln Lys Gln His Ser Leu
1105 1110 1115 1120
Leu Pro Leu Leu His Ala Phe Glu Gln Pro Ser Ala Ala Glu Asn His
1125 1130 1135
Gly Val Val Pro Ala Lys Arg Phe Gln His Ala Val Gln Ala Ala Gly
1140 1145 1150
Ile Gly Pro Val Gly Gln Asp Gly Thr Thr Asp Ile Pro His Leu Ser
1155 1160 1165
Arg Arg Leu Ile Val Lys Tyr Ala Lys Asp Leu Glu Gln Leu Gly Leu
1170 1175 1180
Leu
1185
<210> 7
<211> 1186
<212> PRT
<213> Segniliparus rotundus
<400> 7
Met Thr Gln Ser His Thr Gln Gly Pro Gln Ala Ser Ala Ala His Ser
1 5 10 15
Arg Leu Ala Arg Arg Ala Ala Glu Leu Leu Ala Thr Asp Pro Gln Ala
20 25 30
Ala Ala Thr Leu Pro Asp Pro Glu Val Val Arg Gln Ala Thr Arg Pro
35 40 45
Gly Leu Arg Leu Ala Glu Arg Val Asp Ala Ile Leu Ser Gly Tyr Ala
50 55 60
Asp Arg Pro Ala Leu Gly Gln Arg Ser Phe Gln Thr Val Lys Asp Pro
65 70 75 80
Ile Thr Gly Arg Ser Ser Val Glu Leu Leu Pro Thr Phe Asp Thr Ile
85 90 95
Thr Tyr Arg Glu Leu Arg Glu Arg Ala Thr Ala Ile Ala Ser Asp Leu
100 105 110
Ala His His Pro Gln Ala Pro Ala Lys Pro Gly Asp Phe Leu Ala Ser
115 120 125
Ile Gly Phe Ile Ser Val Asp Tyr Val Ala Ile Asp Ile Ala Gly Val
130 135 140
Phe Ala Gly Leu Thr Ala Val Pro Leu Gln Thr Gly Ala Thr Leu Ala
145 150 155 160
Thr Leu Thr Ala Ile Thr Ala Glu Thr Ala Pro Thr Leu Phe Ala Ala
165 170 175
Ser Ile Glu His Leu Pro Thr Ala Val Asp Ala Val Leu Ala Thr Pro
180 185 190
Ser Val Arg Arg Leu Leu Val Phe Asp Tyr Arg Ala Gly Ser Asp Glu
195 200 205
Asp Arg Glu Ala Val Glu Ala Ala Lys Arg Lys Ile Ala Asp Ala Gly
210 215 220
Ser Ser Val Leu Val Asp Val Leu Asp Glu Val Ile Ala Arg Gly Lys
225 230 235 240
Ser Ala Pro Lys Ala Pro Leu Pro Pro Ala Thr Asp Ala Gly Asp Asp
245 250 255
Ser Leu Ser Leu Leu Ile Tyr Thr Ser Gly Ser Thr Gly Thr Pro Lys
260 265 270
Gly Ala Met Tyr Pro Glu Arg Asn Val Ala His Phe Trp Gly Gly Val
275 280 285
Trp Ala Ala Ala Phe Asp Glu Asp Ala Ala Pro Pro Val Pro Ala Ile
290 295 300
Asn Ile Thr Phe Leu Pro Leu Ser His Val Ala Ser Arg Leu Ser Leu
305 310 315 320
Met Pro Thr Leu Ala Arg Gly Gly Leu Met His Phe Val Ala Lys Ser
325 330 335
Asp Leu Ser Thr Leu Phe Glu Asp Leu Lys Leu Ala Arg Pro Thr Asn
340 345 350
Leu Phe Leu Val Pro Arg Val Val Glu Met Leu Tyr Gln His Tyr Gln
355 360 365
Ser Glu Leu Asp Arg Arg Gly Val Gln Asp Gly Thr Arg Glu Ala Glu
370 375 380
Ala Val Lys Asp Asp Leu Arg Thr Gly Leu Leu Gly Gly Arg Ile Leu
385 390 395 400
Thr Ala Gly Phe Gly Ser Ala Pro Leu Ser Ala Glu Leu Ala Gly Phe
405 410 415
Ile Glu Ser Leu Leu Gln Ile His Leu Val Asp Gly Tyr Gly Ser Thr
420 425 430
Glu Ala Gly Pro Val Trp Arg Asp Gly Tyr Leu Val Lys Pro Pro Val
435 440 445
Thr Asp Tyr Lys Leu Ile Asp Val Pro Glu Leu Gly Tyr Phe Ser Thr
450 455 460
Asp Ser Pro His Pro Arg Gly Glu Leu Ala Ile Lys Thr Gln Thr Ile
465 470 475 480
Leu Pro Gly Tyr Tyr Lys Arg Pro Glu Thr Thr Ala Glu Val Phe Asp
485 490 495
Glu Asp Gly Phe Tyr Leu Thr Gly Asp Val Val Ala Gln Ile Gly Pro
500 505 510
Glu Gln Phe Ala Tyr Val Asp Arg Arg Lys Asn Val Leu Lys Leu Ser
515 520 525
Gln Gly Glu Phe Val Thr Leu Ala Lys Leu Glu Ala Ala Tyr Ser Ser
530 535 540
Ser Pro Leu Val Arg Gln Leu Phe Val Tyr Gly Ser Ser Glu Arg Ser
545 550 555 560
Tyr Leu Leu Ala Val Ile Val Pro Thr Pro Asp Ala Leu Lys Lys Phe
565 570 575
Gly Val Gly Glu Ala Ala Lys Ala Ala Leu Gly Glu Ser Leu Gln Lys
580 585 590
Ile Ala Arg Asp Glu Gly Leu Gln Ser Tyr Glu Val Pro Arg Asp Phe
595 600 605
Ile Ile Glu Thr Asp Pro Phe Thr Val Glu Asn Gly Leu Leu Ser Asp
610 615 620
Ala Arg Lys Ser Leu Arg Pro Lys Leu Lys Glu His Tyr Gly Glu Arg
625 630 635 640
Leu Glu Ala Met Tyr Lys Glu Leu Ala Asp Gly Gln Ala Asn Glu Leu
645 650 655
Arg Asp Ile Arg Arg Gly Val Gln Gln Arg Pro Thr Leu Glu Thr Val
660 665 670
Arg Arg Ala Ala Ala Ala Met Leu Gly Ala Ser Ala Ala Glu Ile Lys
675 680 685
Pro Asp Ala His Phe Thr Asp Leu Gly Gly Asp Ser Leu Ser Ala Leu
690 695 700
Thr Phe Ser Asn Phe Leu His Asp Leu Phe Glu Val Asp Val Pro Val
705 710 715 720
Gly Val Ile Val Ser Ala Ala Asn Thr Leu Gly Ser Val Ala Glu His
725 730 735
Ile Asp Ala Gln Leu Ala Gly Gly Arg Ala Arg Pro Thr Phe Ala Thr
740 745 750
Val His Gly Lys Gly Ser Thr Thr Ile Lys Ala Ser Asp Leu Thr Leu
755 760 765
Asp Lys Phe Ile Asp Glu Gln Thr Leu Glu Ala Ala Lys His Leu Pro
770 775 780
Lys Pro Ala Asp Pro Pro Arg Thr Val Leu Leu Thr Gly Ala Asn Gly
785 790 795 800
Trp Leu Gly Arg Phe Leu Ala Leu Glu Trp Leu Glu Arg Leu Ala Pro
805 810 815
Ala Gly Gly Lys Leu Ile Thr Ile Val Arg Gly Lys Asp Ala Ala Gln
820 825 830
Ala Lys Ala Arg Leu Asp Ala Ala Tyr Glu Ser Gly Asp Pro Lys Leu
835 840 845
Ala Gly His Tyr Gln Asp Leu Ala Ala Thr Thr Leu Glu Val Leu Ala
850 855 860
Gly Asp Phe Ser Glu Pro Arg Leu Gly Leu Asp Glu Ala Thr Trp Asn
865 870 875 880
Arg Leu Ala Asp Glu Val Asp Phe Ile Ser His Pro Gly Ala Leu Val
885 890 895
Asn His Val Leu Pro Tyr Asn Gln Leu Phe Gly Pro Asn Val Ala Gly
900 905 910
Val Ala Glu Ile Ile Lys Leu Ala Ile Thr Thr Arg Ile Lys Pro Val
915 920 925
Thr Tyr Leu Ser Thr Val Ala Val Ala Ala Gly Val Glu Pro Ser Ala
930 935 940
Leu Asp Glu Asp Gly Asp Ile Arg Thr Val Ser Ala Glu Arg Ser Val
945 950 955 960
Asp Glu Gly Tyr Ala Asn Gly Tyr Gly Asn Ser Lys Trp Gly Gly Glu
965 970 975
Val Leu Leu Arg Glu Ala His Asp Arg Thr Gly Leu Pro Val Arg Val
980 985 990
Phe Arg Ser Asp Met Ile Leu Ala His Gln Lys Tyr Thr Gly Gln Val
995 1000 1005
Asn Ala Thr Asp Gln Phe Thr Arg Leu Val Gln Ser Leu Leu Ala Thr
1010 1015 1020
Gly Leu Ala Pro Lys Ser Phe Tyr Glu Leu Asp Ala Gln Gly Asn Arg
1025 1030 1035 1040
Gln Arg Ala His Tyr Asp Gly Ile Pro Val Asp Phe Thr Ala Glu Ser
1045 1050 1055
Ile Thr Thr Leu Gly Gly Asp Gly Leu Glu Gly Tyr Arg Ser Tyr Asn
1060 1065 1070
Val Phe Asn Pro His Arg Asp Gly Val Gly Leu Asp Glu Phe Val Asp
1075 1080 1085
Trp Leu Ile Glu Ala Gly His Pro Ile Thr Arg Ile Asp Asp Tyr Asp
1090 1095 1100
Gln Trp Leu Ser Arg Phe Glu Thr Ser Leu Arg Gly Leu Pro Glu Ser
1105 1110 1115 1120
Lys Arg Gln Ala Ser Val Leu Pro Leu Leu His Ala Phe Ala Arg Pro
1125 1130 1135
Gly Pro Ala Val Asp Gly Ser Pro Phe Arg Asn Thr Val Phe Arg Thr
1140 1145 1150
Asp Val Gln Lys Ala Lys Ile Gly Ala Glu His Asp Ile Pro His Leu
1155 1160 1165
Gly Lys Ala Leu Val Leu Lys Tyr Ala Asp Asp Ile Lys Gln Leu Gly
1170 1175 1180
Leu Leu
1185
<210> 8
<211> 459
<212> PRT
<213> 紫色色桿菌
<400> 8
Met Gln Lys Gln Arg Thr Thr Ser Gln Trp Arg Glu Leu Asp Ala Ala
1 5 10 15
His His Leu His Pro Phe Thr Asp Thr Ala Ser Leu Asn Gln Ala Gly
20 25 30
Ala Arg Val Met Thr Arg Gly Glu Gly Val Tyr Leu Trp Asp Ser Glu
35 40 45
Gly Asn Lys Ile Ile Asp Gly Met Ala Gly Leu Trp Cys Val Asn Val
50 55 60
Gly Tyr Gly Arg Lys Asp Phe Ala Glu Ala Ala Arg Arg Gln Met Glu
65 70 75 80
Glu Leu Pro Phe Tyr Asn Thr Phe Phe Lys Thr Thr His Pro Ala Val
85 90 95
Val Glu Leu Ser Ser Leu Leu Ala Glu Val Thr Pro Ala Gly Phe Asp
100 105 110
Arg Val Phe Tyr Thr Asn Ser Gly Ser Glu Ser Val Asp Thr Met Ile
115 120 125
Arg Met Val Arg Arg Tyr Trp Asp Val Gln Gly Lys Pro Glu Lys Lys
130 135 140
Thr Leu Ile Gly Arg Trp Asn Gly Tyr His Gly Ser Thr Ile Gly Gly
145 150 155 160
Ala Ser Leu Gly Gly Met Lys Tyr Met His Glu Gln Gly Asp Leu Pro
165 170 175
Ile Pro Gly Met Ala His Ile Glu Gln Pro Trp Trp Tyr Lys His Gly
180 185 190
Lys Asp Met Thr Pro Asp Glu Phe Gly Val Val Ala Ala Arg Trp Leu
195 200 205
Glu Glu Lys Ile Leu Glu Ile Gly Ala Asp Lys Val Ala Ala Phe Val
210 215 220
Gly Glu Pro Ile Gln Gly Ala Gly Gly Val Ile Val Pro Pro Ala Thr
225 230 235 240
Tyr Trp Pro Glu Ile Glu Arg Ile Cys Arg Lys Tyr Asp Val Leu Leu
245 250 255
Val Ala Asp Glu Val Ile Cys Gly Phe Gly Arg Thr Gly Glu Trp Phe
260 265 270
Gly His Gln His Phe Gly Phe Gln Pro Asp Leu Phe Thr Ala Ala Lys
275 280 285
Gly Leu Ser Ser Gly Tyr Leu Pro Ile Gly Ala Val Phe Val Gly Lys
290 295 300
Arg Val Ala Glu Gly Leu Ile Ala Gly Gly Asp Phe Asn His Gly Phe
305 310 315 320
Thr Tyr Ser Gly His Pro Val Cys Ala Ala Val Ala His Ala Asn Val
325 330 335
Ala Ala Leu Arg Asp Glu Gly Ile Val Gln Arg Val Lys Asp Asp Ile
340 345 350
Gly Pro Tyr Met Gln Lys Arg Trp Arg Glu Thr Phe Ser Arg Phe Glu
355 360 365
His Val Asp Asp Val Arg Gly Val Gly Met Val Gln Ala Phe Thr Leu
370 375 380
Val Lys Asn Lys Ala Lys Arg Glu Leu Phe Pro Asp Phe Gly Glu Ile
385 390 395 400
Gly Thr Leu Cys Arg Asp Ile Phe Phe Arg Asn Asn Leu Ile Met Arg
405 410 415
Ala Cys Gly Asp His Ile Val Ser Ala Pro Pro Leu Val Met Thr Arg
420 425 430
Ala Glu Val Asp Glu Met Leu Ala Val Ala Glu Arg Cys Leu Glu Glu
435 440 445
Phe Glu Gln Thr Leu Lys Ala Arg Gly Leu Ala
450 455
<210> 9
<211> 468
<212> PRT
<213> 銅綠假單胞菌
<400> 9
Met Asn Ala Arg Leu His Ala Thr Ser Pro Leu Gly Asp Ala Asp Leu
1 5 10 15
Val Arg Ala Asp Gln Ala His Tyr Met His Gly Tyr His Val Phe Asp
20 25 30
Asp His Arg Val Asn Gly Ser Leu Asn Ile Ala Ala Gly Asp Gly Ala
35 40 45
Tyr Ile Tyr Asp Thr Ala Gly Asn Arg Tyr Leu Asp Ala Val Gly Gly
50 55 60
Met Trp Cys Thr Asn Ile Gly Leu Gly Arg Glu Glu Met Ala Arg Thr
65 70 75 80
Val Ala Glu Gln Thr Arg Leu Leu Ala Tyr Ser Asn Pro Phe Cys Asp
85 90 95
Met Ala Asn Pro Arg Ala Ile Glu Leu Cys Arg Lys Leu Ala Glu Leu
100 105 110
Ala Pro Gly Asp Leu Asp His Val Phe Leu Thr Thr Gly Gly Ser Thr
115 120 125
Ala Val Asp Thr Ala Ile Arg Leu Met His Tyr Tyr Gln Asn Cys Arg
130 135 140
Gly Lys Arg Ala Lys Lys His Val Ile Thr Arg Ile Asn Ala Tyr His
145 150 155 160
Gly Ser Thr Phe Leu Gly Met Ser Leu Gly Gly Lys Ser Ala Asp Arg
165 170 175
Pro Ala Glu Phe Asp Phe Leu Asp Glu Arg Ile His His Leu Ala Cys
180 185 190
Pro Tyr Tyr Tyr Arg Ala Pro Glu Gly Leu Gly Glu Ala Glu Phe Leu
195 200 205
Asp Gly Leu Val Asp Glu Phe Glu Arg Lys Ile Leu Glu Leu Gly Ala
210 215 220
Asp Arg Val Gly Ala Phe Ile Ser Glu Pro Val Phe Gly Ser Gly Gly
225 230 235 240
Val Ile Val Pro Pro Ala Gly Tyr His Arg Arg Met Trp Glu Leu Cys
245 250 255
Gln Arg Tyr Asp Val Leu Tyr Ile Ser Asp Glu Val Val Thr Ser Phe
260 265 270
Gly Arg Leu Gly His Phe Phe Ala Ser Gln Ala Val Phe Gly Val Gln
275 280 285
Pro Asp Ile Ile Leu Thr Ala Lys Gly Leu Thr Ser Gly Tyr Gln Pro
290 295 300
Leu Gly Ala Cys Ile Phe Ser Arg Arg Ile Trp Glu Val Ile Ala Glu
305 310 315 320
Pro Asp Lys Gly Arg Cys Phe Ser His Gly Phe Thr Tyr Ser Gly His
325 330 335
Pro Val Ala Cys Ala Ala Ala Leu Lys Asn Ile Glu Ile Ile Glu Arg
340 345 350
Glu Gly Leu Leu Ala His Ala Asp Glu Val Gly Arg Tyr Phe Glu Glu
355 360 365
Arg Leu Gln Ser Leu Arg Asp Leu Pro Ile Val Gly Asp Val Arg Gly
370 375 380
Met Arg Phe Met Ala Cys Val Glu Phe Val Ala Asp Lys Ala Ser Lys
385 390 395 400
Ala Leu Phe Pro Glu Ser Leu Asn Ile Gly Glu Trp Val His Leu Arg
405 410 415
Ala Gln Lys Arg Gly Leu Leu Val Arg Pro Ile Val His Leu Asn Val
420 425 430
Met Ser Pro Pro Leu Ile Leu Thr Arg Glu Gln Val Asp Thr Val Val
435 440 445
Arg Val Leu Arg Glu Ser Ile Glu Glu Thr Val Glu Asp Leu Val Arg
450 455 460
Ala Gly His Arg
465
<210> 10
<211> 454
<212> PRT
<213> 丁香假單胞菌
<400> 10
Met Ser Ala Asn Asn Pro Gln Thr Leu Glu Trp Gln Ala Leu Ser Ser
1 5 10 15
Glu His His Leu Ala Pro Phe Ser Asp Tyr Lys Gln Leu Lys Glu Lys
20 25 30
Gly Pro Arg Ile Ile Thr Arg Ala Glu Gly Val Tyr Leu Trp Asp Ser
35 40 45
Glu Gly Asn Lys Ile Leu Asp Gly Met Ser Gly Leu Trp Cys Val Ala
50 55 60
Ile Gly Tyr Gly Arg Glu Glu Leu Ala Asp Ala Ala Ser Lys Gln Met
65 70 75 80
Arg Glu Leu Pro Tyr Tyr Asn Leu Phe Phe Gln Thr Ala His Pro Pro
85 90 95
Val Leu Glu Leu Ala Lys Ala Ile Ser Asp Ile Ala Pro Glu Gly Met
100 105 110
Asn His Val Phe Phe Thr Gly Ser Gly Ser Glu Gly Asn Asp Thr Met
115 120 125
Leu Arg Met Val Arg His Tyr Trp Ala Leu Lys Gly Gln Pro Asn Lys
130 135 140
Lys Thr Ile Ile Ser Arg Val Asn Gly Tyr His Gly Ser Thr Val Ala
145 150 155 160
Gly Ala Ser Leu Gly Gly Met Thr Tyr Met His Glu Gln Gly Asp Leu
165 170 175
Pro Ile Pro Gly Val Val His Ile Pro Gln Pro Tyr Trp Phe Gly Glu
180 185 190
Gly Gly Asp Met Thr Pro Asp Glu Phe Gly Ile Trp Ala Ala Glu Gln
195 200 205
Leu Glu Lys Lys Ile Leu Glu Leu Gly Val Glu Asn Val Gly Ala Phe
210 215 220
Ile Ala Glu Pro Ile Gln Gly Ala Gly Gly Val Ile Val Pro Pro Asp
225 230 235 240
Ser Tyr Trp Pro Lys Ile Lys Glu Ile Leu Ser Arg Tyr Asp Ile Leu
245 250 255
Phe Ala Ala Asp Glu Val Ile Cys Gly Phe Gly Arg Thr Ser Glu Trp
260 265 270
Phe Gly Ser Asp Phe Tyr Gly Leu Arg Pro Asp Met Met Thr Ile Ala
275 280 285
Lys Gly Leu Thr Ser Gly Tyr Val Pro Met Gly Gly Leu Ile Val Arg
290 295 300
Asp Glu Ile Val Ala Val Leu Asn Glu Gly Gly Asp Phe Asn His Gly
305 310 315 320
Phe Thr Tyr Ser Gly His Pro Val Ala Ala Ala Val Ala Leu Glu Asn
325 330 335
Ile Arg Ile Leu Arg Glu Glu Lys Ile Val Glu Arg Val Arg Ser Glu
340 345 350
Thr Ala Pro Tyr Leu Gln Lys Arg Leu Arg Glu Leu Ser Asp His Pro
355 360 365
Leu Val Gly Glu Val Arg Gly Val Gly Leu Leu Gly Ala Ile Glu Leu
370 375 380
Val Lys Asp Lys Thr Thr Arg Glu Arg Tyr Thr Asp Lys Gly Ala Gly
385 390 395 400
Met Ile Cys Arg Thr Phe Cys Phe Asp Asn Gly Leu Ile Met Arg Ala
405 410 415
Val Gly Asp Thr Met Ile Ile Ala Pro Pro Leu Val Ile Ser Phe Ala
420 425 430
Gln Ile Asp Glu Leu Val Glu Lys Ala Arg Thr Cys Leu Asp Leu Thr
435 440 445
Leu Ala Val Leu Gln Gly
450
<210> 11
<211> 467
<212> PRT
<213> 球形紅細菌
<400> 11
Met Thr Arg Asn Asp Ala Thr Asn Ala Ala Gly Ala Val Gly Ala Ala
1 5 10 15
Met Arg Asp His Ile Leu Leu Pro Ala Gln Glu Met Ala Lys Leu Gly
20 25 30
Lys Ser Ala Gln Pro Val Leu Thr His Ala Glu Gly Ile Tyr Val His
35 40 45
Thr Glu Asp Gly Arg Arg Leu Ile Asp Gly Pro Ala Gly Met Trp Cys
50 55 60
Ala Gln Val Gly Tyr Gly Arg Arg Glu Ile Val Asp Ala Met Ala His
65 70 75 80
Gln Ala Met Val Leu Pro Tyr Ala Ser Pro Trp Tyr Met Ala Thr Ser
85 90 95
Pro Ala Ala Arg Leu Ala Glu Lys Ile Ala Thr Leu Thr Pro Gly Asp
100 105 110
Leu Asn Arg Ile Phe Phe Thr Thr Gly Gly Ser Thr Ala Val Asp Ser
115 120 125
Ala Leu Arg Phe Ser Glu Phe Tyr Asn Asn Val Leu Gly Arg Pro Gln
130 135 140
Lys Lys Arg Ile Ile Val Arg Tyr Asp Gly Tyr His Gly Ser Thr Ala
145 150 155 160
Leu Thr Ala Ala Cys Thr Gly Arg Thr Gly Asn Trp Pro Asn Phe Asp
165 170 175
Ile Ala Gln Asp Arg Ile Ser Phe Leu Ser Ser Pro Asn Pro Arg His
180 185 190
Ala Gly Asn Arg Ser Gln Glu Ala Phe Leu Asp Asp Leu Val Gln Glu
195 200 205
Phe Glu Asp Arg Ile Glu Ser Leu Gly Pro Asp Thr Ile Ala Ala Phe
210 215 220
Leu Ala Glu Pro Ile Leu Ala Ser Gly Gly Val Ile Ile Pro Pro Ala
225 230 235 240
Gly Tyr His Ala Arg Phe Lys Ala Ile Cys Glu Lys His Asp Ile Leu
245 250 255
Tyr Ile Ser Asp Glu Val Val Thr Gly Phe Gly Arg Cys Gly Glu Trp
260 265 270
Phe Ala Ser Glu Lys Val Phe Gly Val Val Pro Asp Ile Ile Thr Phe
275 280 285
Ala Lys Gly Val Thr Ser Gly Tyr Val Pro Leu Gly Gly Leu Ala Ile
290 295 300
Ser Glu Ala Val Leu Ala Arg Ile Ser Gly Glu Asn Ala Lys Gly Ser
305 310 315 320
Trp Phe Thr Asn Gly Tyr Thr Tyr Ser Asn Gln Pro Val Ala Cys Ala
325 330 335
Ala Ala Leu Ala Asn Ile Glu Leu Met Glu Arg Glu Gly Ile Val Asp
340 345 350
Gln Ala Arg Glu Met Ala Asp Tyr Phe Ala Ala Ala Leu Ala Ser Leu
355 360 365
Arg Asp Leu Pro Gly Val Ala Glu Thr Arg Ser Val Gly Leu Val Gly
370 375 380
Cys Val Gln Cys Leu Leu Asp Pro Thr Arg Ala Asp Gly Thr Ala Glu
385 390 395 400
Asp Lys Ala Phe Thr Leu Lys Ile Asp Glu Arg Cys Phe Glu Leu Gly
405 410 415
Leu Ile Val Arg Pro Leu Gly Asp Leu Cys Val Ile Ser Pro Pro Leu
420 425 430
Ile Ile Ser Arg Ala Gln Ile Asp Glu Met Val Ala Ile Met Arg Gln
435 440 445
Ala Ile Thr Glu Val Ser Ala Ala His Gly Leu Thr Ala Lys Glu Pro
450 455 460
Ala Ala Val
465
<210> 12
<211> 459
<212> PRT
<213> 大腸桿菌
<400> 12
Met Asn Arg Leu Pro Ser Ser Ala Ser Ala Leu Ala Cys Ser Ala His
1 5 10 15
Ala Leu Asn Leu Ile Glu Lys Arg Thr Leu Asp His Glu Glu Met Lys
20 25 30
Ala Leu Asn Arg Glu Val Ile Glu Tyr Phe Lys Glu His Val Asn Pro
35 40 45
Gly Phe Leu Glu Tyr Arg Lys Ser Val Thr Ala Gly Gly Asp Tyr Gly
50 55 60
Ala Val Glu Trp Gln Ala Gly Ser Leu Asn Thr Leu Val Asp Thr Gln
65 70 75 80
Gly Gln Glu Phe Ile Asp Cys Leu Gly Gly Phe Gly Ile Phe Asn Val
85 90 95
Gly His Arg Asn Pro Val Val Val Ser Ala Val Gln Asn Gln Leu Ala
100 105 110
Lys Gln Pro Leu His Ser Gln Glu Leu Leu Asp Pro Leu Arg Ala Met
115 120 125
Leu Ala Lys Thr Leu Ala Ala Leu Thr Pro Gly Lys Leu Lys Tyr Ser
130 135 140
Phe Phe Cys Asn Ser Gly Thr Glu Ser Val Glu Ala Ala Leu Lys Leu
145 150 155 160
Ala Lys Ala Tyr Gln Ser Pro Arg Gly Lys Phe Thr Phe Ile Ala Thr
165 170 175
Ser Gly Ala Phe His Gly Lys Ser Leu Gly Ala Leu Ser Ala Thr Ala
180 185 190
Lys Ser Thr Phe Arg Lys Pro Phe Met Pro Leu Leu Pro Gly Phe Arg
195 200 205
His Val Pro Phe Gly Asn Ile Glu Ala Met Arg Thr Ala Leu Asn Glu
210 215 220
Cys Lys Lys Thr Gly Asp Asp Val Ala Ala Val Ile Leu Glu Pro Ile
225 230 235 240
Gln Gly Glu Gly Gly Val Ile Leu Pro Pro Pro Gly Tyr Leu Thr Ala
245 250 255
Val Arg Lys Leu Cys Asp Glu Phe Gly Ala Leu Met Ile Leu Asp Glu
260 265 270
Val Gln Thr Gly Met Gly Arg Thr Gly Lys Met Phe Ala Cys Glu His
275 280 285
Glu Asn Val Gln Pro Asp Ile Leu Cys Leu Ala Lys Ala Leu Gly Gly
290 295 300
Gly Val Met Pro Ile Gly Ala Thr Ile Ala Thr Glu Glu Val Phe Ser
305 310 315 320
Val Leu Phe Asp Asn Pro Phe Leu His Thr Thr Thr Phe Gly Gly Asn
325 330 335
Pro Leu Ala Cys Ala Ala Ala Leu Ala Thr Ile Asn Val Leu Leu Glu
340 345 350
Gln Asn Leu Pro Ala Gln Ala Glu Gln Lys Gly Asp Met Leu Leu Asp
355 360 365
Gly Phe Arg Gln Leu Ala Arg Glu Tyr Pro Asp Leu Val Gln Glu Ala
370 375 380
Arg Gly Lys Gly Met Leu Met Ala Ile Glu Phe Val Asp Asn Glu Ile
385 390 395 400
Gly Tyr Asn Phe Ala Ser Glu Met Phe Arg Gln Arg Val Leu Val Ala
405 410 415
Gly Thr Leu Asn Asn Ala Lys Thr Ile Arg Ile Glu Pro Pro Leu Thr
420 425 430
Leu Thr Ile Glu Gln Cys Glu Leu Val Ile Lys Ala Ala Arg Lys Ala
435 440 445
Leu Ala Ala Met Arg Val Ser Val Glu Glu Ala
450 455
<210> 13
<211> 453
<212> PRT
<213> 河流孤菌
<400> 13
Met Asn Lys Pro Gln Ser Trp Glu Ala Arg Ala Glu Thr Tyr Ser Leu
1 5 10 15
Tyr Gly Phe Thr Asp Met Pro Ser Leu His Gln Arg Gly Thr Val Val
20 25 30
Val Thr His Gly Glu Gly Pro Tyr Ile Val Asp Val Asn Gly Arg Arg
35 40 45
Tyr Leu Asp Ala Asn Ser Gly Leu Trp Asn Met Val Ala Gly Phe Asp
50 55 60
His Lys Gly Leu Ile Asp Ala Ala Lys Ala Gln Tyr Glu Arg Phe Pro
65 70 75 80
Gly Tyr His Ala Phe Phe Gly Arg Met Ser Asp Gln Thr Val Met Leu
85 90 95
Ser Glu Lys Leu Val Glu Val Ser Pro Phe Asp Ser Gly Arg Val Phe
100 105 110
Tyr Thr Asn Ser Gly Ser Glu Ala Asn Asp Thr Met Val Lys Met Leu
115 120 125
Trp Phe Leu His Ala Ala Glu Gly Lys Pro Gln Lys Arg Lys Ile Leu
130 135 140
Thr Arg Trp Asn Ala Tyr His Gly Val Thr Ala Val Ser Ala Ser Met
145 150 155 160
Thr Gly Lys Pro Tyr Asn Ser Val Phe Gly Leu Pro Leu Pro Gly Phe
165 170 175
Val His Leu Thr Cys Pro His Tyr Trp Arg Tyr Gly Glu Glu Gly Glu
180 185 190
Thr Glu Glu Gln Phe Val Ala Arg Leu Ala Arg Glu Leu Glu Glu Thr
195 200 205
Ile Gln Arg Glu Gly Ala Asp Thr Ile Ala Gly Phe Phe Ala Glu Pro
210 215 220
Val Met Gly Ala Gly Gly Val Ile Pro Pro Ala Lys Gly Tyr Phe Gln
225 230 235 240
Ala Ile Leu Pro Ile Leu Arg Lys Tyr Asp Ile Pro Val Ile Ser Asp
245 250 255
Glu Val Ile Cys Gly Phe Gly Arg Thr Gly Asn Thr Trp Gly Cys Val
260 265 270
Thr Tyr Asp Phe Thr Pro Asp Ala Ile Ile Ser Ser Lys Asn Leu Thr
275 280 285
Ala Gly Phe Phe Pro Met Gly Ala Val Ile Leu Gly Pro Glu Leu Ser
290 295 300
Lys Arg Leu Glu Thr Ala Ile Glu Ala Ile Glu Glu Phe Pro His Gly
305 310 315 320
Phe Thr Ala Ser Gly His Pro Val Gly Cys Ala Ile Ala Leu Lys Ala
325 330 335
Ile Asp Val Val Met Asn Glu Gly Leu Ala Glu Asn Val Arg Arg Leu
340 345 350
Ala Pro Arg Phe Glu Glu Arg Leu Lys His Ile Ala Glu Arg Pro Asn
355 360 365
Ile Gly Glu Tyr Arg Gly Ile Gly Phe Met Trp Ala Leu Glu Ala Val
370 375 380
Lys Asp Lys Ala Ser Lys Thr Pro Phe Asp Gly Asn Leu Ser Val Ser
385 390 395 400
Glu Arg Ile Ala Asn Thr Cys Thr Asp Leu Gly Leu Ile Cys Arg Pro
405 410 415
Leu Gly Gln Ser Val Val Leu Cys Pro Pro Phe Ile Leu Thr Glu Ala
420 425 430
Gln Met Asp Glu Met Phe Asp Lys Leu Glu Lys Ala Leu Asp Lys Val
435 440 445
Phe Ala Glu Val Ala
450
<210> 14
<211> 224
<212> PRT
<213> 枯草芽孢桿菌
<400> 14
Met Lys Ile Tyr Gly Ile Tyr Met Asp Arg Pro Leu Ser Gln Glu Glu
1 5 10 15
Asn Glu Arg Phe Met Ser Phe Ile Ser Pro Glu Lys Arg Glu Lys Cys
20 25 30
Arg Arg Phe Tyr His Lys Glu Asp Ala His Arg Thr Leu Leu Gly Asp
35 40 45
Val Leu Val Arg Ser Val Ile Ser Arg Gln Tyr Gln Leu Asp Lys Ser
50 55 60
Asp Ile Arg Phe Ser Thr Gln Glu Tyr Gly Lys Pro Cys Ile Pro Asp
65 70 75 80
Leu Pro Asp Ala His Phe Asn Ile Ser His Ser Gly Arg Trp Val Ile
85 90 95
Cys Ala Phe Asp Ser Gln Pro Ile Gly Ile Asp Ile Glu Lys Thr Lys
100 105 110
Pro Ile Ser Leu Glu Ile Ala Lys Arg Phe Phe Ser Lys Thr Glu Tyr
115 120 125
Ser Asp Leu Leu Ala Lys Asp Lys Asp Glu Gln Thr Asp Tyr Phe Tyr
130 135 140
His Leu Trp Ser Met Lys Glu Ser Phe Ile Lys Gln Glu Gly Lys Gly
145 150 155 160
Leu Ser Leu Pro Leu Asp Ser Phe Ser Val Arg Leu His Gln Asp Gly
165 170 175
Gln Val Ser Ile Glu Leu Pro Asp Ser His Ser Pro Cys Tyr Ile Lys
180 185 190
Thr Tyr Glu Val Asp Pro Gly Tyr Lys Met Ala Val Cys Ala Ala His
195 200 205
Pro Asp Phe Pro Glu Asp Ile Thr Met Val Ser Tyr Glu Glu Leu Leu
210 215 220
<210> 15
<211> 222
<212> PRT
<213> 諾卡氏菌屬sp. NRRL 5646
<400> 15
Met Ile Glu Thr Ile Leu Pro Ala Gly Val Glu Ser Ala Glu Leu Leu
1 5 10 15
Glu Tyr Pro Glu Asp Leu Lys Ala His Pro Ala Glu Glu His Leu Ile
20 25 30
Ala Lys Ser Val Glu Lys Arg Arg Arg Asp Phe Ile Gly Ala Arg His
35 40 45
Cys Ala Arg Leu Ala Leu Ala Glu Leu Gly Glu Pro Pro Val Ala Ile
50 55 60
Gly Lys Gly Glu Arg Gly Ala Pro Ile Trp Pro Arg Gly Val Val Gly
65 70 75 80
Ser Leu Thr His Cys Asp Gly Tyr Arg Ala Ala Ala Val Ala His Lys
85 90 95
Met Arg Phe Arg Ser Ile Gly Ile Asp Ala Glu Pro His Ala Thr Leu
100 105 110
Pro Glu Gly Val Leu Asp Ser Val Ser Leu Pro Pro Glu Arg Glu Trp
115 120 125
Leu Lys Thr Thr Asp Ser Ala Leu His Leu Asp Arg Leu Leu Phe Cys
130 135 140
Ala Lys Glu Ala Thr Tyr Lys Ala Trp Trp Pro Leu Thr Ala Arg Trp
145 150 155 160
Leu Gly Phe Glu Glu Ala His Ile Thr Phe Glu Ile Glu Asp Gly Ser
165 170 175
Ala Asp Ser Gly Asn Gly Thr Phe His Ser Glu Leu Leu Val Pro Gly
180 185 190
Gln Thr Asn Asp Gly Gly Thr Pro Leu Leu Ser Phe Asp Gly Arg Trp
195 200 205
Leu Ile Ala Asp Gly Phe Ile Leu Thr Ala Ile Ala Tyr Ala
210 215 220
<210> 16
<211> 418
<212> PRT
<213> 單胞菌屬sp. JS666
<400> 16
Met Ser Glu Ala Ile Val Val Asn Asn Gln Asn Asp Gln Ser Arg Ala
1 5 10 15
Tyr Ala Ile Pro Leu Glu Asp Ile Asp Val Ser Asn Pro Glu Leu Phe
20 25 30
Arg Asp Asn Thr Met Trp Gly Tyr Phe Glu Arg Leu Arg Arg Glu Asp
35 40 45
Pro Val His Tyr Cys Lys Asp Ser Leu Phe Gly Pro Tyr Trp Ser Val
50 55 60
Thr Lys Phe Lys Asp Ile Met Gln Val Glu Thr His Pro Glu Ile Phe
65 70 75 80
Ser Ser Glu Gly Asn Ile Thr Ile Met Glu Ser Asn Ala Ala Val Thr
85 90 95
Leu Pro Met Phe Ile Ala Met Asp Pro Pro Lys His Asp Val Gln Arg
100 105 110
Met Ala Val Ser Pro Ile Val Ala Pro Glu Asn Leu Ala Lys Leu Glu
115 120 125
Gly Leu Ile Arg Glu Arg Thr Gly Arg Ala Leu Asp Gly Leu Pro Ile
130 135 140
Asn Glu Thr Phe Asp Trp Val Lys Leu Val Ser Ile Asn Leu Thr Thr
145 150 155 160
Gln Met Leu Ala Thr Leu Phe Asp Phe Pro Trp Glu Asp Arg Ala Lys
165 170 175
Leu Thr Arg Trp Ser Asp Val Ala Thr Ala Leu Val Gly Thr Gly Ile
180 185 190
Ile Asp Ser Glu Glu Gln Arg Met Glu Glu Leu Lys Gly Cys Val Gln
195 200 205
Tyr Met Thr Arg Leu Trp Asn Glu Arg Val Asn Val Pro Pro Gly Asn
210 215 220
Asp Leu Ile Ser Met Met Ala His Thr Glu Ser Met Arg Asn Met Thr
225 230 235 240
Pro Glu Glu Phe Leu Gly Asn Leu Ile Leu Leu Ile Val Gly Gly Asn
245 250 255
Asp Thr Thr Arg Asn Ser Met Thr Gly Gly Val Leu Ala Leu Asn Glu
260 265 270
Asn Pro Asp Glu Tyr Arg Lys Leu Cys Ala Asn Pro Ala Leu Ile Ala
275 280 285
Ser Met Val Pro Glu Ile Val Arg Trp Gln Thr Pro Leu Ala His Met
290 295 300
Arg Arg Thr Ala Leu Gln Asp Thr Glu Leu Gly Gly Lys Ser Ile Arg
305 310 315 320
Lys Gly Asp Lys Val Ile Met Trp Tyr Val Ser Gly Asn Arg Asp Pro
325 330 335
Glu Ala Ile Glu Asn Pro Asp Ala Phe Ile Ile Asp Arg Ala Lys Pro
340 345 350
Arg His His Leu Ser Phe Gly Phe Gly Ile His Arg Cys Val Gly Asn
355 360 365
Arg Leu Ala Glu Leu Gln Leu Arg Ile Val Trp Glu Glu Leu Leu Lys
370 375 380
Arg Trp Pro Asn Pro Gly Gln Ile Glu Val Val Gly Ala Pro Glu Arg
385 390 395 400
Val Leu Ser Pro Phe Val Lys Gly Tyr Glu Ser Leu Pro Val Arg Ile
405 410 415
Asn Ala
<210> 17
<211> 420
<212> PRT
<213> 分枝桿菌屬sp. HXN-1500
<400> 17
Met Thr Glu Met Thr Val Ala Ala Ser Asp Ala Thr Asn Ala Ala Tyr
1 5 10 15
Gly Met Ala Leu Glu Asp Ile Asp Val Ser Asn Pro Val Leu Phe Arg
20 25 30
Asp Asn Thr Trp His Pro Tyr Phe Lys Arg Leu Arg Glu Glu Asp Pro
35 40 45
Val His Tyr Cys Lys Ser Ser Met Phe Gly Pro Tyr Trp Ser Val Thr
50 55 60
Lys Tyr Arg Asp Ile Met Ala Val Glu Thr Asn Pro Lys Val Phe Ser
65 70 75 80
Ser Glu Ala Lys Ser Gly Gly Ile Thr Ile Met Asp Asp Asn Ala Ala
85 90 95
Ala Ser Leu Pro Met Phe Ile Ala Met Asp Pro Pro Lys His Asp Val
100 105 110
Gln Arg Lys Thr Val Ser Pro Ile Val Ala Pro Glu Asn Leu Ala Thr
115 120 125
Met Glu Ser Val Ile Arg Gln Arg Thr Ala Asp Leu Leu Asp Gly Leu
130 135 140
Pro Ile Asn Glu Glu Phe Asp Trp Val His Arg Val Ser Ile Glu Leu
145 150 155 160
Thr Thr Lys Met Leu Ala Thr Leu Phe Asp Phe Pro Trp Asp Asp Arg
165 170 175
Ala Lys Leu Thr Arg Trp Ser Asp Val Thr Thr Ala Leu Pro Gly Gly
180 185 190
Gly Ile Ile Asp Ser Glu Glu Gln Arg Met Ala Glu Leu Met Glu Cys
195 200 205
Ala Thr Tyr Phe Thr Glu Leu Trp Asn Gln Arg Val Asn Ala Glu Pro
210 215 220
Lys Asn Asp Leu Ile Ser Met Met Ala His Ser Glu Ser Thr Arg His
225 230 235 240
Met Ala Pro Glu Glu Tyr Leu Gly Asn Ile Val Leu Leu Ile Val Gly
245 250 255
Gly Asn Asp Thr Thr Arg Asn Ser Met Thr Gly Gly Val Leu Ala Leu
260 265 270
Asn Glu Phe Pro Asp Glu Tyr Arg Lys Leu Ser Ala Asn Pro Ala Leu
275 280 285
Ile Ser Ser Met Val Ser Glu Ile Ile Arg Trp Gln Thr Pro Leu Ser
290 295 300
His Met Arg Arg Thr Ala Leu Glu Asp Ile Glu Phe Gly Gly Lys His
305 310 315 320
Ile Arg Gln Gly Asp Lys Val Val Met Trp Tyr Val Ser Gly Asn Arg
325 330 335
Asp Pro Glu Ala Ile Asp Asn Pro Asp Thr Phe Ile Ile Asp Arg Ala
340 345 350
Lys Pro Arg Gln His Leu Ser Phe Gly Phe Gly Ile His Arg Cys Val
355 360 365
Gly Asn Arg Leu Ala Glu Leu Gln Leu Asn Ile Leu Trp Glu Glu Ile
370 375 380
Leu Lys Arg Trp Pro Asp Pro Leu Gln Ile Gln Val Leu Gln Glu Pro
385 390 395 400
Thr Arg Val Leu Ser Pro Phe Val Lys Gly Tyr Glu Ser Leu Pro Val
405 410 415
Arg Ile Asn Ala
420
<210> 18
<211> 420
<212> PRT
<213> 結核分枝桿菌
<400> 18
Met Thr Glu Met Thr Val Ala Ala Asn Asp Ala Thr Asn Ala Ala Tyr
1 5 10 15
Gly Met Ala Leu Glu Asp Ile Asp Val Ser Asn Pro Val Leu Phe Arg
20 25 30
Asp Asn Thr Trp His Pro Tyr Phe Lys Arg Leu Arg Glu Glu Asp Pro
35 40 45
Val His Tyr Cys Lys Ser Ser Met Phe Gly Pro Tyr Trp Ser Val Thr
50 55 60
Lys Tyr Arg Asp Ile Met Ala Val Glu Thr Asn Pro Lys Val Phe Ser
65 70 75 80
Ser Glu Ala Lys Ser Gly Gly Ile Thr Ile Met Asp Asp Asn Ala Ala
85 90 95
Ala Ser Leu Pro Met Phe Ile Ala Met Asp Pro Pro Lys His Asp Val
100 105 110
Gln Arg Lys Thr Val Ser Pro Ile Val Ala Pro Glu Asn Leu Ala Thr
115 120 125
Met Glu Ser Val Ile Arg Gln Arg Thr Ala Asp Leu Leu Asp Gly Leu
130 135 140
Pro Ile Asn Glu Glu Phe Asp Trp Val His Arg Val Ser Ile Asp Leu
145 150 155 160
Thr Thr Lys Met Leu Ala Thr Leu Phe Asp Phe Pro Trp Asp Asp Arg
165 170 175
Ala Lys Leu Thr Arg Trp Ser Asp Val Thr Thr Ala Leu Pro Gly Gly
180 185 190
Gly Ile Ile Asp Ser Glu Glu Gln Arg Met Ala Glu Leu Met Glu Cys
195 200 205
Ala Thr Tyr Phe Thr Glu Leu Trp Asn Gln Arg Val Asn Ala Glu Pro
210 215 220
Lys Asn Asp Leu Ile Ser Met Met Ala His Ser Glu Ser Thr Arg His
225 230 235 240
Met Ala Pro Glu Glu Tyr Leu Gly Asn Ile Val Leu Leu Ile Val Gly
245 250 255
Gly Asn Asp Thr Thr Arg Asn Ser Met Thr Gly Gly Val Leu Ala Leu
260 265 270
Asn Glu Phe Pro Asp Glu Tyr Arg Lys Leu Ser Ala Asn Pro Ala Leu
275 280 285
Ile Ser Ser Met Val Ser Glu Ile Ile Arg Trp Gln Thr Pro Leu Ser
290 295 300
His Met Arg Arg Thr Ala Leu Glu Asp Ile Glu Phe Gly Gly Lys His
305 310 315 320
Ile Arg Gln Gly Asp Lys Val Val Met Trp Tyr Val Ser Gly Asn Arg
325 330 335
Asp Pro Glu Ala Ile Asp Asn Pro Asp Thr Phe Ile Ile Asp Arg Ala
340 345 350
Lys Pro Arg Gln His Leu Ser Phe Gly Phe Gly Ile His Arg Cys Val
355 360 365
Gly Asn Arg Leu Ala Glu Leu Gln Leu Asn Ile Leu Trp Glu Glu Ile
370 375 380
Leu Lys Arg Trp Pro Asp Pro Leu Gln Ile Gln Val Leu Gln Glu Pro
385 390 395 400
Thr Arg Val Leu Ser Pro Phe Val Lys Gly Tyr Glu Ser Leu Pro Val
405 410 415
Arg Ile Asn Ala
420
<210> 19
<211> 405
<212> PRT
<213> 單胞菌屬sp. JS666
<400> 19
Met Ser Glu Thr Val Ile Ile Ala Gly Ala Gly Gln Ala Ala Gly Gln
1 5 10 15
Ala Val Ala Ser Leu Arg Gln Glu Gly Phe Asp Gly Arg Ile Val Leu
20 25 30
Val Gly Ala Glu Pro Val Leu Pro Tyr Gln Arg Pro Pro Leu Ser Lys
35 40 45
Ala Phe Leu Ala Gly Thr Leu Pro Leu Glu Arg Leu Phe Leu Lys Pro
50 55 60
Pro Ala Phe Tyr Glu Gln Ala Arg Val Asp Thr Leu Leu Gly Val Ala
65 70 75 80
Val Thr Glu Leu Asp Ala Ala Arg Arg Gln Val Arg Leu Asp Asp Gly
85 90 95
Arg Glu Leu Ala Phe Asp His Leu Leu Leu Ala Thr Gly Gly Arg Ala
100 105 110
Arg Arg Leu Asp Cys Pro Gly Ala Asp His Pro Arg Leu His Tyr Leu
115 120 125
Arg Thr Val Ala Asp Val Asp Gly Ile Arg Ala Ala Leu Arg Pro Gly
130 135 140
Ala Arg Leu Val Leu Ile Gly Gly Gly Tyr Val Gly Leu Glu Ile Ala
145 150 155 160
Ala Val Ala Ala Lys Leu Gly Leu Ala Val Thr Val Leu Glu Ala Ala
165 170 175
Pro Thr Val Leu Ala Arg Val Thr Cys Pro Ala Val Ala Arg Phe Phe
180 185 190
Glu Ser Val His Arg Gln Ala Gly Val Thr Ile Arg Cys Ala Thr Thr
195 200 205
Val Ser Gly Ile Glu Gly Asp Ala Ser Leu Ala Arg Val Val Thr Gly
210 215 220
Asp Gly Glu Arg Ile Asp Ala Asp Leu Val Ile Ala Gly Ile Gly Leu
225 230 235 240
Leu Pro Asn Val Glu Leu Ala Gln Ala Ala Gly Leu Val Cys Asp Asn
245 250 255
Gly Ile Val Val Asp Glu Glu Cys Arg Thr Ser Val Pro Gly Ile Phe
260 265 270
Ala Ala Gly Asp Cys Thr Gln His Pro Asn Ala Ile Tyr Asp Ser Arg
275 280 285
Leu Arg Leu Glu Ser Val His Asn Ala Ile Glu Gln Gly Lys Thr Ala
290 295 300
Ala Ala Ala Met Cys Gly Lys Ala Arg Pro Tyr Arg Gln Val Pro Trp
305 310 315 320
Phe Trp Ser Asp Gln Tyr Asp Leu Lys Leu Gln Thr Ala Gly Leu Asn
325 330 335
Arg Gly Tyr Asp Gln Val Val Met Arg Gly Ser Thr Asp Asn Arg Ser
340 345 350
Phe Ala Ala Phe Tyr Leu Arg Asp Gly Arg Leu Leu Ala Val Asp Ala
355 360 365
Val Asn Arg Pro Val Glu Phe Met Val Ala Lys Ala Leu Ile Ala Asn
370 375 380
Arg Thr Val Ile Ala Pro Glu Arg Leu Ala Asp Glu Arg Ile Ala Ala
385 390 395 400
Lys Asp Leu Ala Gly
405
<210> 20
<211> 424
<212> PRT
<213> 分枝桿菌屬sp. HXN-1500
<400> 20
Met Ile His Thr Gly Val Thr Glu Ala Val Val Val Val Gly Ala Gly
1 5 10 15
Gln Ala Gly Ala Gln Thr Val Thr Ser Leu Arg Gln Arg Gly Phe Glu
20 25 30
Gly Gln Ile Thr Leu Leu Gly Asp Glu Pro Ala Leu Pro Tyr Gln Arg
35 40 45
Pro Pro Leu Ser Lys Ala Phe Leu Ala Gly Thr Leu Pro Leu Asp Arg
50 55 60
Leu Tyr Leu Arg Pro Ala Ala Phe Tyr Gln Gln Ala His Val Asp Val
65 70 75 80
Met Val Asp Thr Gly Val Ser Glu Leu Asp Thr Glu Asn Arg Arg Ile
85 90 95
Arg Leu Thr Asp Gly Arg Ala Ile Ser Phe Asp His Leu Val Leu Ala
100 105 110
Thr Gly Gly Arg Pro Arg Pro Leu Ala Cys Pro Gly Ala Asp His Pro
115 120 125
Arg Val His Tyr Leu Arg Thr Val Thr Asp Val Asp Arg Ile Arg Ser
130 135 140
Gln Phe His Pro Gly Thr Arg Leu Val Leu Val Gly Gly Gly Tyr Ile
145 150 155 160
Gly Leu Glu Ile Ala Ala Val Ala Ala Glu Leu Gly Leu Thr Val Thr
165 170 175
Val Leu Glu Ala Gln Thr Thr Val Leu Ala Arg Val Thr Cys Pro Thr
180 185 190
Val Ala Arg Phe Phe Glu His Thr His Arg Arg Ala Gly Val Thr Ile
195 200 205
Arg Cys Ala Thr Thr Val Thr Arg Ile His Asp Ser Ser Ser Thr Ala
210 215 220
Arg Ile Glu Leu Asp Ser Gly Glu Tyr Ile Asp Ala Asp Leu Val Ile
225 230 235 240
Val Gly Ile Gly Leu Leu Pro Asn Val Asp Leu Ala Ser Ala Ala Gly
245 250 255
Leu Thr Cys Glu Ser Gly Ile Val Val Asp Ser Arg Cys Gln Thr Ser
260 265 270
Ala Pro Gly Ile Tyr Ala Ala Gly Asp Cys Thr Gln Tyr Pro Ser Pro
275 280 285
Ile Tyr Gly Arg Pro Leu His Leu Glu Ser Val His Asn Ala Ile Glu
290 295 300
Gln Ala Lys Thr Ala Ala Ala Ala Ile Leu Gly Arg Asp Glu Pro Phe
305 310 315 320
Arg Gln Val Pro Trp Phe Trp Ser Asp Gln Tyr Asn Ile Lys Leu Gln
325 330 335
Thr Ala Gly Val Asn Glu Gly Tyr Asp Asp Val Ile Ile Arg Gly Asp
340 345 350
Pro Ala Ser Ala Ser Phe Ala Ala Phe Tyr Leu Arg Ala Gly Lys Leu
355 360 365
Leu Ala Val Asp Ala Ile Asn Arg Pro Arg Glu Phe Met Ala Ser Lys
370 375 380
Thr Leu Ile Ala Glu Arg Ala Glu Val Asp Pro Thr Gln Leu Ala Asp
385 390 395 400
Glu Ser Leu Pro Pro Thr Ala Leu Ala Ala Ala Val Asn Gly Pro Thr
405 410 415
Arg Ala Thr Ser Pro Thr Ser Leu
420
<210> 21
<211> 106
<212> PRT
<213> 單胞菌屬sp. JS666
<400> 21
Met Thr Lys Val Thr Phe Ile Glu His Asn Gly Thr Val Arg Asn Val
1 5 10 15
Asp Val Asp Asp Gly Leu Ser Val Met Glu Ala Ala Val Asn Asn Leu
20 25 30
Val Pro Gly Ile Asp Gly Asp Cys Gly Gly Ala Cys Ala Cys Ala Thr
35 40 45
Cys His Val His Ile Asp Ala Ala Trp Leu Asp Lys Leu Pro Pro Met
50 55 60
Glu Ala Met Glu Lys Ser Met Leu Glu Phe Ala Glu Gly Arg Asn Glu
65 70 75 80
Ser Ser Arg Leu Gly Cys Gln Ile Lys Leu Ser Pro Ala Leu Asp Gly
85 90 95
Ile Val Val Arg Thr Pro Leu Gly Gln His
100 105
<210> 22
<211> 106
<212> PRT
<213> 分枝桿菌屬sp. HXN-1500
<400> 22
Met Pro Lys Ile Thr Tyr Ile Asp Tyr Thr Gly Thr Ser Arg Cys Val
1 5 10 15
Asp Ala Glu Asn Gly Met Ser Leu Met Glu Ile Ala Ile Asn Asn Asn
20 25 30
Val Pro Gly Ile Asp Gly Asp Cys Gly Gly Glu Cys Ala Cys Ala Thr
35 40 45
Cys His Val His Val Asp Ala Asp Trp Leu Asp Lys Leu Pro Pro Ser
50 55 60
Ser Asp Gln Glu Val Ser Met Leu Glu Phe Cys Asp Gly Val Asp His
65 70 75 80
Thr Ser Arg Leu Gly Cys Gln Ile Lys Ile Cys Pro Thr Leu Asp Gly
85 90 95
Ile Val Val Arg Thr Pro Ala Ala Gln His
100 105
<210> 23
<211> 395
<212> PRT
<213> 枯草芽孢桿菌
<400> 23
Met Thr Ile Ala Ser Ser Thr Ala Ser Ser Glu Phe Leu Lys Asn Pro
1 5 10 15
Tyr Ser Phe Tyr Asp Thr Leu Arg Ala Val His Pro Ile Tyr Lys Gly
20 25 30
Ser Phe Leu Lys Tyr Pro Gly Trp Tyr Val Thr Gly Tyr Glu Glu Thr
35 40 45
Ala Ala Ile Leu Lys Asp Ala Arg Phe Lys Val Arg Thr Pro Leu Pro
50 55 60
Glu Ser Ser Thr Lys Tyr Gln Asp Leu Ser His Val Gln Asn Gln Met
65 70 75 80
Met Leu Phe Gln Asn Gln Pro Asp His Arg Arg Leu Arg Thr Leu Ala
85 90 95
Ser Gly Ala Phe Thr Pro Arg Thr Thr Glu Ser Tyr Gln Pro Tyr Ile
100 105 110
Ile Glu Thr Val His His Leu Leu Asp Gln Val Gln Gly Lys Lys Lys
115 120 125
Met Glu Val Ile Ser Asp Phe Ala Phe Pro Leu Ala Ser Phe Val Ile
130 135 140
Ala Asn Ile Ile Gly Val Pro Glu Glu Asp Arg Glu Gln Leu Lys Glu
145 150 155 160
Trp Ala Ala Ser Leu Ile Gln Thr Ile Asp Phe Thr Arg Ser Arg Lys
165 170 175
Ala Leu Thr Glu Gly Asn Ile Met Ala Val Gln Ala Met Ala Tyr Phe
180 185 190
Lys Glu Leu Ile Gln Lys Arg Lys Arg His Pro Gln Gln Asp Met Ile
195 200 205
Ser Met Leu Leu Lys Gly Arg Glu Lys Asp Lys Leu Thr Glu Glu Glu
210 215 220
Ala Ala Ser Thr Cys Ile Leu Leu Ala Ile Ala Gly His Glu Thr Thr
225 230 235 240
Val Asn Leu Ile Ser Asn Ser Val Leu Cys Leu Leu Gln His Pro Glu
245 250 255
Gln Leu Leu Lys Leu Arg Glu Asn Pro Asp Leu Ile Gly Thr Ala Val
260 265 270
Glu Glu Cys Leu Arg Tyr Glu Ser Pro Thr Gln Met Thr Ala Arg Val
275 280 285
Ala Ser Glu Asp Ile Asp Ile Cys Gly Val Thr Ile Arg Gln Gly Glu
290 295 300
Gln Val Tyr Leu Leu Leu Gly Ala Ala Asn Arg Asp Pro Ser Ile Phe
305 310 315 320
Thr Asn Pro Asp Val Phe Asp Ile Thr Arg Ser Pro Asn Pro His Leu
325 330 335
Ser Phe Gly His Gly His His Val Cys Leu Gly Ser Ser Leu Ala Arg
340 345 350
Leu Glu Ala Gln Ile Ala Ile Asn Thr Leu Leu Gln Arg Met Pro Ser
355 360 365
Leu Asn Leu Ala Asp Phe Glu Trp Arg Tyr Arg Pro Leu Phe Gly Phe
370 375 380
Arg Ala Leu Glu Glu Leu Pro Val Thr Phe Glu
385 390 395
- 還沒有人留言評論。精彩留言會獲得點贊!