用于特異性轉變靶向DNA序列的核酸堿基的基因組序列的修飾方法及其使用的分子復合體與流程
本發明涉及基因組序列的修飾方法、及其使用的核酸序列識別模塊與核酸堿基轉變酶的復合體,該基因組序列的修飾方法能夠不伴隨DNA的雙鏈的切割(無切割或者切割一根鏈)也不進行外源DNA片段的插入,而修飾基因組的特定區域內的核酸堿基。
背景技術:
近年來,作為在各種生物種類中修飾靶基因、基因組區域的技術,基因組編輯受到關注。以往,作為基因組編輯的方法,提出了利用將具有序列非依賴的DNA切割能力的分子與具有序列識別能力的分子組合而成的人工核酸酶的方法(非專利文獻1)。
已有報告例如,使用由鋅指DNA結合結構域與非特異性DNA切割結構域連接而成的鋅指核酸酶(ZFN),在作為宿主的植物細胞或昆蟲細胞中,進行DNA中的靶基因座的重組的方法(專利文獻1);使用由作為植物病原菌黃單胞菌屬具有的DNA結合模塊的轉錄激活子樣(TAL)效應子,與DNA核酸內切酶連接而成的TALEN,在特定的核苷酸序列內或其相鄰部位進行切割、修飾靶基因的方法(專利文獻2);或者,利用由在真細菌和古細菌具有的獲得性免疫系統中起作用的DNA序列CRISPR(Clustered Regularly interspaced short palindromic repeats),和與CRISPR一起具有重要的作用的核酸酶Cas(CRISPR-associated)蛋白家族組合而成的CRISPR-Cas9系統的方法(專利文獻3)。進一步,也報告了使用PPR蛋白質與核酸酶連接而成的人工核酸酶,在該特定序列的附近切割靶基因的方法(專利文獻4),其中,PPR蛋白質構建為使得利用包括35個氨基酸的并識別1個核酸堿基的PPR基序的連續,來識別特定的核苷酸序列。
現有技術文獻
專利文獻
專利文獻1:日本專利第4968498號公報
專利文獻2:日本特表2013-513389號公報
專利文獻3:日本特表2010-519929號公報
專利文獻4:日本特開2013-128413號公報
非專利文獻
非專利文獻1:Kelvin M Esvelt,Harris H Wang(2013)Genome-scale engineering for systems and synthetic biology,Molecular Systems Biology 9:641
技術實現要素:
發明要解決的問題
迄今為止提出的基因組編輯技術,基本上是以DNA雙鏈切割(double-DNA breaks:DSB)為前提的,但由于伴隨意想不到的基因組修飾,因此有強細胞毒性、染色體重排等副作用,存在諸如以下共通問題:在基因治療中的可靠性受損、通過核苷酸修飾的細胞存活數極少、在靈長類卵細胞、單細胞微生物中基因修飾本身難以進行。
因此,本發明的目的在于,提供一種不DSB、不伴隨外源DNA片段的插入,即通過不切割雙鏈DNA或者切割一條鏈來修飾基因的特定序列的核酸堿基的新型基因組編輯方法,以及用于其的核酸序列識別模塊及核酸堿基轉變酶的復合體。
解決問題的方法
本發明人等為了解決上述課題反復進行了深入研究,結果構思了不伴隨DSB,采用基于DNA堿基的轉變反應的堿基轉變。雖然已經知曉基于DNA堿基的脫氨基反應的堿基轉變反應本身,但尚未實現用其對DNA的特定的序列進行識別而對任意部位靶向化,并通過DNA堿基的堿基轉變而特異性修飾被靶向化的DNA。
因此,通過使用催化脫氨基反應的脫氨酶作為進行這樣的核酸堿基轉變的酶并將其與具有DNA序列識別能力的分子連接,從而在包含特定的DNA序列的區域中進行基于核酸堿基轉變的基因組序列的修飾。
具體而言,使用CRISPR-Cas系統(CRISPR-突變Cas)進行。即,制作了用于編碼將用于募集Cas蛋白質的RNA(反式-激活crRNA:tracrRNA)連接至包含與待修飾的基因的靶序列互補的序列的、基因組特異性CRISPR-RNA:crRNA(gRNA)而成的RNA分子的DNA,另一方面,制作了由編碼雙鏈DNA的任意一條或兩條鏈的切割能力已失活的突變Cas蛋白質的DNA(dCas)與脫氨酶基因連接而成的DNA,并將這些DNA導入包含待修飾的基因的宿主酵母細胞。結果,成功地在包括靶序列的靶基因的幾百個核苷酸范圍內隨機導入突變。與使用對雙鏈DNA的任意DNA鏈均不切割的雙重突變Cas蛋白質的情況相比,使用切割任意一條鏈的突變Cas蛋白質的情況下,突變導入效率升高。另外,明確了根據切割DNA雙鏈中的哪條不同,突變區域的面積和突變的多樣性也發生變化。此外,通過對靶基因中的多個區域進行靶向化,可以有效地引入突變。即,確認了將已導入DNA的宿主細胞接種于非選擇性培養基,針對隨機選擇的菌落檢查靶基因的序列,結果幾乎針對全部菌落導入了突變。另外,也確認了通過分別對2個以上的靶基因中具有的區域進行靶向化,可同時進行多個部位的基因組編輯。進一步證實:該方法可以同時向二倍體或多倍體基因組的等位基因中引入突變;不僅可以向真核細胞引入突變,而且可以在原核細胞如大腸桿菌中引入突變;不論生物種類如何均可廣泛應用。另外,通過在期望的時期一過性地進行核酸堿基轉變反應,可有效地進行迄今為止效率較低的必需基因的編輯。
本發明人等基于這些見解進一步反復研究,從而完成了本發明。
即,本發明如下所述。
[1]一種修飾雙鏈DNA的靶向位點的方法,其包括:使由核酸堿基轉變酶、和與選擇的雙鏈DNA中的靶核苷酸序列進行特異性結合的核酸序列識別模塊結合而成的復合體,與該雙鏈DNA接觸,不切割該靶向位點中的該雙鏈DNA的至少一條鏈,而使該靶向位點的1個以上的核苷酸缺失或轉變為其它的1個以上的核苷酸,或者向該靶向位點插入1個以上的核苷酸的工序。
[2]根據上述[1]所述的方法,其中,上述核酸序列識別模塊選自:Cas的至少1種DNA切割能力已失活的CRISPR-Cas系統、鋅指基序、TAL效應子及PPR基序。
[3]根據上述[1]所述的方法,其中,上述核酸序列識別模塊為Cas的至少1種DNA切割能力已失活的CRISPR-Cas系統。
[4]根據上述[1]~[3]中任一項所述的方法,其中,使用與不同的靶核苷酸序列分別進行特異性結合的兩種以上的核酸序列識別模塊。
[5]根據上述[4]所述的方法,其中,上述不同的靶核苷酸序列存在于不同的基因內。
[6]根據上述[1]~[5]中任一項所述的方法,其中,上述核酸堿基轉變酶為脫氨酶。
[7]根據上述[6]所述的方法,其中,上述脫氨酶為AID(AICDA)。
[8]根據上述[1]~[7]中任一項所述的方法,其中,所述雙鏈DNA與所述復合物的接觸,通過向具有所述雙鏈DNA的細胞中導入編碼所述復合體的核酸來進行。
[9]根據上述[8]所述的方法,其中,所述細胞是原核生物細胞。
[10]根據上述[8]所述的方法,其中,所述細胞是真核生物細胞。
[11]根據上述[8]所述的方法,其中,所述細胞是微生物細胞。
[12]根據上述[8]所述的方法,其中,所述細胞是植物細胞。
[13]根據上述[8]所述的方法,其中,所述細胞是昆蟲細胞。
[14]根據上述[8]所述的方法,其中,所述細胞是動物細胞。
[15]根據上述[8]所述的方法,其中,所述細胞是脊椎動物細胞。
[16]根據上述[8]所述的方法,其中,所述細胞是哺乳動物細胞。
[17]根據上述[9]~[16]中任一項所述的方法,其中,所述細胞為多倍體細胞,并且對同源染色體上的全部靶向化的等位基因內的位點進行修飾。
[18]根據上述[8]~[17]中任一項所述的方法,其包括:將以能夠控制表達時期的形式包含編碼上述復合物的核酸的表達載體,導入所述細胞的步驟,以及在需要固定雙鏈DNA的靶位點的修飾的時期,誘導該核酸的表達的步驟。
[19]根據上述[18]所述的方法,其中,雙鏈DNA中的靶核苷酸序列存在于對于上述細胞必需的基因內。
[20]核酸修飾酶復合體,該復合體由與雙鏈DNA中的靶核苷酸序列特異性結合的核酸序列識別模塊,和核酸堿基轉變酶結合而成,在靶向位點中,不切割該雙鏈DNA的至少一條鏈,使該靶向位點的1個以上的核苷酸缺失或轉變為其它的1個以上的核苷酸,或者向該靶向位點插入1個以上的核苷酸。
[21]編碼上述[20]所述的核酸修飾酶復合體的核酸。
發明的效果
根據本發明的基因組編輯技術,由于不伴隨外來DNA的插入或雙鏈DNA切割,因此安全性方面優異,即使在常規方法中以基因重組的形式而在生物學上或者法律上存在爭議的情況中,作為解決方案的可能也經常存在。另外,理論上能夠將突變導入的范圍從一個堿基的針點(pin point)至幾百個堿基的范圍進行廣泛設定,也可以應用于利用向迄今為止幾乎不可能進行的特定的限定區域隨機導入突變來進行的局部進化誘導。
附圖說明
[圖1]示出了使用CRISPR-Cas系統的本發明的基因修飾方法的機理的圖。
[圖2]示出了使用芽殖酵母(budding yeast),對由CRISPR-Cas系統與來源于七腮鰻(Petromyzon marinus)的PmCDA1脫氨酶組合而成的本發明的基因修飾方法的效果進行驗證的結果。
[圖3]示出了由使用具有切口酶(nickase)活性的Cas9的D10A突變體的CRISPR-Cas9系統、與脫氨酶和PmCDA1進行組合的情況(nCas9D10A-PmCDA1)下,和使用具有DNA雙鏈裂解能力的常規型Cas9的情況下,進行表達誘導后的細胞存活數的變化的圖。
[圖4]示出了使用源自人的AID脫氨酶,以與dCas9隔著SH3結構域及其結合配體進行結合的方式構建了表達構建體,將構建成的表達構建體與兩種gRNA(以target4及target5的序列為靶)一起導入芽殖酵母的結果的圖。
[圖5]示出了通過使用切割任意DNA單鏈的Cas9,突變導入效率提高的圖。
[圖6]示出了根據是否切割雙鏈DNA的情況、或切割任意DNA一根鏈的情況不同,突變導入區域的大小和頻率發生變化的圖。
[圖7]示出通過對鄰近的2個區域進行靶向,可以實現極高的突變導入效率的圖。
[圖8]示出本發明的基因修飾方法不需要基于標記進行選擇的圖。確定了序列的全部菌落導入了突變。
[圖9]示出根據本發明的基因修飾方法,可以對基因組中多個部位進行同時編輯的圖。上圖示出了各基因的靶向部位的核苷酸序列及氨基酸序列,該核苷酸序列上方的箭頭指示靶核苷酸序列。箭尾或者箭頭的數字表示靶核苷酸序列末端在ORF上的位置。下圖示出了紅色(R)及白色(W)菌落的各個5克隆中的靶向部位的測序結果。顯示序列中在利用白色字體的字母表示的核苷酸中發生了堿基轉變。需要說明的是,對刀豆氨酸的反應性(CanR)而言,R顯示抗性、S顯示敏感性。
[圖10]顯示根據本發明的基因修飾方法,可以同時向二倍體基因組的同源染色體上的兩個等位基因導入突變的圖。圖10A顯示對于ade1基因(上圖)及can1基因的各自的純合突變(homomutation)導入效率,圖10B顯示在紅色菌落中實際導入了純合突變(下圖)。另外,顯示在白色菌落中也發生了雜合突變(heteromutation)(上圖)。
[圖11]顯示根據本發明的基因修飾方法,能夠進行作為原核細胞的大腸桿菌的基因組編輯的圖。圖11A為使用的質粒的模式圖。圖11B顯示可以將galK基因內的區域作為靶標,有效地導入突變(CAA→TAA)。圖11C顯示對于利用非選擇性培養基(none)、含有25μg/ml利福平(rifampicin)(Rif 25)及50μg/ml利福平(Rif 50)的培養基選出的菌落,各2個克隆進行測序分析的結果。確認導入了賦予利福平抗性的突變(上圖)。估計利福平抗性株的出現頻率為10%左右(下圖)。
[圖12]顯示基于指導RNA的長度而調節編輯的堿基部位的圖。圖12A顯示在使靶核苷酸序列長為20堿基的情況下、為24堿基的情況下的編輯的堿基部位的概念圖。圖12B顯示以gsiA基因為靶標,改變靶核苷酸序列長度進行編輯的結果。突變部位為粗體,“T”及“A”表示在克隆內完全導入了突變(C→T或G→A);“t”表示以50%以上的比例在克隆內導入了突變(C→T)(不完全克隆);“c”表示在克隆內,突變(C→T)的導入效率低于50%。
[圖13]實施例11使用的突變導入用溫度敏感性質粒的模式圖。
[圖14]顯示實施例11中的突變導入的方案的圖。
[圖15]顯示實施例11中向rpoB基因導入突變的結果的圖。
[圖16]顯示實施例11中向galK基因導入突變的結果的圖。
具體實施方式
本發明提供:不切割待修飾的雙鏈DNA的至少一條鏈,而是通過將該雙鏈DNA中靶核苷酸序列及其附近的核苷酸轉變為其它核苷酸,從而修飾該雙鏈DNA的該靶向位點的方法。該方法包括:通過使由核酸堿基轉變酶、和可與該雙鏈DNA中的靶核苷酸序列特異性結合的核酸序列識別模塊結合而成的復合體,與該雙鏈DNA接觸,而使該靶向位點,即靶核苷酸序列及其附近的核苷酸轉變為其它核苷酸的工序。
在本發明中,對雙鏈DNA的“修飾”是指使DNA鏈上的某核苷酸(例如:dC)缺失或轉變為其它核苷酸(例如:dT、dA或dG),或者向DNA鏈上的核苷酸之間插入核苷酸或者核苷酸序列。這里,對待修飾的雙鏈DNA沒有特別限制,優選為基因組DNA。另外,雙鏈DNA的“靶向位點”是指,核酸序列識別模塊可特異性識別并結合的“靶核苷酸序列”的全部或者一部分,或指其和該靶核苷酸序列的附近(5’上游及3’下游中任意一種或兩種),根據目的不同,該范圍可以在1堿基~幾百個堿基的長度之間適當調節。
在本發明中,“核酸序列識別模塊”是指,具有對DNA鏈上的特定的核苷酸序列(即,靶核苷酸序列)進行特異性識別并結合的能力的分子或分子復合體。核酸序列識別模塊可以通過與靶核苷酸序列結合,使與該模塊連接的核酸堿基轉變酶在雙鏈DNA的靶向位點發揮特異性的作用。
在本發明中,“核酸堿基轉變酶”是指,可以通過催化DNA堿基的嘌呤或嘧啶環上的取代基轉變為其它基團或原子的反應,不切割DNA鏈,將靶核苷酸轉變為其它核苷酸的酶。
在本發明中,“核酸修飾酶復合體”是指,包含由上述核酸序列識別模塊和核酸堿基轉變酶連接而成的復合體,賦予了特定的核苷酸序列識別能力的具有核酸堿基轉變酶活性的分子復合體。在此,“復合體”不僅包括由多個分子構成的形式,也包括像融合蛋白那樣的在單個分子內具有核酸序列識別模塊和核酸堿基轉變酶的形式。
用于本發明的核酸堿基轉變酶只要可以催化上述反應即可,沒有特別限制,可列舉例如,催化氨基轉變為羰基的脫氨基反應的、屬于核酸/核苷酸脫氨酶超家族的脫氨酶。優選的核酸堿基轉變酶可列舉:可以將胞嘧啶或5-甲基胞嘧啶分別轉變為尿嘧啶或胸腺嘧啶的胞苷脫氨酶、可以將腺嘌呤轉變為次黃嘌呤的腺苷脫氨酶、可以將鳥嘌呤轉變為黃嘌呤的鳥苷脫氨酶等。作為胞苷脫氨酶,可列舉更優選作為在脊椎動物的獲得性免疫中在免疫球蛋白基因中導入突變的酶的活化誘導的胞苷脫氨酶(下文,也稱為AID)等。
對核酸堿基轉變酶的來源沒有特別限制,可以使用例如:源自七腮鰻的PmCDA1(Petromyzon marinus cytosine deaminase 1)、源自哺乳動物(例如:人、豬、牛、馬、猴等)的AID(Activation-induced cytidine deaminase;AICDA)。分別將PmCDA1的CDS的堿基序列及氨基酸序列顯示于序列編號1和2中,將人AID的CDS的堿基序列及氨基酸序列顯示于序列編號3和4中。
對利用本發明的核酸修飾酶復合體的核酸序列識別模塊進行識別的雙鏈DNA中的靶核苷酸序列而言,只要可以與該模塊特異性結合即可,沒有特別的限制,可以為雙鏈DNA中的任意序列。就靶核苷酸序列的長度而言,只要足以與核酸序列識別模塊進行特異性結合即可,例如:在向哺乳動物的基因組DNA中特定的部位導入突變時,根據其基因組尺寸為12個核苷酸以上,優選為15個核苷酸以上,更優選為17個核苷酸以上。對長度的上限沒有特別的限制,優選為25個核苷酸以下,更優選為22個核苷酸以下。
作為本發明的核酸修飾酶復合體的核酸序列識別模塊,可以使用例如:Cas的至少1種DNA切割能力已失活的CRISPR-Cas系統(CRISPR-突變Cas)、鋅指基序、TAL效應子及PPR基序等,除此以外,包含限制酶、轉錄因子、RNA聚合酶等可與DNA進行特異性結合的蛋白質的DNA結合結構域且不具有DNA雙鏈切割能力的片段等,但不限于這些。可列舉優選:CRISPR-突變Cas、鋅指基序、TAL效應子、PPR基序等。
鋅指基序由3~6個Cys2His2不同型的鋅指單元(1個手指識別約3個堿基)連接而成,可以識別9~18個堿基的靶核苷酸序列。鋅指基序可以根據Modular assembly法(Nat Biotechnol(2002)20:135-141)、OPEN法(MolCell(2008)31:294-301)、CoDA法(Nat Methods(2011)8:67-69)、大腸桿菌單雜交法(Nat Biotechnol(2008)26:695-701)等公知的方法制作。對于制作鋅指基序的的細節,可以參照上述專利文獻1。
對TAL效應子而言,具有以約34氨基酸作為單位的模塊的重復結構,利用1個模塊的第12及13個氨基酸殘基(稱為RVD)確定結合穩定性和堿基特異性。由于各個模塊的獨立性較高,因此可以僅將模塊相連來制作對靶核苷酸序列特異性的的TAL效應子。TAL效應子可以利用Open resource的制作方法(REAL法(Curr Protoc Mol Biol(2012)Chapter 12:Unit 12.15)、FLASH法(Nat Biotechnol(2012)30:460-465)、Golden Gate法(Nucleic Acids Res(2011)39:e82)等)進行構建,比較簡便地設計相對于靶核苷酸序列的TAL效應子。對于制作TAL效應子的細節,可以參照上述專利文獻2。
對PPR基序而言,構建為使得通過包含35個氨基酸的1個核酸堿基識別PPR基序的連續來識別特定的核苷酸序列,僅利用各基序的第1、4及第ii(倒數第2)的氨基酸識別靶向堿基。由于對基序構成沒有依賴性,不受兩側的基序的干涉,因此與TAL效應子相同地,可以僅將PPR基序相連來制作對靶核苷酸序列特異性的PPR蛋白質。對于制作PPR基序的細節,可以參照上述專利文獻4。
另外,在使用限制酶、轉錄因子、RNA聚合酶等片段的情況下,由于它們的蛋白質的DNA結合結構域是眾所周知的,因此可以容易地設計、構建包含該結構域且不具有DNA雙鏈切割能力的片段。
上述任意核酸序列識別模塊也可以以上述核酸堿基轉變酶的融合蛋白的形式提供,或者,也可以將SH3結構域、PDZ結構域、GK結構域、GB結構域等蛋白質結合結構域和它們的結合配偶體,分別與核酸序列識別模塊和與核酸堿基轉變酶融合,通過該結構域和它們的結合配體的相互作用而以蛋白質復合體的形式提供。或者,也可以將核酸序列識別模塊與核酸堿基轉變酶和與內含肽(intein)分別融合,利用各蛋白質合成后的接合將兩者連接。
就包含核酸序列識別模塊與核酸堿基轉變酶結合而成的復合體(包括融合蛋白)的本發明的核酸修飾酶復合體與雙鏈DNA的接觸而言,雖然可以以無細胞體系的酶反應的形式進行,但鑒于本發明的主要的目的,優選通過向具有目的的雙鏈DNA(例如,基因組DNA)的細胞導入編碼該復合體的核酸來實施。
因此,對核酸序列識別模塊與核酸堿基轉變酶而言,優選以編碼它們的融合蛋白的核酸的形式制備,或者,以使得利用結合結構域、內含肽等翻譯成蛋白質之后,可以在宿主細胞內形成復合體的形態,以分別編碼它們的核酸的形式進行制備。在此,核酸可以是DNA也可以是RNA。在為DNA時,優選為雙鏈DNA,并且以在宿主細胞內配置為在功能性的啟動子的控制下的表達載體的形態提供。在為RNA時,優選為單鏈RNA。
由于核酸序列識別模塊和核酸堿基轉變酶結合而成的本發明的復合體不伴隨DNA雙鏈切割(DSB),因此,能夠進行毒性較低的基因組編輯,本發明的基因修飾方法可以適用于范圍較廣的生物材料。因此,就待導入核酸序列識別模塊及/或核酸堿基轉變酶編碼核酸的細胞而言,從作為原核生物的大腸桿菌等細菌、作為低等真核生物的酵母等微生物的細胞,到包括人等哺乳動物的脊椎動物、昆蟲、植物等高等真核生物的細胞,可以包括所有生物種類的細胞。
對編碼鋅指基序、TAL效應子、PPR基序等核酸序列識別模塊的DNA而言,可以通過上述任意方法獲得各模塊。對編碼限制酶、轉錄因子、RNA聚合酶等序列識別模塊的DNA而言,可以通過以下進行克隆:例如基于它們的cDNA序列信息,來合成使得覆蓋編碼該蛋白質的期望部分(包含DNA結合結構域的部分)的區域的寡DNA引物,并利用從產生該蛋白質的細胞制備的總RNA或者mRNA級分作為模板,通過RT-PCR法擴增。
編碼核酸堿基轉變酶的DNA也可以同樣地通過以下進行克隆:基于待使用的酶的cDNA序列信息來合成寡DNA引物,使用由產生該酶的細胞制備的總RNA或者mRNA級分作為模板,通過RT-PCR法擴增。例如,就編碼七腮鰻的PmCDA1的DNA而言,可以基于NCBI數據庫登記的cDNA序列(accession No.EF094822),針對CDS的上游及下游設計適當的引物,通過RT-PCR法從源自七腮鰻的mRNA進行克隆。另外,就編碼人AID的DNA而言,可以基于NCBI數據庫登記的cDNA序列(accession No.AB040431),針對CDS的上游及下游設計適當的引物,例如通過RT-PCR法從源自人淋巴結的mRNA進行克隆。
已克隆的DNA可以直接或根據需要利用限制酶進行消化,或在加入適當的接頭及/或核定位信號(在目的的雙鏈DNA為線粒體、或葉綠體DNA的情況下,為各細胞器定位信號)之后,與編碼核酸序列識別模塊的DNA進行接合,來制備編碼融合蛋白的DNA。或者,可以通過將編碼核酸序列識別模塊的DNA和編碼核酸堿基轉變酶的DNA分別與編碼結合結構域或者其結合配偶體的DNA進行融合,或也可以將兩種DNA通過與編碼分離內含肽的DNA進行融合,使得核酸序列識別轉變模塊和核酸堿基轉變酶在宿主細胞內翻譯,然后再形成復合體。在這些情況下,也可以根據需要,在一方或雙方DNA的適當的位置連接接頭及/或核定位信號。
對編碼核酸序列識別模塊的DNA和編碼核酸堿基轉變酶的DNA而言,可以化學合成DNA鏈,或者也可以通過將合成的部分重疊的寡DNA短鏈利用PCR法、Gibson Assembly法進行連接,構建編碼其全長的DNA。利用組合化學合成或PCR法或者Gibson Assembly法來構建全長DNA的優點在于,可以在整個CDS全長上設計與待導入該DNA的宿主配合的使用密碼子。通過在表達異種DNA時,將該DNA序列轉變為在宿主生物中使用頻率高的密碼子,可以期待使蛋白質表達量增大。就使用的宿主中的密碼子使用頻率的數據而言,可以使用例如在(公財)Kazusa DNA研究所的主頁上公開的遺傳密碼使用頻率數據庫(http://www.kazusa.or.jp/codon/index.html),還可以參照記載在各宿主中密碼子的使用頻率的文獻。只要參照取得的數據和準備導入的DNA序列,將該DNA序列中使用的密碼子中在宿主中使用頻率低的密碼子,轉變為編碼同一氨基酸且使用頻率高的密碼子即可。
包含編碼核酸序列識別模塊及/或核酸堿基轉變酶的DNA表達載體,可以通過例如將該DNA連接至適當的表達載體的啟動子的下游來制造。
作為表達載體,可以使用源自大腸桿菌的質粒(例如,pBR322、pBR325、pUC12、pUC13);源自枯草桿菌的質粒(例如,pUB110、pTP5、pC194);源自酵母的質粒(例如,pSH19、pSH15);昆蟲細胞表達質粒(例如:pFast-Bac);動物細胞表達質粒(例如:pA1-11、pXT1、pRc/CMV、pRc/RSV、pcDNAI/Neo);λ噬菌體等噬菌體;桿狀病毒等昆蟲病毒載體(例如:BmNPV、AcNPV);逆轉錄病毒、痘苗病毒、腺病毒等動物病毒載體等。
作為啟動子,只要是對應于基因表達的宿主的適當的啟動子即可。由于伴隨DSB的常規法的毒性的緣故,有時宿主細胞的存活率顯著降低,因此期望通過使用誘導啟動子增加誘導開始為止的細胞數量,但由于表達本發明的核酸修飾酶復合體也可得到充分的細胞增殖,因此可以無限制地使用構成啟動子。
例如,在宿主為動物細胞的情況下,可以使用SRα啟動子、SV40啟動子、LTR啟動子、CMV(巨細胞病毒)啟動子、RSV(勞斯肉瘤病毒)啟動子、MoMuLV(Moloney小鼠白血病病毒)LTR、HSV-TK(單純皰疹病毒胸苷激酶)啟動子等。其中,優選CMV啟動子、SRα啟動子等。
當宿主是大腸桿菌的情況下,優選trp啟動子、lac啟動子、recA啟動子、λPL啟動子、lpp啟動子、T7啟動子等。
當宿主是芽孢桿菌屬的情況下,優選SPO1啟動子、SPO2啟動子、penP啟動子等。
當宿主是酵母的情況下,優選Gal1/10啟動子、PHO5啟動子、PGK啟動子、GAP啟動子、ADH啟動子等。
當宿主是昆蟲細胞的情況下,優選多角體蛋白啟動子、P10啟動子等。
當宿主是植物細胞的情況下,優選CaMV35S啟動子、CaMV19S啟動子、NOS啟動子等。
作為表達載體,除了上述以外,可以根據需要使用含有增強子、剪接信號、終止子、polyA添加信號、選擇標記如藥物抗性基因、營養缺陷型互補基因等、復制起點等的載體。
編碼核酸序列識別模塊及/或核酸堿基轉變酶的RNA,可以通過例如以編碼上述核酸序列識別模塊及/或核酸堿基轉變酶的DNA編碼載體作為模板,通過本身已知的體外轉錄系統轉錄為mRNA來制備。
可以通過將包含編碼核酸序列識別模塊及/或核酸堿基轉變酶的DNA表達載體導入宿主細胞并培養該宿主細胞,在細胞內表達核酸序列識別模塊和核酸堿基轉變酶的復合體。
作為宿主,可以使用例如:埃希氏菌屬、芽孢桿菌屬、酵母、昆蟲細胞、昆蟲、動物細胞等。
作為埃希氏桿菌屬,可以使用例如:大腸桿菌(Escherichia coli)K12?DH1[Proc.Natl.Acad.Sci.USA,60,160(1968)]、大腸桿菌JM103[Nucleic Acids Research,9,309(1981)]、大腸桿菌JA221[Journal of Molecular Biology,120,517(1978)]、大腸桿菌HB101[Journal of Molecular Biology,41,459(1969)]、大腸桿菌C600[Genetics,39,440(1954)]等。
作為芽孢桿菌屬,可以使用例如:枯草芽孢桿菌(Bacillus subtilis)MI114[Gene,24,255(1983)]、枯草芽孢桿菌207-21[Journal of Biochemistry,95,87(1984)]等。
作為酵母,可以使用例如:釀酒酵母(Saccharomyces cerevisiae)AH22、AH22R-、NA87-11A、DKD-5D、20B-12、粟酒裂殖酵母(Schizosaccharomyces pombe)NCYC1913、NCYC2036、巴斯德畢赤酵母(Pichia pastoris)KM71等。
作為昆蟲細胞,可以使用例如:在病毒為AcNPV的情況下,源自甘藍夜蛾的幼蟲來源的株系細胞(Spodoptera frugiperda cell;Sf細胞)、Trichoplusia ni的中腸MG1細胞、源自Trichoplusia ni的卵的High FiveTM細胞、源自Mamestra brassicae的細胞、源自Estigmena acrea的細胞等。在病毒為BmNPV的情況下,作為昆蟲細胞,可以使用源自蠶的株系細胞(Bombyx mori N細胞;BmN細胞)等。作為該Sf細胞,可以使用例如:Sf9細胞(ATCC CRL1711)、Sf21細胞[以上,In Vivo,13,213-217(1977)]等。
作為昆蟲,可以使用例如:蠶的幼蟲、果蠅、蟋蟀等[Nature,315,592(1985)]。
作為動物細胞,可以使用例如:猴COS-7細胞、猴Vero細胞、中國倉鼠卵巣(CHO)細胞、dhfr基因缺陷CHO細胞、小鼠L細胞、小鼠AtT-20細胞、小鼠骨髓瘤細胞、大鼠GH3細胞、人FL細胞等細胞株、人及其它哺乳動物的iPS細胞、ES細胞等多功能性干細胞、由各種組織制備的初代培養細胞。進一步,也可以使用斑馬魚胚胎、非洲爪蟾卵母細胞等。
作為植物細胞,可以使用從各種植物(例如:水稻、小麥、玉米等谷物,番茄、黃瓜、茄子等商品作物,康乃馨、洋桔梗等園藝植物,煙草、擬南芥等實驗植物等)制備的懸浮培養細胞、愈傷組織、原生質體、葉切片、根切片等。
上述任意宿主細胞可以為單倍體(一倍體)、多倍體(例如,二倍體、三倍體、四倍體等)。常規的突變導入方法中,作為原則僅向同源染色體的一條中導入突變而形成雜合基因型,因此,如果不是優勢突變,就不會表達需要的特質,而純合化耗時耗力,大多情況不方便。與此相對,根據本發明,由于可以對基因組內的同源染色體上的等位基因全部導入突變,因此即使為劣勢突變,也可以在該代表達需要的特質(圖10),從克服了常規法的癥結的方面考慮,極其有用。
對表達載體的導入而言,可以根據宿主的種類,按照公知的方法(例如:溶菌酶法、感受態法、PEG法、CaCl2共沉淀法、電穿孔法、顯微注射法、粒子槍法、脂質轉化法、農桿菌法等)進行實施。
對于大腸桿菌,可以按照例如:Proc.Natl.Acad.Sci.USA,69,2110(1972)、Gene,17,107(1982)等中記載的方法進行轉化。
對于芽孢桿菌屬,可以按照例如:Molecular&General Genetics,168,111(1979)等中記載的方法進行載體導入。
對于酵母,可以按照例如:Methods in Enzymology,194,182-187(1991),Proc.Natl.Acad.Sci.USA,75,1929(1978)等中記載的方法進行載體導入。
對于昆蟲細胞及昆蟲,可以按照例如:Bio/Technology,6,47-55(1988)等中記載的方法進行載體導入。
對于動物細胞,可以按照例如:細胞工程學增刊8新細胞工程學實驗方案,263-267(1995)(秀潤公司發行),Virology,52,456(1973)中記載的方法進行載體導入。
就已導入載體的細胞的培養而言,可以根據宿主的種類,按照公知的方法實施。
例如,在培養大腸桿菌或芽孢桿菌的情況下,作為用于培養的培養基優選液體培養基。另外,培養基優選含有轉化體的生長所必需的碳源、氮源、無機物等。這里,作為碳源,可以舉出例如:葡萄糖、糊精、可溶性淀粉、蔗糖等;作為氮源,可以舉出例如:銨鹽類、硝酸鹽類、玉米漿、蛋白胨、酪蛋白、肉浸膏、大豆餅、馬鈴薯提取液等無機或有機物質;作為無機物,可以舉出例如:氯化鈣、磷酸二氫鈉、氯化鎂等。另外,也可以向培養基添加酵母提取物、維生素類、生長促進因子等。培養基的pH優選為約5~約8。
作為培養大腸桿菌時的培養基,例如,優選包含葡萄糖、酪蛋白氨基酸的M9培養基[Journal of Experiments in Molecular Genetics,431-433,Cold Spring Harbor Laboratory,New York 1972]。根據需要,為了使啟動子有效地工作,例如也可以向培養基添加3β-吲哚基丙烯酸的那樣的試劑。大腸桿菌的培養通常在約15~約43℃進行。根據需要,可以進行通氣、攪拌。
芽孢桿菌的培養通常在約30~約40℃進行。根據需要,可以進行通氣、攪拌。
作為培養酵母時的培養基,可列舉例如:Burkholder最小培養基[Proc.Natl.Acad.Sci.USA,77,4505(1980)]、0.5%含酪蛋白氨基酸的SD培養基[Proc.Natl.Acad.Sci.USA,81,5330(1984)]等。培養基的pH優選為約5~約8。培養通常在約20℃~約35℃進行。可以根據需要進行通氣、攪拌。
作為培養昆蟲細胞或昆蟲時的培養基,可以使用例如向Grace's Insect Medium[Nature,195,788(1962)]中適當添加了滅活的10%牛血清等添加物的培養基等。培養基的pH優選為約6.2~約6.4。培養通常在約27℃進行。可以根據需要進行通氣、攪拌。
作為培養動物細胞時的培養基,可以使用例如:包含約5~約20%的胎牛血清的最小必要培養基(MEM)[Science,122,501(1952)]、Dulbecco改良的Eagle培養基(DMEM)[Virology,8,396(1959)]、RPMI 1640培養基[The Journal of the American Medical Association,199,519(1967)]、199培養基[Proceeding of the Society for the Biological Medicine,73,1(1950)]等。培養基的pH優選為約6~約8。培養通常在約30℃~約40℃進行。可以根據需要進行通氣、攪拌。
作為培養植物細胞的培養基,可以使用MS培養基、LS培養基、B5培養基等。培養基的pH優選為約5~約8。培養通常在約20℃~約30℃進行。可以根據需要進行通氣、攪拌。
如上所述操作,可以在細胞內表達核酸序列識別模塊和核酸堿基轉變酶的復合體,即核酸修飾酶復合體。
編碼核酸序列識別模塊及/或核酸堿基轉變酶RNA向宿主細胞的導入,可以通過顯微注射法、脂質轉化法等進行。RNA的導入可以1次或者隔著適當的間隔反復進行多次(例如:2~5次)。
由導入至細胞內的表達載體或RNA分子,表達核酸序列識別模塊和核酸堿基轉變酶的復合體時,該核酸序列識別模塊特異性識別并結合目的的雙鏈DNA(例如,基因組DNA)內的靶核苷酸序列,利用連接于該核酸序列識別模塊的核酸堿基轉變酶的作用,靶向位點(可以在包含靶核苷酸序列的全部或者一部分或它們的附近的幾百個堿基的范圍內適當調節)的有義鏈或者反義鏈發生堿基轉變,在雙鏈DNA內產生錯配(例如:將PmCDA1、AID等胞苷脫氨酶作為核酸堿基轉變酶使用的情況下,靶向位點的有義鏈或者反義鏈上的胞嘧啶被轉變為尿嘧啶,產生U:G或者G:U錯配)。通過以下導入各種突變:該錯配未被正確修復,或修復使得相反鏈的堿基與已轉變的鏈的堿基成對(上述的例子中,為T-A或者A-T),修復時進一步取代為其它核苷酸(例如:U→A、G),或者發生1個~數十個堿基的缺失或者插入。
對鋅指基序而言,由于與靶核苷酸序列特異性結合的鋅指的制作效率不高,另外,結合特異性高的鋅指的篩選較為復雜,因此,制作實際發揮功能的多個鋅指基序并不容易。就TAL效應子、PPR基序而言,比鋅指基序的靶核酸序列識別的自由度高,但需要每次根據靶核苷酸序列設計并構建巨大的蛋白質,因此在效率方面存在問題。
與此相對,由于CRISPR-Cas系統是通過相對于靶核苷酸序列互補的指導RNA來識別目的的雙鏈DNA的序列,因此可以通過僅合成可與靶核苷酸序列形成特異性的Hybrid的寡DNA,以任意序列為靶標。
因此,在本發明更優選的實施形態中,使用了Cas的至少1種DNA切割能力已失活的CRISPR-Cas系統(CRISPR-突變Cas)作為核酸序列識別模塊。
圖1為顯示將CRISPR-突變Cas用作核酸序列識別模塊的,本發明的雙鏈DNA修飾方法的模式圖。
使用了CRISPR-突變Cas的本發明的核酸序列識別模塊,是以RNA分子與突變Cas蛋白質的復合體的形式提供的,其中,所述RNA分子由與靶核苷酸序列互補的指導RNA、對突變Cas蛋白質的募集必需的tracrRNA組成。
本發明使用的Cas蛋白質只要屬于CRISPR系統即可,沒有特別限制,優選為Cas9。作為Cas9,可列舉例如,源自化膿性鏈球菌(Streptococcus pyogenes)的Cas9(SpCas9)、源自嗜熱鏈球菌(Streptococcus thermophilus)的Cas9(StCas9)等,但不限于這些。優選為SpCas9。作為本發明使用的突變Cas,Cas蛋白質的雙鏈DNA的兩條鏈的切割能力已失活的、和僅失活一條鏈的切割能力的具有切口酶活性的均可以使用。例如,在為SpCas9的情況下,可以使用第10位的Asp殘基轉變為Ala殘基的、欠缺對形成與指導RNA互補的鏈的相反鏈的切割能力的D10A突變體,或者,第840位的His殘基轉變為Ala殘基的、欠缺與指導RNA互補的鏈的切割能力的H840A突變體,進一步可使用其雙突變體,但也可以使用其它突變Cas。
對核酸堿基轉變酶而言,通過與上述鋅指等的連接方式相同的方法,以突變Cas的復合體的形式提供。或者,也可以將核酸堿基轉變酶和突變Cas,利用與作為RNA適體的MS2F6、PP7等和由它們的結合蛋白質構成的RNA支架進行結合。指導RNA與靶核苷酸序列形成互補鏈,接著tracrRNA募集突變Cas,對DNA切割部位識別序列PAM(protospacer adjacent motif)(當使用SpCas9的情況下,PAM為NGG(N為任意堿基)3個堿基,理論上,基因組上的任意部位都可以被靶向化)進行識別,不切割一條或者兩條DNA,利用連接于突變Cas的核酸堿基轉變酶的作用,使靶向部位(可以在包含靶核苷酸序列的全部或者一部分的幾百個堿基的范圍內適當調節)產生堿基轉變,在雙鏈DNA內產生錯配。通過以下導入各種突變:該錯配未被正確修復,修復使得相反鏈的堿基與已轉變鏈的堿基成對,修復時進一步被轉變為其它核苷酸,或者發生1個~數十個堿基的缺失或者插入,(例如:參照圖2)。
在將CRISPR-突變Cas用作核酸序列識別模塊的情況下,也與將鋅指等用作核酸序列識別模塊的情況相同地,優選將核酸序列識別模塊和核酸堿基轉變酶,以編碼它們的核酸的形態導入具有目的的雙鏈DNA的細胞。
編碼Cas的DNA,可以通過與對于編碼核酸堿基轉變酶的DNA的上述方法同樣的方法從產生該酶的細胞進行克隆。另外,突變Cas可以通過以下獲得:在已克隆的Cas編碼DNA中,使用本身公知的部位特異性的突變誘發法,以將對DNA切割活性重要的部位的氨基酸殘基(例如:在為Cas9的情況下,可列舉第10位的Asp殘基、第840位的His殘基,但不限于這些)轉變為其它氨基酸的方式導入突變。
或者,對編碼突變Cas的DNA而言,也可以針對編碼核酸序列識別模塊的DNA、編碼核酸堿基轉變酶的DNA,利用與上述相同的方法和化學合成或PCR法或Gibson Assembly法的組合,構建成具有適于使用的宿主細胞的表達的密碼子使用的DNA形式。例如:將為了在真核細胞中表達SpCas9的最優化的CDS序列及氨基酸序列示于序列編號5及6。如果將序列編號5所示的序列中的堿基編號29的“A”轉變為“C”,則可以得到編碼D10A突變體的DNA,如果將堿基編號2518-2519的“CA”轉變為“GC”,則可以得到編碼H840A突變體的DNA。
對編碼突變CasDNA和編碼核酸堿基轉變酶的DNA而言,可以連接成使得作為融合蛋白表達,也可以設計成使得使用結合結構域、內含肽等分別進行表達,通過蛋白質間相互作用、蛋白質接合在宿主細胞內形成復合體。
對得到的編碼突變Cas及/或核酸堿基轉變酶的DNA而言,可以根據宿主不同,插入至與上述相同的表達載體的啟動子的下游。
另一方面,編碼指導RNA及tracrRNA的DNA可以設計成,將相對于靶核苷酸序列互補的指導RNA序列與已知的tracrRNA序列(例如:gttttagagctagaaatagcaagttaaaataaggctagtccgttatcaacttgaaaaagtggcaccgagtcggtggtgctttt;序列編號7)連接而成的寡DNA序列,并用DNA/RNA合成儀進行化學合成。
對指導RNA序列的長度而言,只要是可相對于靶核苷酸序列特異性結合即可,沒有特別限制,例如為15~30個核苷酸,優選為18~24個核苷酸。
編碼指導RNA及tracrRNA的DNA也可以根據宿主不同,插入至與上述相同的表達載體,作為啟動子,優選使用pol III類的啟動子(例如,SNR6、SNR52、SCR1、RPR1、U6、H1啟動子等)及終止子(例如,T6序列)。
對編碼突變Cas及/或核酸堿基轉變酶的RNA而言,可以通過例如,以對編碼上述突變Cas及/或核酸堿基轉變酶的DNA進行編碼的載體作為模板,通過本身公知的體外轉錄系統轉錄為mRNA來制備。
可以將指導RNA-tracrRNA設計成由與靶核苷酸序列互補的序列與已知的tracrRNA序列連接而成的寡RNA序列,并用DNA/RNA合成儀進行化學合成。
對編碼突變Cas及/或核酸堿基轉變酶的DNA或者RNA、指導RNA-tracrRNA或編碼其的DNA而言,可以根據宿主不同,通過與上述相同的方法導入宿主細胞。
常規型的人工核酸酶中,由于伴隨DNA雙鏈切割(DSB),因此,對基因組內的序列進行靶向時,發生染色體的無序的切割(脫靶切割)導致的增殖障礙和細胞死亡。該影響對很多微生物、原核生物是特別致命的,阻礙了其適用性。在本發明中,由于通過不切割DNA的對DNA堿基上的取代基的轉變反應(特別是脫氨基反應)進行突變導入,因此,可以實現毒性的大幅降低。實際上,如下文的實施例所示,在將芽殖酵母作為宿主使用的比較試驗中,確認在使用了具有常規型的DSB活性的Cas9的情況下,由表達誘導導致細胞存活數減少,與此相對,在組合了突變Cas和核酸堿基轉變酶的本發明的技術中,細胞繼續增殖,細胞存活數增加(圖3)。
需要說明的是,本發明的雙鏈DNA的修飾不會妨礙在靶向位點(可以在包含靶核苷酸序列的全部或者一部分的幾百個堿基的范圍內適當調節)以外產生該雙鏈DNA的切割。然而,本發明的最大的優點之一,是避免了由脫靶(off-target)切割導致的毒性,如果考慮到原則上可以適用于任何生物種類,則在優選的一種實施形態中,本發明的雙鏈DNA的修飾不僅在選擇的雙鏈DNA的靶向位點不伴隨DNA鏈的切割,在除其以外的部位也是。
另外,如下文的實施例所示,與使用兩條鏈均不能切斷的突變Cas的情況相比,作為突變Cas使用了具有僅能切割雙鏈DNA之中的一條鏈的切口酶(nickase)活性的Cas的情況下(圖5),突變導入效率升高。因此,例如,可以通過在核酸序列識別模塊和核酸堿基轉變酶以外,進一步連接具有切口酶活性的蛋白質,并在靶核苷酸序列的附近僅切割DNA一根鏈,從而在避免基于DSB的強毒性的同時,使突變導入效率提高。
進一步,對具有切割不同鏈的兩種切口酶活性的突變Cas的效果進行比較,結果顯示一種的突變部位集中于靶核苷酸序列的中央附近,相對于此,另一種向從靶核苷酸序列到達幾百個堿基的區域隨機導入了多種突變(圖6)。因此,通過選擇切口酶待切割的鏈,可以向特定的核苷酸或核苷酸區域導入點性突變,或者向比較寬的范圍隨機導入多種突變,可以根據目的不同而分開使用。例如,如果將前者的技術用于基因疾病iPS細胞,則可以通過在修復了由患者自身的細胞制作的iPS細胞中的病原基因的突變之后,通過使其分化為目的體細胞,制作排斥風險更低的細胞移植療法劑。
在下文的實施例7中,示出了可以對特定的核苷酸中幾乎在針點上導入突變。像這樣,為了對期望的核苷酸針點性導入突變,只要設定靶核苷酸序列使得希望導入突變的核苷酸和靶核苷酸序列的位置關系存在規律性即可。例如,在使用CRIPR-Cas系統核酸序列作為識別模塊、且使用核酸堿基轉變酶作為AID的情況下,可以設計靶核苷酸序列,使得希望導入突變的C(或者其相反鏈的G)在離靶核苷酸序列的5’端2~5個核苷酸的位置。如上所述,指導RNA序列的長度可以在15~30個核苷酸,優選為18~24個核苷酸之間適當設定。由于指導RNA序列為與靶核苷酸序列互補的序列,因此改變指導RNA序列的長度,導致靶核苷酸序列的長度也發生變化,但無論核苷酸長度如何,都保持對存在于離5’端2~5個核苷酸的位置的C或G易于導入突變的規律性(圖12)。因此,通過適當選擇靶核苷酸序列(作為其互補鏈的指導RNA)的長度,可以移動能夠導入突變堿基的部位。由此,也可以解除由DNA切割部位識別序列PAM(NGG)導致的限制,進一步提高突變導入的自由度。
如下文的實施例所示,與以單獨的核苷酸序列為靶相比,通過相對于鄰近的多個靶核苷酸序列而制作序列識別模塊并同時使用,突變導入效率大幅升高(圖7)。效果是,從使得兩個靶核苷酸序列的一部分發生重復的情到即使兩者分開600bp左右的情況下,都同樣實現突變誘導。另外,在靶核苷酸序列存在于相同方向(靶核苷酸序列存在于相同鏈上)(圖7)、以及在對向(靶核苷酸序列存在于雙鏈DNA的各自鏈上)(圖4)的兩種情況下,均可發生。
如下文的實施例所示,就本發明的基因組序列的修飾方法而言,如果選擇適當的靶核苷酸序列,則可以在表達本發明的核酸修飾酶復合體的幾乎全部的細胞中導入突變(圖8)。因此,不需要在常規基因組編輯中所必需的選擇標識基因的插入和選擇。其在使基因操作顯著地變得簡便的同時,由于沒有了重組外源DNA的生物,因此對作物育種等的適用性大幅變寬。
另外,由于突變導入效率極高,且不需要利用標識的選擇,因此在本發明的基因組序列的修飾方法中,可以對完全不同的位置的多個DNA區域作為靶進行修飾(圖9)。因此,在本發明優選的一種實施形態中,可以使用與不同的靶核苷酸序列(可以在1個目的基因內,也可以在不同的2個以上目的基因內。這些目的基因可以位置在相同的染色體上,也可以在不同的染色體上。)分別特異性結合的、兩種以上的核酸序列識別模塊。在這種情況下,將這些核酸序列識別模塊的各1個與核酸堿基編輯酶形成核酸修飾酶復合體。這里,核酸堿基編輯酶可以使用共通的酶。例如,在使用CRISPR-Cas系統作為核酸序列識別模塊的情況下,可以對于Cas蛋白質和核酸堿基轉變酶的復合體(包含融合蛋白)使用共通的物質,制作并使用兩種以上的嵌合RNA作為指導RNA-tracrRNA,所述嵌合RNA使是由與不同的靶核苷酸序列分別形成互補鏈的2種以上的指導RNA的每一種分別與tracrRNA形成的。另一方面,在使用鋅指基序、TAL效應子等作為核酸序列識別模塊的情況下,例如可以將與不同靶核苷酸特異性結合的各核酸序列識別模塊,與核酸堿基轉變酶進行融合。
為了使本發明的核酸修飾酶復合體在宿主細胞內表達,如上所述將包含編碼該核酸修飾酶復合體的DNA的表達載體、編碼該核酸修飾酶復合體的RNA導入宿主細胞,但為了有效地導入突變,優選能保持規定時期以上、規定水平以上的核酸修飾酶復合體的表達。從該觀點出發,確定在宿主細胞內導入能夠自主復制的表達載體(質粒等),但由于該質粒等為外源DNA,因此,優選在順利實現了導入突變后迅速地將其除去。因此,雖然根據宿主細胞的種類等不同而改變,但優選例如:從導入表達載體到經過6小時~2天后,使用在本技術領域周知的各種質粒除去法,從宿主細胞除去已導入的質粒。
或者,只要可得到足以進行突變導入的核酸修飾酶復合體的表達,也優選使用在宿主細胞內不具有自主復制能力的表達載體(例如:缺乏在宿主細胞中發揮功能的復制起點及/或編碼對復制必須的蛋白質的基因的載體等)、RNA,通過一過性地的表達向目的的雙鏈DNA導入突變。
由于在使本發明的核酸修飾酶復合體在宿主細胞內進行表達,并進行核酸堿基轉變反應的時期,靶基因的表達被抑制,因此,迄今為止難以將對宿主細胞的生存所必需的基因作為靶基因直接編輯(產生宿主的生長障礙、突變導入效率不穩定、對與靶標不同的部位的突變等副作用)。本發明通過在期望的時期中,僅在需要發生核酸堿基轉變反應、固定靶向位點的修飾的時期,使本發明的核酸修飾酶復合體一過性地在宿主細胞內表達,從而成功有效地實現必需基因的直接編輯。需要發生核酸堿基轉變反應、固定靶向位點的修飾的時期根據宿主細胞的種類、培養條件等不同,但認為通常需要2~20代。例如,在宿主細胞為酵母、細菌(例如,大腸桿菌)的情況下,需要在5~10代之間誘導核酸修飾酶復合體的表達。本領域技術人員可以基于使用的培養條件下的宿主細胞的倍增時間,適當確定適宜的表達誘導期。例如,將芽殖酵母在0.02%半乳糖誘導培養基中進行液體培養時,可舉例為20~40小時的表達誘導期。就本發明的編碼核酸修飾酶復合體的核酸的表達誘導期而言,也可以在對宿主細胞不產生副作用的范圍內,超出上述“需要固定靶向位點的修飾的時期”并延長。
作為將本發明的核酸修飾酶復合體在期望的時期進行一過性地表達的方法,可列舉制作以能夠控制表達期的形態,包含編碼該核酸修飾酶復合體的核酸(在CRISPR-Cas系統中,為編碼指導RNA-tracrRNA的DNA、和編碼突變Cas及核酸堿基取代酶的DNA)的構建體(表達載體),并導入宿主細胞內的方法。“以能夠控制表達期的形態”,具體而言,可列舉將編碼本發明的核酸修飾酶復合體的核酸,置于誘導性的調節區域的控制下的形態。對“誘導性的調節區域”沒有特別限制,例如,在細菌(例如,大腸桿菌)、酵母等微生物細胞中,可列舉溫度敏感性(ts)突變阻遏物和控制其的操縱基因(operator)的控制子(operon)。作為ts突變阻遏物,可列舉例如源自λ噬菌體的cI阻遏物的ts突變體,但不限于這些。在為λ噬菌體cI阻遏物(ts)的情況下,在30℃以下(例如,28℃)與操縱基因結合而抑制下游的基因表達,但在37℃以上(例如,42℃)的高溫從操縱基因解離,因此可誘導基因表達(圖13及14)。因此,通過將已導入編碼核酸修飾酶復合體的核酸的宿主細胞,通常在30℃以下培養,在適當的時期將溫度升高至37℃以上并培養一定時期,使其進行核酸堿基轉變反應,并在向靶基因導入突變后迅速降回30℃以下,從而可以使得抑制靶基因表達的時期最短,即使在對宿主細胞所必需的基因進行靶向的情況下,也可以壓抑副作用并有效地進行編輯(圖15)。
在利用溫度敏感性突變的情況下,可以通過例如,將對載體的自主復制所必須的蛋白質的溫度敏感性突變體,搭載于包含編碼本發明的核酸修飾酶復合體的DNA載體,使該核酸修飾酶復合體表達之后,該載體不能迅速地自主復制,并隨著細胞分裂自然脫落。作為這樣的溫度敏感性突變蛋白質,可列舉pSC101 ori的復制所必須的Rep101 ori的溫度敏感性突變體,但不限于這些。對Rep101 ori(ts)而言,雖然在30℃以下(例如,28℃)能夠作用于pSC101 ori并進行質粒的自主復制,但處于37℃以上(例如,42℃)時喪失功能,質粒不能自主復制。因此,通過與上述λ噬菌體的cI阻遏物(ts)組合使用,可以使本發明的核酸修飾酶復合體的一過性地表達和質粒除去同時進行。
另一方面,將動物細胞、昆蟲細胞、植物細胞等高等真核細胞作為宿主細胞的情況下,在誘導啟動子(例如,金屬硫蛋白啟動子(利用重金屬離子誘導)、熱休克蛋白質啟動子(利用熱休克誘導)、Tet-ON/Tet-OFF類啟動子(利用添加或除去四環素或其衍生物進行誘導)、類固醇響應啟動子(利用類固醇激素或其衍生物進行誘導)等)的控制下,將編碼本發明的核酸修飾酶復合體的DNA導入宿主細胞內,在適當的時期向培養基添加誘導物質(或從培養基除去)來誘導該核酸修飾酶復合體的表達,培養一定時期,進行核酸堿基轉變反應,可以實現向靶基因導入突變后的核酸修飾酶復合體的一過性地表達。
需要說明的是,在大腸桿菌等原核細胞中也可以利用誘導啟動子。作為這樣的誘導啟動子,可列舉例如:lac啟動子(利用IPTG誘導)、cspA啟動子(利用冷休克誘導)、araBAD啟動子(利用阿拉伯糖誘導)等,但不限于這些。
或者,也可以將上述的誘導啟動子用作在以動物細胞、昆蟲細胞、植物細胞等高等真核細胞作為宿主細胞時的載體除去方法。即,通過使其搭載宿主細胞發揮功能的復制起點、和編碼對其復制所必須的蛋白質的核酸(例如,如果為動物細胞,則為SV40 ori和大T抗原、oriP和EBNA-1等),利用上述誘導啟動子來控制編碼該蛋白質的核酸的表達,雖然在誘導物質的存在下載體能夠進行自主復制,但如果除去誘導物質則不能進行自主復制,隨著細胞分裂,載體將自然脫落(在Tet-OFF類載體中,相反地,通過添加四環素、多西環素導致不能進行自主復制)。
以下,根據實施例對本發明進行說明。但是,本發明并不限于這些實施例。
實施例
在下文的實施例1~6中,如下所述地進行了實驗。
<細胞株、培養、轉化(transformation)、表達誘導>
使用芽殖酵母Saccharomyces cerevisiae BY4741株(需要亮氨酸及尿嘧啶),利用標準的YPDA培養基或符合營養要求的撤出成分(Dropout)組成的SD培養基進行培養。培養在從25℃到30℃之間,通過利用瓊脂板的靜置培養或利用液體培養基的振蕩培養進行。對轉化而言,使用乙酸鋰法,利用符合適當的營養要求的SD培養基進行選擇。對基于半乳糖的表達誘導而言,利用適當的SD培養基預培養一夜,然后接種于將碳源從2%葡萄糖代替為2%棉子糖的SR培養基繼續培養一夜,進一步接種于將碳源代替為0.2~2%半乳糖的SGal培養基繼續培養3小時到二夜左右,進行表達誘導。
對于細胞存活數及Can1突變率的測定,將細胞懸濁液適當稀釋于SD平板培養基及SD-Arg+60mg/l刀豆氨酸平板培養基或者SD+300mg/l刀豆氨酸平板培養基中并涂布,將3天后出現的菌落數量作為細胞存活數進行計數。將SD平板中的生存菌落數作為總細胞數,將刀豆氨酸平板中的生存菌落數量作為抗性突變株數,計算并評價突變率。對突變導入部位而言,通過菌落PCR法擴增包含各株的靶標基因區域的DNA片段,然后進行DNA測序,以酵母基因組數據庫(http://www.yeastgenome.org/)的序列為基礎進行比對分析,并進行鑒定。
<核酸操作>
對DNA而言,利用PCR法、限制酶處理、接合、Gibson Assembly法/人工化學合成中的任一種方法進行加工、構建。對質粒而言,作為酵母-大腸桿菌穿梭載體,使用亮氨酸選擇用的pRS315、及尿嘧啶選擇用的pRS426作為主鏈。對質粒而言,利用大腸桿菌株XL-10 gold或DH5α進行擴增,通過乙酸鋰法導入酵母。
<構建體>
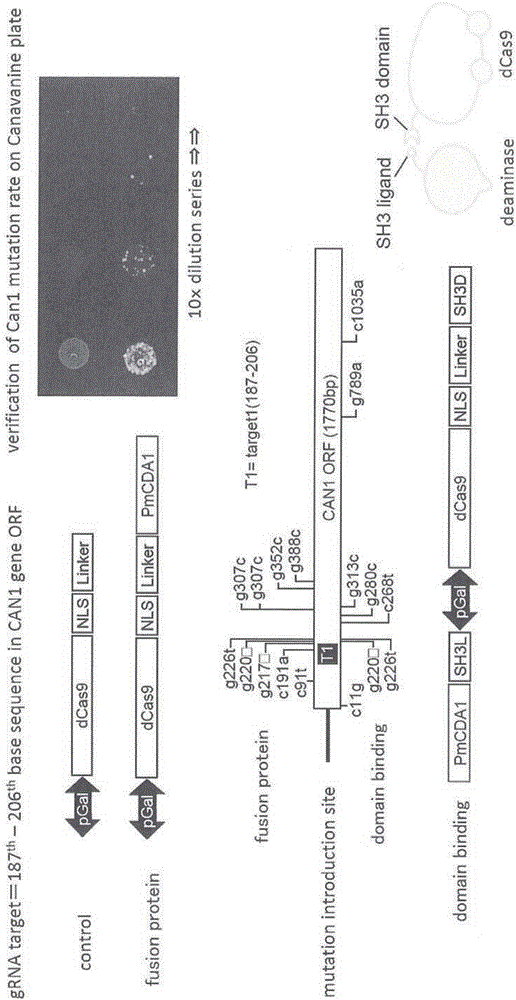
對誘導表達而言,使用了為半乳糖誘導型且為雙向啟動子的芽殖酵母pGal1/10(序列編號8)。將核躍遷信號(Nuclear transition signal)(ccc aag aag aag agg aag gtg;序列編號9(PKKKRV;編碼序列編號10))添加至已最優化為真核表達用的密碼子的源自化膿性鏈球菌(Streptococcus pyogenes)的Cas9基因ORF(序列編號5),將添加而成的產物連至啟動子下游,通過接頭序列與脫氨酶基因(源自七腮鰻(Petromyzon marinus)的PmCDA1或源自人的hAID)的ORF(序列編號1或3)相連,以融合蛋白的形式進行表達。作為接頭序列,雖然選擇、組合使用了GS接頭(ggt gga gga ggt tct;序列編號11(GGGGS;編碼序列編號12)的重復)、Flag標簽(gac tat aag gac cacgac gga gac tac aag gat cat gat att gat tac aaa gac gat gac gat aag;序列編號13(DYKDHDGDYKDHDIDYKDDDDK;編碼序列編號14))、Strep-標簽(tgg agc cac ccg cag ttc gaa aaa;序列編號15(WSHPQFEK;編碼序列編號16))、其它結構域,但這里,特別使用了2xGS、SH3結構域(序列編號17及18)、Flag標簽相連而成的。作為終止子,與源自芽殖酵母的ADH1終止子(序列編號19)及Top2終止子(序列編號20)相連。另外,在結構域結合方式中,將Cas9基因ORF通過2xGS接頭與SH3結構域相連并作為一個蛋白質,將SH3ligand序列(序列編號21及22)添加至脫氨酶并作為另一蛋白質,使其連接至Gal1/10啟動子的兩個方向并同時表達。將它們裝配至pRS315質粒。
為了除去各自單側的DNA鏈的切割能力,向Cas9中導入了將第10位的天冬氨酸轉變為丙氨酸的突變(D10A,對應DNA序列突變a29c)、及第840位的組氨酸轉變為丙氨酸的突變(H840A,對應DNA序列突變ca2518gc)。
對gRNA而言,以與tracrRNA(源自Streptococcus pyogenes;序列編號7)的嵌合結構的形式配置在SNR52啟動子(序列編號23)和Sup4終止子(序列編號24)之間,裝配至pRS426質粒。作為gRNA靶堿基序列,使用了CAN1基因ORF的187-206(gatacgttctctatggagga;序列編號25)(target1)、786-805(ttggagaaacccaggtgcct;序列編號26)(target3)、793-812(aacccaggtgcctggggtcc;序列編號27)(target4)、563-582(ttggccaagtcattcaattt;序列編號28)(target2)及767-786的互補鏈序列(ataacggaatccaactgggc;序列編號29)(target5r)。在同時表達多個靶標的情況下,可以將從啟動子到終止子的序列作為1組,并將多個組裝配至同一質粒。與Cas9-脫氨酶表達用質粒一起導入細胞,并使其在細胞內表達,形成gRNA-tracrRNA與Cas9-脫氨酶的復合體。
實施例1利用將CRISPR-Cas的DNA序列識別能力與脫氨酶PmCDA1連接來修飾基因組序列
為了試驗利用了脫氨酶和CRISPR-Cas的核酸序列識別能力的本發明的基因組序列的修飾技術的效果,嘗試了向編碼刀豆氨酸轉運蛋白的CAN1基因導入突變,使得由于基因缺陷而產生刀豆氨酸抗性。使用與CAN1基因ORF的187-206(target1)互補的序列作為gRNA,構建用該gRNA與源自Streptococcus pyogenes的tracrRNA連接而成的嵌合RNA的表達載體,構建表達向源自Streptococcus pyogenes的Cas9(SpCas9)導入突變(D10A及H840A)從而喪失了核酸酶活性的dCas9,與作為脫氨酶的源自七腮鰻的PmCDA1進行融合而成的蛋白質的載體,并利用乙酸鋰法導入芽殖酵母內,使它們共表達。結果示于圖2。在含有刀豆氨酸的SD平板上培養,則僅導入并表達gRNA-tracrRNA和dCas9-PmCDA1的細胞形成了菌落。選取抗性菌落并測序了CAN1基因區域的序列,結果確認在靶核苷酸序列(target1)內及其附近導入了突變。
實施例2大幅減輕副作用、毒性
在常規型的Cas9及其它人工核酸酶(ZFN、TALEN)中,如果以基因組內的序列為靶標,則產生認為由染色體的無規律切割引起的增殖障礙和細胞死亡。該影響在很多的微生物、原核生物中是特別致命的,阻礙了適用性。
因此,為了驗證本發明的基因組序列的修飾技術的安全性及對細胞的毒性,進行了常規型的CRISPR-Cas9的比較實驗。通過以CAN1基因內序列(target 3、4)作為gRNA靶標,從半乳糖表達誘導開始后立即到誘導后6小時的生存細胞在SD平板上的菌落形成能力進行了測定。結果示于圖3。在常規型Cas9中,由于增殖受到阻礙而誘導細胞死亡,因此細胞存活數減少,與此相對,在本技術(nCas9D10A-PmCDA1)中,細胞可以持續增殖,細胞存活數大幅增加。
實施例3不同結合類型的使用
檢查了如果Cas9和脫氨酶不是融合蛋白形式,而是通過結合結構域和其配體而形成核酸修飾酶復合體,是否也能進行向靶向基因的突變導入。使用了實施例1使用的dCas9作為Cas9,代替PmCDA1使用了人AID作為脫氨酶,將前者與SH3結構域、后者與其結合配體融合,制作成圖4所示的各種構建體。另外,使用CAN1基因內序列(target 4、5r)作為gRNA靶標。將它們的構建體導入芽殖酵母。其結果,即使在通過結合結構域將dCas9與脫氨酶結合的情況下,也向CAN1基因的靶向位點有效地導入了突變(圖4)。通過向dCas9導入多個結合結構域,突變導入效率顯著提高。主要的突變導入部位為ORF的第782位(g782c)。
實施例4由使用切口酶(Nickase)引起的高效化和突變模式的變化
代替dCas9使用了僅切割與gRNA互補的鏈的D10A突變體nCas9(D10A)、或僅切割與gRNA互補的鏈的相反鏈的H840A突變體nCas9(H840A),除此以外,與實施例1同樣地進行,向CAN1基因導入突變,檢查含有刀豆氨酸的SD平板上產生的菌落的CAN1基因區域的序列,結果發現在前者(nCas9(D10A))中,效率比dCas9升高(圖5),并且突變集中在靶標序列的中央(圖6)。因此,根據該方法可以進行點性突變導入。另一方面,結果發現,在后者(nCas9(H840A))中,效率比dCas9升高(圖5),并且在從靶向的核苷酸到達幾百個堿基的區域導入了多個隨機突變(圖6)。
即使改變靶核苷酸序列,也可以確認同樣顯著的突變導入,在使用CRISPR-Cas9系統和胞苷脫氨酶的本基因組編輯體系中,確認到如表1所示,在存在于從靶核苷酸序列(20bp)的5’側到2~5bp左右的范圍內的胞嘧啶優先進行脫氨基化。因此,通過基于該規律性設定靶核苷酸序列,并進一步與nCas9(D10A)組合,能夠進行1個核苷酸單位的精密的基因組編輯。另一方面,如果使用nCas9(H840A),則可以在靶核苷酸序列的附近的幾百個bp左右的范圍同時插入多個突變。進一步,存在通過改變脫氨酶的結合類型,而進一步改變部位特異性的可能性。
根據這些結果顯示,可以根據目的不同,分別使用Cas9蛋白質的種類。
[表1]
實施例5通過將鄰近的多個DNA序列作為靶標,效率協同性地升高
與單獨的靶標相比,通過同時使用鄰近的多個靶標,效率大幅升高(圖7)。事實上,10~20%的細胞具有刀豆氨酸抗性突變(target3、4)。圖中的gRNA1和gRNA2分別以target3和target4為靶。使用PmCDA1作為脫氨酶。確認了不僅在序列的一部分重復的情況下(target3、4)產生該效果,在分離600bp的情況下(target1、3)也產生該效果。另外,在DNA序列為相同方向(target3、4),和對向(target4、5)(圖4)的兩種情況下均產生該效果。
實施例6不需要選擇標識的基因修飾
對于實施例5中以target3和target4為靶的細胞(Target3、4),從非選擇性(不含刀豆氨酸)的平板(不含有Leu及Ura的SD平板)上長出的菌落中,隨機選出10個測序了CAN1基因區域的序列。結果,檢查的全部菌落均在CAN1基因的靶向位點導入了突變(圖8)。即,根據本發明,如果選擇適當的靶向序列,可以期待在表達的細胞的基本全部中發生編輯。因此,不需要常規基因操作中必需的標識基因的插入和選擇。其在使基因操作顯著地變得簡便的同時,由于不是外來DNA重組生物,因此對作物育種等的適用性大幅變寬。
在以下的實施例中,對與實施例1~6共同的實驗技術,與上述同樣地進行。
實施例7多個部位(不同基因)的同時編輯
在通常的基因操作方法中,由于有各種限制,因此通常在一次操作中只能進行一個位置的突變。因此,試驗了根據本發明的方法是否能能夠同時進行多個部位的突變操作。
將芽殖酵母株BY4741株的Ade1基因ORF的3-22位作為第1靶核苷酸序列(Ade1 target5:GTCAATTACGAAGACTGAAC;序列編號30),將Can1基因ORF的767-786位(互補鏈)作為第2靶核苷酸序列(Can1target8(786-767;ATAACGGAATCCAACTGGGC;序列編號29),使分別包含編碼與它們互補的核苷酸序列的兩種gRNA與tracrRNA(序列編號7)的嵌合RNA的DNA兩者,搭載于同一質粒(pRS426),并與包含編碼突變Cas9與PmCDA1的融合蛋白的核酸的質粒nCas9 D10A-PmCDA1一起,導入BY4741株并使其表達,驗證對兩個基因的突變導入。以保持質粒的SD撤除成分培養基(Dropout,尿嘧啶及亮氨酸缺陷;SD-UL)為基礎,培養細胞。適當稀釋細胞,涂布至SD-UL及添加刀豆氨酸的培養基,形成菌落。于28℃培養2天后,觀察菌落,分別計數由ade1突變引起的紅色菌落的發生率,和在刀豆氨酸培養基中的存活率。將結果示于表2中。
[表2]
作為表型,向Ade1基因及Can1基因的兩者均導入了突變的比例高,為約31%。
下面,利用PCR對SD-UL培養基上的菌落進行擴增并測序分析。對包含Ade1、Can1各自的ORF區域進行拓寬,獲得靶標序列周邊500b左右的序列的測序信息。具體而言,分析了紅菌落5個和白菌落5個,結果全部紅菌落的Ade1基因ORF的第5位的C轉變為G,全部白菌落的第5位的C轉變為T(圖9)。雖然靶標的突變率為100%,但由于作為目的在于基因破壞的突變率,需要使第5位的C變為G而成為終止密碼子,因此,認為期望的突變率為50%。相同地,對于Can1基因,雖然確認在全部克隆中,發生了ORF的第782位的G的突變(圖9),但由于僅變為C的突變能提供刀豆氨酸抗性,因此,作為期望的突變率為70%。在檢查的范圍內,對于兩種基因同時得到期望的突變的比例為40%(10個克隆中占4個克隆),事實上得到了較高的效率。
實施例8多倍體基因組的編輯
雖然很多生物具有二倍體以上的基因組倍數性,但在常規的突變導入方法中,作為原則僅向同源染色體的一條中導入突變而形成雜合基因型,因此,如果不是優勢突變,就得不到需要的特性,而純合化則耗時耗力。因此,試驗了根據本發明的技術,是否能夠向基因組內的同源染色體上的全部靶等位基因內導入突變。
即,在二倍體株的芽殖酵母YPH501株中,進行Ade1及Can1基因的同時編輯。由于這些基因突變的表型(紅色菌落及刀豆氨酸抗性)是劣勢表型,因此,如果不是導入了基因的兩方的突變(純合突變),則這些表型不會表現。
將Ade1基因ORF的1173-1154位(互補鏈)(Ade1 target1:GTCAATAGGATCCCCTTTT;序列編號31)或者3-22位(Ade1 target5:GTCAATTACGAAGACTGAAC;序列編號30)作為第1靶核苷酸序列,將Can1基因ORF的767-786位(互補鏈)作為第2靶核苷酸序列(Can1 target8:ATAACGGAATCCAACTGGGC;序列編號29),使分別包含編碼與它們互補的核苷酸序列的兩種gRNA與tracrRNA(序列編號7)的嵌合RNA的DNA兩者,搭載于同一質粒(pRS426),并與包含編碼突變Cas9與PmCDA1的融合蛋白的核酸的質粒nCas9 D10A-PmCDA1一起,導入BY4741株并使其表達,驗證了向各基因的突變導入。
菌落計數的結果可知,可以以高概率(40%~70%)獲得每個表型的特征(圖10A)。
進一步,為了確認突變,進行了白菌落和紅菌落各自的Ade1靶區域的測序,結果在白菌落中靶向部位確認到顯示雜合突變的測序信號的重疊(圖10B的上圖,↓處G和T的信號發生重疊)。確認由于發生雜合突變而沒有出現表型。另一方面,在紅菌落中,確認了沒有重疊的信號,為純合突變(圖10B下圖,↓處為T的信號)。
實施例9大腸桿菌中的基因組編輯
在本實施例中,驗證在作為細菌的代表性模型生物的大腸桿菌中,本技術有效地發揮功能。特別是,由于在細菌中,常規的核酸酶型的基因組編輯技術是致死的而難以適用,因此強調本技術的優越性。另外,與作為真核生物的模型細胞的酵母一起,顯示無論在原核還是真核生物種類中均可廣泛適用。
向包含雙向啟動子區域的Streptococcus pyogenes Cas9基因導入D10A及H840A的氨基酸突變(dCas9),構建通過接頭序列以與PmCDA1的融合蛋白的形式表達的構建體,進一步制作了同時搭載編碼與各靶核苷酸序列互補的序列的嵌合gRNA的質粒(全長核苷酸序列示于序列編號32。向該序列中n20的部分導入與各靶標序列互補的序列)(圖11A)。
首先,將以大腸桿菌galK基因ORF的426-445位(T CAA TGG GCT AAC TAC GTT C;序列編號33)為靶核苷酸序列導入的質粒,利用鈣法或電穿孔法轉化至各種大腸桿菌株(XL10-gold、DH5a、MG1655、BW25113),添加SOC培養基恢復培養一夜,然后通過含有氨芐青霉素LB培養基選擇保持質粒的細胞,使其形成菌落。通過由菌落PCR直接測序,驗證了突變導入。結果示于圖11B。
隨機選出獨立的菌落(1~3),進行測序分析,結果確認了有60%以上的概率將ORF的427位的C轉變為T(克隆2、3),從而產生終止密碼子(TAA)的基因發生破壞。
下面,以作為必需基因的rpoB基因ORF的1530-1549堿基區域的互補序列(5’-GGTCCATAAACTGAGACAGC-3’;序列編號34)作為靶標,通過與上述同樣的方法導入特定的點突變,嘗試了對大腸桿菌賦予利福平抗性功能。對利用含有非選擇性培養基(none)、25μg/ml利福平(Rif25)及50μg/ml利福平(Rif50)的培養基選擇出的菌落進行測序分析,結果確認,通過將ORF的1546位的G轉變為A,導入了將Asp(GAC)變為Asn(AAC)的氨基酸突變,賦予了利福平抗性(圖11C,上圖)。將經轉化處理后的細胞懸濁液的10倍稀釋系列,分別點在含有非選擇性培養基(none)、25μg/ml利福平(Rif25)及50μg/ml利福平(Rif50)培養基上并培養,結果可見以約10%的頻率得到了利福平抗性株(圖11C,下圖)。
像這樣,根據本技術,不僅可以破壞基因,還可以通過特定的點突變來添加新的功能。另外,在直接編輯必需基因的方面,也顯示了本技術的優越性。
實施例10利用gRNA長度來調節編輯堿基部位
以往,針對靶核苷酸序列的gRNA長度以20b為基礎,并以存在于從其5’末端到2~5b(PAM序列的上游15~19b)的部位的胞嘧啶(或相反鏈的鳥嘌呤)作為突變靶標。檢查了如果表達使該gRNA長度改變的核酸,與其對應的作為靶標的堿基的部位是否移動(圖12A)。
將利用大腸桿菌進行的實驗例示于圖12B。檢索在大腸桿菌基因組上的胞嘧啶多個排列的部位,使用作為putative ABC-轉運蛋白的gsiA基因進行實驗。檢查了將靶的長度變為24bp、22bp、20bp、18bp時的取代的胞嘧啶,結果在20bp(標準長度)的情況下,第898、899位的胞嘧啶被取代為胸腺嘧啶。可知,在靶向部位長于20bp的情況下,第896、897位的胞嘧啶也被取代,在靶向部位較短的情況下,第900、901位的胞嘧啶也可被取代,實際上可以通過改變gRNA的長度使靶向部位移動。
實施例11溫度依賴性基因組編輯質粒的開發
將本發明的核酸修飾酶復合體設計為利用高溫條件進行表達誘導的方式的質粒。目的在于通過限定表達狀態進行控制而將效率最優化,并且減輕副作用(產生宿主的生長障礙、突變導入效率不穩定、對與靶標不同的部位的突變)。同時,通過組合使用利用高溫停止質粒的復制的方法,意圖在編輯后同時且容易地進行質粒的除去。以下示出實驗的細節。
將溫度敏感性質粒pSC101-Rep101系(pSC101 ori的序列示于序列編號35,溫度敏感性Rep101的序列示于序列編號36)作為主鏈(backbone),使用溫度敏感性的λ阻遏物(cI857)系統表達誘導。向基因組編輯用的λ阻遏物導入RecA抗性的G113E突變,使得在SOS應答下也正常發揮功能(序列編號37)。將dCas9-PmCDA1(序列編號38)連接至Right Operator(序列編號39)、gRNA(序列編號40)連接至Left Operator(序列編號41)的下游,使得控制表達(構建成的表達載體的所有核苷酸序列示于序列編號42)。在30℃以下培養時期,由于分別抑制了gRNA、dCas9-PmCDA1的轉錄、表達,因此細胞可以正常增殖。當在37℃以上培養時,誘導gRNA的轉錄和dCas9-PmCDA1的表達,而與此同時抑制質粒的復制,因此,一過性地地供給對基因組編輯所必須的核酸修飾酶復合體,可以在編輯之后容易地除去質粒(圖13)。
堿基取代的具體的方案示于圖14。
進行質粒構建時的培養溫度設為28℃左右,首先建立保持所需質粒的大腸桿菌菌落。下面,直接使用菌落,或當菌株改變的情況下提取質粒,再次進行向目標菌株的轉化,使用得到的菌落,在28℃液體培養一夜。隨后,利用培養基稀釋,在42℃進行從1小時到一夜左右的誘導培養,適當稀釋細胞懸濁液并鋪展或點在板上,得到單個菌落。
作為驗證實驗,進行了向作為必需基因的rpoB的點突變導入。作為RNA聚合酶的構成因素之一的rpoB如果缺失或失能,則大腸桿菌死亡。一方面,已知通過向特定的部位引入點突變,獲得相對于作為抗生素的利福平(Rif)的抗性。因此,為了導入這樣的點突變而選定靶向部位,并進行測定。
結果示于圖15。左上圖的左為LB(添加氯霉素)平板,右為添加了利福平的LB(添加氯霉素)平板,利用這些平板準備了在28℃*42℃培養時有或無氯霉素的樣品。雖然在28℃培養的情況下,Rif抗性的比例較低,但在42℃培養的情況下,以極高的效率得到了利福平抗性。對實際LB上得到的菌落(非選擇)的8個菌落測序,結果可知,在42℃培養的菌株有60%以上的比例的第1546位的鳥嘌呤(g)被取代為腺嘌呤(A)(左下及右上圖)。可知在實際的測序譜(右下圖)中,堿基也被完全取代了。
類似地,進行對參與半乳糖代謝的因素之一的galK的堿基取代。由于對大腸桿菌而言,galK通過代謝作為半乳糖的類似物的2-脫氧半乳糖(2DOG)而致死,因此,將其用作選擇方法。分別設定了靶向位點,使得對target 8引起錯義突變,target 12成為終止密碼子(圖16右下)。
結果示于圖16。在左上和左下圖中,左側為LB(添加氯霉素)平板,右側為添加了2-DOG的LB(添加氯霉素)平板,利用這些平板準備了在28℃、42℃培養時有或無氯霉素的樣品。在target 8中,在添加2-DOG的平板中僅輕微產生菌落(左上圖),但對LB上的菌落(紅框)3個菌落測序,發現全部菌落中的第61位胞嘧啶(C)被取代為胸腺嘧啶(T)(右上)。推測該突變不足以造成galK的失能。另一方面,在target 12中,在28℃、42℃培養的任一情況下,在2-DOG添加板中均得到菌落(左下圖)。對LB上的3個菌落進行測序,結果,全部菌落中的第271位胞嘧啶被胸腺嘧啶取代(右下)。顯示即使在這樣的不同的靶中,也可以更穩定且高效地導入突變。
包含在此提到的專利及專利申請說明書的全部出版物中記載的內容,通過在此引用,將其整體與已寫明的程度相同地并入本說明書。
本申請分別基于2014年3月5日及同年9月30日在日本提交的日本特愿2014-43348及日本特愿2014-201859,通過引用將其內容全部并入本說明書。
工業實用性
根據本發明,可以不伴隨外源DNA的插入也不伴隨DNA雙鏈的切割,安全地向任意生物種類導入部位特異性的突變。另外,能夠將突變導入的范圍在從一個堿基的針點(pin point)至幾百個堿基的范圍進行廣泛設定,也可以應用于利用向迄今為止幾乎不可能進行的特定的限定區域隨機導入突變的局部進化誘導,極其有用。
SEQUENCE LISTING
<110> 國立大學法人神戶大學(NATIONAL UNIVERSITY CORPORATION KOBE UNIVERSITY)
<120> 用于特異性轉變靶向DNA序列的核酸堿基的基因組序列的修飾方法、及其使用的分子復合體(Method of modifying genomic sequence comprising specifically
converting nucleobase in targeted DNA sequence and molecular
complex used therefor)
<130> 092301
<150> JP 2014-043348
<151> 2014-03-05
<150> JP 2014-201859
<151> 2014-09-30
<160> 42
<170> PatentIn version 3.5
<210> 1
<211> 624
<212> DNA
<213> 七腮鰻(Petromyzon marinus)
<220>
<221> CDS
<222> (1)..(624)
<400> 1
atg acc gac gct gag tac gtg aga atc cat gag aag ttg gac atc tac 48
Met Thr Asp Ala Glu Tyr Val Arg Ile His Glu Lys Leu Asp Ile Tyr
1 5 10 15
acg ttt aag aaa cag ttt ttc aac aac aaa aaa tcc gtg tcg cat aga 96
Thr Phe Lys Lys Gln Phe Phe Asn Asn Lys Lys Ser Val Ser His Arg
20 25 30
tgc tac gtt ctc ttt gaa tta aaa cga cgg ggt gaa cgt aga gcg tgt 144
Cys Tyr Val Leu Phe Glu Leu Lys Arg Arg Gly Glu Arg Arg Ala Cys
35 40 45
ttt tgg ggc tat gct gtg aat aaa cca cag agc ggg aca gaa cgt ggc 192
Phe Trp Gly Tyr Ala Val Asn Lys Pro Gln Ser Gly Thr Glu Arg Gly
50 55 60
att cac gcc gaa atc ttt agc att aga aaa gtc gaa gaa tac ctg cgc 240
Ile His Ala Glu Ile Phe Ser Ile Arg Lys Val Glu Glu Tyr Leu Arg
65 70 75 80
gac aac ccc gga caa ttc acg ata aat tgg tac tca tcc tgg agt cct 288
Asp Asn Pro Gly Gln Phe Thr Ile Asn Trp Tyr Ser Ser Trp Ser Pro
85 90 95
tgt gca gat tgc gct gaa aag atc tta gaa tgg tat aac cag gag ctg 336
Cys Ala Asp Cys Ala Glu Lys Ile Leu Glu Trp Tyr Asn Gln Glu Leu
100 105 110
cgg ggg aac ggc cac act ttg aaa atc tgg gct tgc aaa ctc tat tac 384
Arg Gly Asn Gly His Thr Leu Lys Ile Trp Ala Cys Lys Leu Tyr Tyr
115 120 125
gag aaa aat gcg agg aat caa att ggg ctg tgg aac ctc aga gat aac 432
Glu Lys Asn Ala Arg Asn Gln Ile Gly Leu Trp Asn Leu Arg Asp Asn
130 135 140
ggg gtt ggg ttg aat gta atg gta agt gaa cac tac caa tgt tgc agg 480
Gly Val Gly Leu Asn Val Met Val Ser Glu His Tyr Gln Cys Cys Arg
145 150 155 160
aaa ata ttc atc caa tcg tcg cac aat caa ttg aat gag aat aga tgg 528
Lys Ile Phe Ile Gln Ser Ser His Asn Gln Leu Asn Glu Asn Arg Trp
165 170 175
ctt gag aag act ttg aag cga gct gaa aaa cga cgg agc gag ttg tcc 576
Leu Glu Lys Thr Leu Lys Arg Ala Glu Lys Arg Arg Ser Glu Leu Ser
180 185 190
att atg att cag gta aaa ata ctc cac acc act aag agt cct gct gtt 624
Ile Met Ile Gln Val Lys Ile Leu His Thr Thr Lys Ser Pro Ala Val
195 200 205
<210> 2
<211> 208
<212> PRT
<213> 七腮鰻
<400> 2
Met Thr Asp Ala Glu Tyr Val Arg Ile His Glu Lys Leu Asp Ile Tyr
1 5 10 15
Thr Phe Lys Lys Gln Phe Phe Asn Asn Lys Lys Ser Val Ser His Arg
20 25 30
Cys Tyr Val Leu Phe Glu Leu Lys Arg Arg Gly Glu Arg Arg Ala Cys
35 40 45
Phe Trp Gly Tyr Ala Val Asn Lys Pro Gln Ser Gly Thr Glu Arg Gly
50 55 60
Ile His Ala Glu Ile Phe Ser Ile Arg Lys Val Glu Glu Tyr Leu Arg
65 70 75 80
Asp Asn Pro Gly Gln Phe Thr Ile Asn Trp Tyr Ser Ser Trp Ser Pro
85 90 95
Cys Ala Asp Cys Ala Glu Lys Ile Leu Glu Trp Tyr Asn Gln Glu Leu
100 105 110
Arg Gly Asn Gly His Thr Leu Lys Ile Trp Ala Cys Lys Leu Tyr Tyr
115 120 125
Glu Lys Asn Ala Arg Asn Gln Ile Gly Leu Trp Asn Leu Arg Asp Asn
130 135 140
Gly Val Gly Leu Asn Val Met Val Ser Glu His Tyr Gln Cys Cys Arg
145 150 155 160
Lys Ile Phe Ile Gln Ser Ser His Asn Gln Leu Asn Glu Asn Arg Trp
165 170 175
Leu Glu Lys Thr Leu Lys Arg Ala Glu Lys Arg Arg Ser Glu Leu Ser
180 185 190
Ile Met Ile Gln Val Lys Ile Leu His Thr Thr Lys Ser Pro Ala Val
195 200 205
<210> 3
<211> 600
<212> DNA
<213> 人(Homo sapiens)
<220>
<221> CDS
<222> (1)..(600)
<400> 3
atg gac agc ctc ttg atg aac cgg agg aag ttt ctt tac caa ttc aaa 48
Met Asp Ser Leu Leu Met Asn Arg Arg Lys Phe Leu Tyr Gln Phe Lys
1 5 10 15
aat gtc cgc tgg gct aag ggt cgg cgt gag acc tac ctg tgc tac gta 96
Asn Val Arg Trp Ala Lys Gly Arg Arg Glu Thr Tyr Leu Cys Tyr Val
20 25 30
gtg aag agg cgt gac agt gct aca tcc ttt tca ctg gac ttt ggt tat 144
Val Lys Arg Arg Asp Ser Ala Thr Ser Phe Ser Leu Asp Phe Gly Tyr
35 40 45
ctt cgc aat aag aac ggc tgc cac gtg gaa ttg ctc ttc ctc cgc tac 192
Leu Arg Asn Lys Asn Gly Cys His Val Glu Leu Leu Phe Leu Arg Tyr
50 55 60
atc tcg gac tgg gac cta gac cct ggc cgc tgc tac cgc gtc acc tgg 240
Ile Ser Asp Trp Asp Leu Asp Pro Gly Arg Cys Tyr Arg Val Thr Trp
65 70 75 80
ttc acc tcc tgg agc ccc tgc tac gac tgt gcc cga cat gtg gcc gac 288
Phe Thr Ser Trp Ser Pro Cys Tyr Asp Cys Ala Arg His Val Ala Asp
85 90 95
ttt ctg cga ggg aac ccc tac ctc agt ctg agg atc ttc acc gcg cgc 336
Phe Leu Arg Gly Asn Pro Tyr Leu Ser Leu Arg Ile Phe Thr Ala Arg
100 105 110
ctc tac ttc tgt gag gac cgc aag gct gag ccc gag ggg ctg cgg cgg 384
Leu Tyr Phe Cys Glu Asp Arg Lys Ala Glu Pro Glu Gly Leu Arg Arg
115 120 125
ctg cac cgc gcc ggg gtg caa ata gcc atc atg acc ttc aaa gat tat 432
Leu His Arg Ala Gly Val Gln Ile Ala Ile Met Thr Phe Lys Asp Tyr
130 135 140
ttt tac tgc tgg aat act ttt gta gaa aac cat gaa aga act ttc aaa 480
Phe Tyr Cys Trp Asn Thr Phe Val Glu Asn His Glu Arg Thr Phe Lys
145 150 155 160
gcc tgg gaa ggg ctg cat gaa aat tca gtt cgt ctc tcc aga cag ctt 528
Ala Trp Glu Gly Leu His Glu Asn Ser Val Arg Leu Ser Arg Gln Leu
165 170 175
cgg cgc atc ctt ttg ccc ctg tat gag gtt gat gac tta cga gac gca 576
Arg Arg Ile Leu Leu Pro Leu Tyr Glu Val Asp Asp Leu Arg Asp Ala
180 185 190
ttt cgt act ttg gga ctt ctc gac 600
Phe Arg Thr Leu Gly Leu Leu Asp
195 200
<210> 4
<211> 200
<212> PRT
<213> 人
<400> 4
Met Asp Ser Leu Leu Met Asn Arg Arg Lys Phe Leu Tyr Gln Phe Lys
1 5 10 15
Asn Val Arg Trp Ala Lys Gly Arg Arg Glu Thr Tyr Leu Cys Tyr Val
20 25 30
Val Lys Arg Arg Asp Ser Ala Thr Ser Phe Ser Leu Asp Phe Gly Tyr
35 40 45
Leu Arg Asn Lys Asn Gly Cys His Val Glu Leu Leu Phe Leu Arg Tyr
50 55 60
Ile Ser Asp Trp Asp Leu Asp Pro Gly Arg Cys Tyr Arg Val Thr Trp
65 70 75 80
Phe Thr Ser Trp Ser Pro Cys Tyr Asp Cys Ala Arg His Val Ala Asp
85 90 95
Phe Leu Arg Gly Asn Pro Tyr Leu Ser Leu Arg Ile Phe Thr Ala Arg
100 105 110
Leu Tyr Phe Cys Glu Asp Arg Lys Ala Glu Pro Glu Gly Leu Arg Arg
115 120 125
Leu His Arg Ala Gly Val Gln Ile Ala Ile Met Thr Phe Lys Asp Tyr
130 135 140
Phe Tyr Cys Trp Asn Thr Phe Val Glu Asn His Glu Arg Thr Phe Lys
145 150 155 160
Ala Trp Glu Gly Leu His Glu Asn Ser Val Arg Leu Ser Arg Gln Leu
165 170 175
Arg Arg Ile Leu Leu Pro Leu Tyr Glu Val Asp Asp Leu Arg Asp Ala
180 185 190
Phe Arg Thr Leu Gly Leu Leu Asp
195 200
<210> 5
<211> 4116
<212> DNA
<213> 人工序列
<220>
<223> 化膿性鏈球菌來源的Cas9 CDS, 優化用于真核生物表達
<220>
<221> CDS
<222> (1)..(4116)
<400> 5
atg gac aag aag tac tcc att ggg ctc gat atc ggc aca aac agc gtc 48
Met Asp Lys Lys Tyr Ser Ile Gly Leu Asp Ile Gly Thr Asn Ser Val
1 5 10 15
ggt tgg gcc gtc att acg gac gag tac aag gtg ccg agc aaa aaa ttc 96
Gly Trp Ala Val Ile Thr Asp Glu Tyr Lys Val Pro Ser Lys Lys Phe
20 25 30
aaa gtt ctg ggc aat acc gat cgc cac agc ata aag aag aac ctc att 144
Lys Val Leu Gly Asn Thr Asp Arg His Ser Ile Lys Lys Asn Leu Ile
35 40 45
ggc gcc ctc ctg ttc gac tcc ggg gag acg gcc gaa gcc acg cgg ctc 192
Gly Ala Leu Leu Phe Asp Ser Gly Glu Thr Ala Glu Ala Thr Arg Leu
50 55 60
aaa aga aca gca cgg cgc aga tat acc cgc aga aag aat cgg atc tgc 240
Lys Arg Thr Ala Arg Arg Arg Tyr Thr Arg Arg Lys Asn Arg Ile Cys
65 70 75 80
tac ctg cag gag atc ttt agt aat gag atg gct aag gtg gat gac tct 288
Tyr Leu Gln Glu Ile Phe Ser Asn Glu Met Ala Lys Val Asp Asp Ser
85 90 95
ttc ttc cat agg ctg gag gag tcc ttt ttg gtg gag gag gat aaa aag 336
Phe Phe His Arg Leu Glu Glu Ser Phe Leu Val Glu Glu Asp Lys Lys
100 105 110
cac gag cgc cac cca atc ttt ggc aat atc gtg gac gag gtg gcg tac 384
His Glu Arg His Pro Ile Phe Gly Asn Ile Val Asp Glu Val Ala Tyr
115 120 125
cat gaa aag tac cca acc ata tat cat ctg agg aag aag ctt gta gac 432
His Glu Lys Tyr Pro Thr Ile Tyr His Leu Arg Lys Lys Leu Val Asp
130 135 140
agt act gat aag gct gac ttg cgg ttg atc tat ctc gcg ctg gcg cat 480
Ser Thr Asp Lys Ala Asp Leu Arg Leu Ile Tyr Leu Ala Leu Ala His
145 150 155 160
atg atc aaa ttt cgg gga cac ttc ctc atc gag ggg gac ctg aac cca 528
Met Ile Lys Phe Arg Gly His Phe Leu Ile Glu Gly Asp Leu Asn Pro
165 170 175
gac aac agc gat gtc gac aaa ctc ttt atc caa ctg gtt cag act tac 576
Asp Asn Ser Asp Val Asp Lys Leu Phe Ile Gln Leu Val Gln Thr Tyr
180 185 190
aat cag ctt ttc gaa gag aac ccg atc aac gca tcc gga gtt gac gcc 624
Asn Gln Leu Phe Glu Glu Asn Pro Ile Asn Ala Ser Gly Val Asp Ala
195 200 205
aaa gca atc ctg agc gct agg ctg tcc aaa tcc cgg cgg ctc gaa aac 672
Lys Ala Ile Leu Ser Ala Arg Leu Ser Lys Ser Arg Arg Leu Glu Asn
210 215 220
ctc atc gca cag ctc cct ggg gag aag aag aac ggc ctg ttt ggt aat 720
Leu Ile Ala Gln Leu Pro Gly Glu Lys Lys Asn Gly Leu Phe Gly Asn
225 230 235 240
ctt atc gcc ctg tca ctc ggg ctg acc ccc aac ttt aaa tct aac ttc 768
Leu Ile Ala Leu Ser Leu Gly Leu Thr Pro Asn Phe Lys Ser Asn Phe
245 250 255
gac ctg gcc gaa gat gcc aag ctt caa ctg agc aaa gac acc tac gat 816
Asp Leu Ala Glu Asp Ala Lys Leu Gln Leu Ser Lys Asp Thr Tyr Asp
260 265 270
gat gat ctc gac aat ctg ctg gcc cag atc ggc gac cag tac gca gac 864
Asp Asp Leu Asp Asn Leu Leu Ala Gln Ile Gly Asp Gln Tyr Ala Asp
275 280 285
ctt ttt ttg gcg gca aag aac ctg tca gac gcc att ctg ctg agt gat 912
Leu Phe Leu Ala Ala Lys Asn Leu Ser Asp Ala Ile Leu Leu Ser Asp
290 295 300
att ctg cga gtg aac acg gag atc acc aaa gct ccg ctg agc gct agt 960
Ile Leu Arg Val Asn Thr Glu Ile Thr Lys Ala Pro Leu Ser Ala Ser
305 310 315 320
atg atc aag cgc tat gat gag cac cac caa gac ttg act ttg ctg aag 1008
Met Ile Lys Arg Tyr Asp Glu His His Gln Asp Leu Thr Leu Leu Lys
325 330 335
gcc ctt gtc aga cag caa ctg cct gag aag tac aag gaa att ttc ttc 1056
Ala Leu Val Arg Gln Gln Leu Pro Glu Lys Tyr Lys Glu Ile Phe Phe
340 345 350
gat cag tct aaa aat ggc tac gcc gga tac att gac ggc gga gca agc 1104
Asp Gln Ser Lys Asn Gly Tyr Ala Gly Tyr Ile Asp Gly Gly Ala Ser
355 360 365
cag gag gaa ttt tac aaa ttt att aag ccc atc ttg gaa aaa atg gac 1152
Gln Glu Glu Phe Tyr Lys Phe Ile Lys Pro Ile Leu Glu Lys Met Asp
370 375 380
ggc acc gag gag ctg ctg gta aag ctt aac aga gaa gat ctg ttg cgc 1200
Gly Thr Glu Glu Leu Leu Val Lys Leu Asn Arg Glu Asp Leu Leu Arg
385 390 395 400
aaa cag cgc act ttc gac aat gga agc atc ccc cac cag att cac ctg 1248
Lys Gln Arg Thr Phe Asp Asn Gly Ser Ile Pro His Gln Ile His Leu
405 410 415
ggc gaa ctg cac gct atc ctc agg cgg caa gag gat ttc tac ccc ttt 1296
Gly Glu Leu His Ala Ile Leu Arg Arg Gln Glu Asp Phe Tyr Pro Phe
420 425 430
ttg aaa gat aac agg gaa aag att gag aaa atc ctc aca ttt cgg ata 1344
Leu Lys Asp Asn Arg Glu Lys Ile Glu Lys Ile Leu Thr Phe Arg Ile
435 440 445
ccc tac tat gta ggc ccc ctc gcc cgg gga aat tcc aga ttc gcg tgg 1392
Pro Tyr Tyr Val Gly Pro Leu Ala Arg Gly Asn Ser Arg Phe Ala Trp
450 455 460
atg act cgc aaa tca gaa gag acc atc act ccc tgg aac ttc gag gaa 1440
Met Thr Arg Lys Ser Glu Glu Thr Ile Thr Pro Trp Asn Phe Glu Glu
465 470 475 480
gtc gtg gat aag ggg gcc tct gcc cag tcc ttc atc gaa agg atg act 1488
Val Val Asp Lys Gly Ala Ser Ala Gln Ser Phe Ile Glu Arg Met Thr
485 490 495
aac ttt gat aaa aat ctg cct aac gaa aag gtg ctt cct aaa cac tct 1536
Asn Phe Asp Lys Asn Leu Pro Asn Glu Lys Val Leu Pro Lys His Ser
500 505 510
ctg ctg tac gag tac ttc aca gtt tat aac gag ctc acc aag gtc aaa 1584
Leu Leu Tyr Glu Tyr Phe Thr Val Tyr Asn Glu Leu Thr Lys Val Lys
515 520 525
tac gtc aca gaa ggg atg aga aag cca gca ttc ctg tct gga gag cag 1632
Tyr Val Thr Glu Gly Met Arg Lys Pro Ala Phe Leu Ser Gly Glu Gln
530 535 540
aag aaa gct atc gtg gac ctc ctc ttc aag acg aac cgg aaa gtt acc 1680
Lys Lys Ala Ile Val Asp Leu Leu Phe Lys Thr Asn Arg Lys Val Thr
545 550 555 560
gtg aaa cag ctc aaa gaa gac tat ttc aaa aag att gaa tgt ttc gac 1728
Val Lys Gln Leu Lys Glu Asp Tyr Phe Lys Lys Ile Glu Cys Phe Asp
565 570 575
tct gtt gaa atc agc gga gtg gag gat cgc ttc aac gca tcc ctg gga 1776
Ser Val Glu Ile Ser Gly Val Glu Asp Arg Phe Asn Ala Ser Leu Gly
580 585 590
acg tat cac gat ctc ctg aaa atc att aaa gac aag gac ttc ctg gac 1824
Thr Tyr His Asp Leu Leu Lys Ile Ile Lys Asp Lys Asp Phe Leu Asp
595 600 605
aat gag gag aac gag gac att ctt gag gac att gtc ctc acc ctt acg 1872
Asn Glu Glu Asn Glu Asp Ile Leu Glu Asp Ile Val Leu Thr Leu Thr
610 615 620
ttg ttt gaa gat agg gag atg att gaa gaa cgc ttg aaa act tac gct 1920
Leu Phe Glu Asp Arg Glu Met Ile Glu Glu Arg Leu Lys Thr Tyr Ala
625 630 635 640
cat ctc ttc gac gac aaa gtc atg aaa cag ctc aag agg cgc cga tat 1968
His Leu Phe Asp Asp Lys Val Met Lys Gln Leu Lys Arg Arg Arg Tyr
645 650 655
aca gga tgg ggg cgg ctg tca aga aaa ctg atc aat ggg atc cga gac 2016
Thr Gly Trp Gly Arg Leu Ser Arg Lys Leu Ile Asn Gly Ile Arg Asp
660 665 670
aag cag agt gga aag aca atc ctg gat ttt ctt aag tcc gat gga ttt 2064
Lys Gln Ser Gly Lys Thr Ile Leu Asp Phe Leu Lys Ser Asp Gly Phe
675 680 685
gcc aac cgg aac ttc atg cag ttg atc cat gat gac tct ctc acc ttt 2112
Ala Asn Arg Asn Phe Met Gln Leu Ile His Asp Asp Ser Leu Thr Phe
690 695 700
aag gag gac atc cag aaa gca caa gtt tct ggc cag ggg gac agt ctt 2160
Lys Glu Asp Ile Gln Lys Ala Gln Val Ser Gly Gln Gly Asp Ser Leu
705 710 715 720
cac gag cac atc gct aat ctt gca ggt agc cca gct atc aaa aag gga 2208
His Glu His Ile Ala Asn Leu Ala Gly Ser Pro Ala Ile Lys Lys Gly
725 730 735
ata ctg cag acc gtt aag gtc gtg gat gaa ctc gtc aaa gta atg gga 2256
Ile Leu Gln Thr Val Lys Val Val Asp Glu Leu Val Lys Val Met Gly
740 745 750
agg cat aag ccc gag aat atc gtt atc gag atg gcc cga gag aac caa 2304
Arg His Lys Pro Glu Asn Ile Val Ile Glu Met Ala Arg Glu Asn Gln
755 760 765
act acc cag aag gga cag aag aac agt agg gaa agg atg aag agg att 2352
Thr Thr Gln Lys Gly Gln Lys Asn Ser Arg Glu Arg Met Lys Arg Ile
770 775 780
gaa gag ggt ata aaa gaa ctg ggg tcc caa atc ctt aag gaa cac cca 2400
Glu Glu Gly Ile Lys Glu Leu Gly Ser Gln Ile Leu Lys Glu His Pro
785 790 795 800
gtt gaa aac acc cag ctt cag aat gag aag ctc tac ctg tac tac ctg 2448
Val Glu Asn Thr Gln Leu Gln Asn Glu Lys Leu Tyr Leu Tyr Tyr Leu
805 810 815
cag aac ggc agg gac atg tac gtg gat cag gaa ctg gac atc aat cgg 2496
Gln Asn Gly Arg Asp Met Tyr Val Asp Gln Glu Leu Asp Ile Asn Arg
820 825 830
ctc tcc gac tac gac gtg gat cat atc gtg ccc cag tct ttt ctc aaa 2544
Leu Ser Asp Tyr Asp Val Asp His Ile Val Pro Gln Ser Phe Leu Lys
835 840 845
gat gat tct att gat aat aaa gtg ttg aca aga tcc gat aaa aat aga 2592
Asp Asp Ser Ile Asp Asn Lys Val Leu Thr Arg Ser Asp Lys Asn Arg
850 855 860
ggg aag agt gat aac gtc ccc tca gaa gaa gtt gtc aag aaa atg aaa 2640
Gly Lys Ser Asp Asn Val Pro Ser Glu Glu Val Val Lys Lys Met Lys
865 870 875 880
aat tat tgg cgg cag ctg ctg aac gcc aaa ctg atc aca caa cgg aag 2688
Asn Tyr Trp Arg Gln Leu Leu Asn Ala Lys Leu Ile Thr Gln Arg Lys
885 890 895
ttc gat aat ctg act aag gct gaa cga ggt ggc ctg tct gag ttg gat 2736
Phe Asp Asn Leu Thr Lys Ala Glu Arg Gly Gly Leu Ser Glu Leu Asp
900 905 910
aaa gcc ggc ttc atc aaa agg cag ctt gtt gag aca cgc cag atc acc 2784
Lys Ala Gly Phe Ile Lys Arg Gln Leu Val Glu Thr Arg Gln Ile Thr
915 920 925
aag cac gtg gcc caa att ctc gat tca cgc atg aac acc aag tac gat 2832
Lys His Val Ala Gln Ile Leu Asp Ser Arg Met Asn Thr Lys Tyr Asp
930 935 940
gaa aat gac aaa ctg att cga gag gtg aaa gtt att act ctg aag tct 2880
Glu Asn Asp Lys Leu Ile Arg Glu Val Lys Val Ile Thr Leu Lys Ser
945 950 955 960
aag ctg gtc tca gat ttc aga aag gac ttt cag ttt tat aag gtg aga 2928
Lys Leu Val Ser Asp Phe Arg Lys Asp Phe Gln Phe Tyr Lys Val Arg
965 970 975
gag atc aac aat tac cac cat gcg cat gat gcc tac ctg aat gca gtg 2976
Glu Ile Asn Asn Tyr His His Ala His Asp Ala Tyr Leu Asn Ala Val
980 985 990
gta ggc act gca ctt atc aaa aaa tat ccc aag ctt gaa tct gaa ttt 3024
Val Gly Thr Ala Leu Ile Lys Lys Tyr Pro Lys Leu Glu Ser Glu Phe
995 1000 1005
gtt tac gga gac tat aaa gtg tac gat gtt agg aaa atg atc gca 3069
Val Tyr Gly Asp Tyr Lys Val Tyr Asp Val Arg Lys Met Ile Ala
1010 1015 1020
aag tct gag cag gaa ata ggc aag gcc acc gct aag tac ttc ttt 3114
Lys Ser Glu Gln Glu Ile Gly Lys Ala Thr Ala Lys Tyr Phe Phe
1025 1030 1035
tac agc aat att atg aat ttt ttc aag acc gag att aca ctg gcc 3159
Tyr Ser Asn Ile Met Asn Phe Phe Lys Thr Glu Ile Thr Leu Ala
1040 1045 1050
aat gga gag att cgg aag cga cca ctt atc gaa aca aac gga gaa 3204
Asn Gly Glu Ile Arg Lys Arg Pro Leu Ile Glu Thr Asn Gly Glu
1055 1060 1065
aca gga gaa atc gtg tgg gac aag ggt agg gat ttc gcg aca gtc 3249
Thr Gly Glu Ile Val Trp Asp Lys Gly Arg Asp Phe Ala Thr Val
1070 1075 1080
cgg aag gtc ctg tcc atg ccg cag gtg aac atc gtt aaa aag acc 3294
Arg Lys Val Leu Ser Met Pro Gln Val Asn Ile Val Lys Lys Thr
1085 1090 1095
gaa gta cag acc gga ggc ttc tcc aag gaa agt atc ctc ccg aaa 3339
Glu Val Gln Thr Gly Gly Phe Ser Lys Glu Ser Ile Leu Pro Lys
1100 1105 1110
agg aac agc gac aag ctg atc gca cgc aaa aaa gat tgg gac ccc 3384
Arg Asn Ser Asp Lys Leu Ile Ala Arg Lys Lys Asp Trp Asp Pro
1115 1120 1125
aag aaa tac ggc gga ttc gat tct cct aca gtc gct tac agt gta 3429
Lys Lys Tyr Gly Gly Phe Asp Ser Pro Thr Val Ala Tyr Ser Val
1130 1135 1140
ctg gtt gtg gcc aaa gtg gag aaa ggg aag tct aaa aaa ctc aaa 3474
Leu Val Val Ala Lys Val Glu Lys Gly Lys Ser Lys Lys Leu Lys
1145 1150 1155
agc gtc aag gaa ctg ctg ggc atc aca atc atg gag cga tca agc 3519
Ser Val Lys Glu Leu Leu Gly Ile Thr Ile Met Glu Arg Ser Ser
1160 1165 1170
ttc gaa aaa aac ccc atc gac ttt ctc gag gcg aaa gga tat aaa 3564
Phe Glu Lys Asn Pro Ile Asp Phe Leu Glu Ala Lys Gly Tyr Lys
1175 1180 1185
gag gtc aaa aaa gac ctc atc att aag ctt ccc aag tac tct ctc 3609
Glu Val Lys Lys Asp Leu Ile Ile Lys Leu Pro Lys Tyr Ser Leu
1190 1195 1200
ttt gag ctt gaa aac ggc cgg aaa cga atg ctc gct agt gcg ggc 3654
Phe Glu Leu Glu Asn Gly Arg Lys Arg Met Leu Ala Ser Ala Gly
1205 1210 1215
gag ctg cag aaa ggt aac gag ctg gca ctg ccc tct aaa tac gtt 3699
Glu Leu Gln Lys Gly Asn Glu Leu Ala Leu Pro Ser Lys Tyr Val
1220 1225 1230
aat ttc ttg tat ctg gcc agc cac tat gaa aag ctc aaa ggg tct 3744
Asn Phe Leu Tyr Leu Ala Ser His Tyr Glu Lys Leu Lys Gly Ser
1235 1240 1245
ccc gaa gat aat gag cag aag cag ctg ttc gtg gaa caa cac aaa 3789
Pro Glu Asp Asn Glu Gln Lys Gln Leu Phe Val Glu Gln His Lys
1250 1255 1260
cac tac ctt gat gag atc atc gag caa ata agc gaa ttc tcc aaa 3834
His Tyr Leu Asp Glu Ile Ile Glu Gln Ile Ser Glu Phe Ser Lys
1265 1270 1275
aga gtg atc ctc gcc gac gct aac ctc gat aag gtg ctt tct gct 3879
Arg Val Ile Leu Ala Asp Ala Asn Leu Asp Lys Val Leu Ser Ala
1280 1285 1290
tac aat aag cac agg gat aag ccc atc agg gag cag gca gaa aac 3924
Tyr Asn Lys His Arg Asp Lys Pro Ile Arg Glu Gln Ala Glu Asn
1295 1300 1305
att atc cac ttg ttt act ctg acc aac ttg ggc gcg cct gca gcc 3969
Ile Ile His Leu Phe Thr Leu Thr Asn Leu Gly Ala Pro Ala Ala
1310 1315 1320
ttc aag tac ttc gac acc acc ata gac aga aag cgg tac acc tct 4014
Phe Lys Tyr Phe Asp Thr Thr Ile Asp Arg Lys Arg Tyr Thr Ser
1325 1330 1335
aca aag gag gtc ctg gac gcc aca ctg att cat cag tca att acg 4059
Thr Lys Glu Val Leu Asp Ala Thr Leu Ile His Gln Ser Ile Thr
1340 1345 1350
ggg ctc tat gaa aca aga atc gac ctc tct cag ctc ggt gga gac 4104
Gly Leu Tyr Glu Thr Arg Ile Asp Leu Ser Gln Leu Gly Gly Asp
1355 1360 1365
agc agg gct gac 4116
Ser Arg Ala Asp
1370
<210> 6
<211> 1372
<212> PRT
<213> 人工序列
<220>
<223> 合成構建物(Synthetic Construct)
<400> 6
Met Asp Lys Lys Tyr Ser Ile Gly Leu Asp Ile Gly Thr Asn Ser Val
1 5 10 15
Gly Trp Ala Val Ile Thr Asp Glu Tyr Lys Val Pro Ser Lys Lys Phe
20 25 30
Lys Val Leu Gly Asn Thr Asp Arg His Ser Ile Lys Lys Asn Leu Ile
35 40 45
Gly Ala Leu Leu Phe Asp Ser Gly Glu Thr Ala Glu Ala Thr Arg Leu
50 55 60
Lys Arg Thr Ala Arg Arg Arg Tyr Thr Arg Arg Lys Asn Arg Ile Cys
65 70 75 80
Tyr Leu Gln Glu Ile Phe Ser Asn Glu Met Ala Lys Val Asp Asp Ser
85 90 95
Phe Phe His Arg Leu Glu Glu Ser Phe Leu Val Glu Glu Asp Lys Lys
100 105 110
His Glu Arg His Pro Ile Phe Gly Asn Ile Val Asp Glu Val Ala Tyr
115 120 125
His Glu Lys Tyr Pro Thr Ile Tyr His Leu Arg Lys Lys Leu Val Asp
130 135 140
Ser Thr Asp Lys Ala Asp Leu Arg Leu Ile Tyr Leu Ala Leu Ala His
145 150 155 160
Met Ile Lys Phe Arg Gly His Phe Leu Ile Glu Gly Asp Leu Asn Pro
165 170 175
Asp Asn Ser Asp Val Asp Lys Leu Phe Ile Gln Leu Val Gln Thr Tyr
180 185 190
Asn Gln Leu Phe Glu Glu Asn Pro Ile Asn Ala Ser Gly Val Asp Ala
195 200 205
Lys Ala Ile Leu Ser Ala Arg Leu Ser Lys Ser Arg Arg Leu Glu Asn
210 215 220
Leu Ile Ala Gln Leu Pro Gly Glu Lys Lys Asn Gly Leu Phe Gly Asn
225 230 235 240
Leu Ile Ala Leu Ser Leu Gly Leu Thr Pro Asn Phe Lys Ser Asn Phe
245 250 255
Asp Leu Ala Glu Asp Ala Lys Leu Gln Leu Ser Lys Asp Thr Tyr Asp
260 265 270
Asp Asp Leu Asp Asn Leu Leu Ala Gln Ile Gly Asp Gln Tyr Ala Asp
275 280 285
Leu Phe Leu Ala Ala Lys Asn Leu Ser Asp Ala Ile Leu Leu Ser Asp
290 295 300
Ile Leu Arg Val Asn Thr Glu Ile Thr Lys Ala Pro Leu Ser Ala Ser
305 310 315 320
Met Ile Lys Arg Tyr Asp Glu His His Gln Asp Leu Thr Leu Leu Lys
325 330 335
Ala Leu Val Arg Gln Gln Leu Pro Glu Lys Tyr Lys Glu Ile Phe Phe
340 345 350
Asp Gln Ser Lys Asn Gly Tyr Ala Gly Tyr Ile Asp Gly Gly Ala Ser
355 360 365
Gln Glu Glu Phe Tyr Lys Phe Ile Lys Pro Ile Leu Glu Lys Met Asp
370 375 380
Gly Thr Glu Glu Leu Leu Val Lys Leu Asn Arg Glu Asp Leu Leu Arg
385 390 395 400
Lys Gln Arg Thr Phe Asp Asn Gly Ser Ile Pro His Gln Ile His Leu
405 410 415
Gly Glu Leu His Ala Ile Leu Arg Arg Gln Glu Asp Phe Tyr Pro Phe
420 425 430
Leu Lys Asp Asn Arg Glu Lys Ile Glu Lys Ile Leu Thr Phe Arg Ile
435 440 445
Pro Tyr Tyr Val Gly Pro Leu Ala Arg Gly Asn Ser Arg Phe Ala Trp
450 455 460
Met Thr Arg Lys Ser Glu Glu Thr Ile Thr Pro Trp Asn Phe Glu Glu
465 470 475 480
Val Val Asp Lys Gly Ala Ser Ala Gln Ser Phe Ile Glu Arg Met Thr
485 490 495
Asn Phe Asp Lys Asn Leu Pro Asn Glu Lys Val Leu Pro Lys His Ser
500 505 510
Leu Leu Tyr Glu Tyr Phe Thr Val Tyr Asn Glu Leu Thr Lys Val Lys
515 520 525
Tyr Val Thr Glu Gly Met Arg Lys Pro Ala Phe Leu Ser Gly Glu Gln
530 535 540
Lys Lys Ala Ile Val Asp Leu Leu Phe Lys Thr Asn Arg Lys Val Thr
545 550 555 560
Val Lys Gln Leu Lys Glu Asp Tyr Phe Lys Lys Ile Glu Cys Phe Asp
565 570 575
Ser Val Glu Ile Ser Gly Val Glu Asp Arg Phe Asn Ala Ser Leu Gly
580 585 590
Thr Tyr His Asp Leu Leu Lys Ile Ile Lys Asp Lys Asp Phe Leu Asp
595 600 605
Asn Glu Glu Asn Glu Asp Ile Leu Glu Asp Ile Val Leu Thr Leu Thr
610 615 620
Leu Phe Glu Asp Arg Glu Met Ile Glu Glu Arg Leu Lys Thr Tyr Ala
625 630 635 640
His Leu Phe Asp Asp Lys Val Met Lys Gln Leu Lys Arg Arg Arg Tyr
645 650 655
Thr Gly Trp Gly Arg Leu Ser Arg Lys Leu Ile Asn Gly Ile Arg Asp
660 665 670
Lys Gln Ser Gly Lys Thr Ile Leu Asp Phe Leu Lys Ser Asp Gly Phe
675 680 685
Ala Asn Arg Asn Phe Met Gln Leu Ile His Asp Asp Ser Leu Thr Phe
690 695 700
Lys Glu Asp Ile Gln Lys Ala Gln Val Ser Gly Gln Gly Asp Ser Leu
705 710 715 720
His Glu His Ile Ala Asn Leu Ala Gly Ser Pro Ala Ile Lys Lys Gly
725 730 735
Ile Leu Gln Thr Val Lys Val Val Asp Glu Leu Val Lys Val Met Gly
740 745 750
Arg His Lys Pro Glu Asn Ile Val Ile Glu Met Ala Arg Glu Asn Gln
755 760 765
Thr Thr Gln Lys Gly Gln Lys Asn Ser Arg Glu Arg Met Lys Arg Ile
770 775 780
Glu Glu Gly Ile Lys Glu Leu Gly Ser Gln Ile Leu Lys Glu His Pro
785 790 795 800
Val Glu Asn Thr Gln Leu Gln Asn Glu Lys Leu Tyr Leu Tyr Tyr Leu
805 810 815
Gln Asn Gly Arg Asp Met Tyr Val Asp Gln Glu Leu Asp Ile Asn Arg
820 825 830
Leu Ser Asp Tyr Asp Val Asp His Ile Val Pro Gln Ser Phe Leu Lys
835 840 845
Asp Asp Ser Ile Asp Asn Lys Val Leu Thr Arg Ser Asp Lys Asn Arg
850 855 860
Gly Lys Ser Asp Asn Val Pro Ser Glu Glu Val Val Lys Lys Met Lys
865 870 875 880
Asn Tyr Trp Arg Gln Leu Leu Asn Ala Lys Leu Ile Thr Gln Arg Lys
885 890 895
Phe Asp Asn Leu Thr Lys Ala Glu Arg Gly Gly Leu Ser Glu Leu Asp
900 905 910
Lys Ala Gly Phe Ile Lys Arg Gln Leu Val Glu Thr Arg Gln Ile Thr
915 920 925
Lys His Val Ala Gln Ile Leu Asp Ser Arg Met Asn Thr Lys Tyr Asp
930 935 940
Glu Asn Asp Lys Leu Ile Arg Glu Val Lys Val Ile Thr Leu Lys Ser
945 950 955 960
Lys Leu Val Ser Asp Phe Arg Lys Asp Phe Gln Phe Tyr Lys Val Arg
965 970 975
Glu Ile Asn Asn Tyr His His Ala His Asp Ala Tyr Leu Asn Ala Val
980 985 990
Val Gly Thr Ala Leu Ile Lys Lys Tyr Pro Lys Leu Glu Ser Glu Phe
995 1000 1005
Val Tyr Gly Asp Tyr Lys Val Tyr Asp Val Arg Lys Met Ile Ala
1010 1015 1020
Lys Ser Glu Gln Glu Ile Gly Lys Ala Thr Ala Lys Tyr Phe Phe
1025 1030 1035
Tyr Ser Asn Ile Met Asn Phe Phe Lys Thr Glu Ile Thr Leu Ala
1040 1045 1050
Asn Gly Glu Ile Arg Lys Arg Pro Leu Ile Glu Thr Asn Gly Glu
1055 1060 1065
Thr Gly Glu Ile Val Trp Asp Lys Gly Arg Asp Phe Ala Thr Val
1070 1075 1080
Arg Lys Val Leu Ser Met Pro Gln Val Asn Ile Val Lys Lys Thr
1085 1090 1095
Glu Val Gln Thr Gly Gly Phe Ser Lys Glu Ser Ile Leu Pro Lys
1100 1105 1110
Arg Asn Ser Asp Lys Leu Ile Ala Arg Lys Lys Asp Trp Asp Pro
1115 1120 1125
Lys Lys Tyr Gly Gly Phe Asp Ser Pro Thr Val Ala Tyr Ser Val
1130 1135 1140
Leu Val Val Ala Lys Val Glu Lys Gly Lys Ser Lys Lys Leu Lys
1145 1150 1155
Ser Val Lys Glu Leu Leu Gly Ile Thr Ile Met Glu Arg Ser Ser
1160 1165 1170
Phe Glu Lys Asn Pro Ile Asp Phe Leu Glu Ala Lys Gly Tyr Lys
1175 1180 1185
Glu Val Lys Lys Asp Leu Ile Ile Lys Leu Pro Lys Tyr Ser Leu
1190 1195 1200
Phe Glu Leu Glu Asn Gly Arg Lys Arg Met Leu Ala Ser Ala Gly
1205 1210 1215
Glu Leu Gln Lys Gly Asn Glu Leu Ala Leu Pro Ser Lys Tyr Val
1220 1225 1230
Asn Phe Leu Tyr Leu Ala Ser His Tyr Glu Lys Leu Lys Gly Ser
1235 1240 1245
Pro Glu Asp Asn Glu Gln Lys Gln Leu Phe Val Glu Gln His Lys
1250 1255 1260
His Tyr Leu Asp Glu Ile Ile Glu Gln Ile Ser Glu Phe Ser Lys
1265 1270 1275
Arg Val Ile Leu Ala Asp Ala Asn Leu Asp Lys Val Leu Ser Ala
1280 1285 1290
Tyr Asn Lys His Arg Asp Lys Pro Ile Arg Glu Gln Ala Glu Asn
1295 1300 1305
Ile Ile His Leu Phe Thr Leu Thr Asn Leu Gly Ala Pro Ala Ala
1310 1315 1320
Phe Lys Tyr Phe Asp Thr Thr Ile Asp Arg Lys Arg Tyr Thr Ser
1325 1330 1335
Thr Lys Glu Val Leu Asp Ala Thr Leu Ile His Gln Ser Ile Thr
1340 1345 1350
Gly Leu Tyr Glu Thr Arg Ile Asp Leu Ser Gln Leu Gly Gly Asp
1355 1360 1365
Ser Arg Ala Asp
1370
<210> 7
<211> 83
<212> DNA
<213> 化膿性鏈球菌
<400> 7
gttttagagc tagaaatagc aagttaaaat aaggctagtc cgttatcaac ttgaaaaagt 60
ggcaccgagt cggtggtgct ttt 83
<210> 8
<211> 665
<212> DNA
<213> 釀酒酵母(Saccharomyces cerevisiae)
<400> 8
tttcaaaaat tcttactttt tttttggatg gacgcaaaga agtttaataa tcatattaca 60
tggcattacc accatataca tatccatata catatccata tctaatctta cttatatgtt 120
gtggaaatgt aaagagcccc attatcttag cctaaaaaaa ccttctcttt ggaactttca 180
gtaatacgct taactgctca ttgctatatt gaagtacgga ttagaagccg ccgagcgggt 240
gacagccctc cgaaggaaga ctctcctccg tgcgtcctcg tcttcaccgg tcgcgttcct 300
gaaacgcaga tgtgcctcgc gccgcactgc tccgaacaat aaagattcta caatactagc 360
ttttatggtt atgaagagga aaaattggca gtaacctggc cccacaaacc ttcaaatgaa 420
cgaatcaaat taacaaccat aggatgataa tgcgattagt tttttagcct tatttctggg 480
gtaattaatc agcgaagcga tgatttttga tctattaaca gatatataaa tgcaaaaact 540
gcataaccac tttaactaat actttcaaca ttttcggttt gtattacttc ttattcaaat 600
gtaataaaag tatcaacaaa aaattgttaa tatacctcta tactttaacg tcaaggagaa 660
aaaac 665
<210> 9
<211> 21
<212> DNA
<213> 人工序列
<220>
<223> 核躍遷信號(Nuclear transition signal).
<220>
<221> CDS
<222> (1)..(21)
<400> 9
ccc aag aag aag agg aag gtg 21
Pro Lys Lys Lys Arg Lys Val
1 5
<210> 10
<211> 7
<212> PRT
<213> 人工序列
<220>
<223> 合成構建物
<400> 10
Pro Lys Lys Lys Arg Lys Val
1 5
<210> 11
<211> 15
<212> DNA
<213> 人工序列
<220>
<223> GS linker
<220>
<221> CDS
<222> (1)..(15)
<400> 11
ggt gga gga ggt tct 15
Gly Gly Gly Gly Ser
1 5
<210> 12
<211> 5
<212> PRT
<213> 人工序列
<220>
<223> 合成構建物
<400> 12
Gly Gly Gly Gly Ser
1 5
<210> 13
<211> 66
<212> DNA
<213> 人工序列
<220>
<223> Flag tag
<220>
<221> CDS
<222> (1)..(66)
<400> 13
gac tat aag gac cac gac gga gac tac aag gat cat gat att gat tac 48
Asp Tyr Lys Asp His Asp Gly Asp Tyr Lys Asp His Asp Ile Asp Tyr
1 5 10 15
aaa gac gat gac gat aag 66
Lys Asp Asp Asp Asp Lys
20
<210> 14
<211> 22
<212> PRT
<213> 人工序列
<220>
<223> 合成構建物
<400> 14
Asp Tyr Lys Asp His Asp Gly Asp Tyr Lys Asp His Asp Ile Asp Tyr
1 5 10 15
Lys Asp Asp Asp Asp Lys
20
<210> 15
<211> 24
<212> DNA
<213> 人工序列
<220>
<223> Strep-tag
<220>
<221> CDS
<222> (1)..(24)
<400> 15
tgg agc cac ccg cag ttc gaa aaa 24
Trp Ser His Pro Gln Phe Glu Lys
1 5
<210> 16
<211> 8
<212> PRT
<213> 人工序列
<220>
<223> 合成構建物
<400> 16
Trp Ser His Pro Gln Phe Glu Lys
1 5
<210> 17
<211> 171
<212> DNA
<213> 人工序列
<220>
<223> SH3 結構域
<220>
<221> CDS
<222> (1)..(171)
<400> 17
gca gag tat gtg cgg gcc ctc ttt gac ttt aat ggg aat gat gaa gaa 48
Ala Glu Tyr Val Arg Ala Leu Phe Asp Phe Asn Gly Asn Asp Glu Glu
1 5 10 15
gat ctt ccc ttt aag aaa gga gac atc ctg aga atc cgg gat aag cct 96
Asp Leu Pro Phe Lys Lys Gly Asp Ile Leu Arg Ile Arg Asp Lys Pro
20 25 30
gaa gag cag tgg tgg aat gca gag gac agc gaa gga aag agg ggg atg 144
Glu Glu Gln Trp Trp Asn Ala Glu Asp Ser Glu Gly Lys Arg Gly Met
35 40 45
att cct gtc cct tac gtg gag aag tat 171
Ile Pro Val Pro Tyr Val Glu Lys Tyr
50 55
<210> 18
<211> 57
<212> PRT
<213> 人工序列
<220>
<223> 合成構建物
<400> 18
Ala Glu Tyr Val Arg Ala Leu Phe Asp Phe Asn Gly Asn Asp Glu Glu
1 5 10 15
Asp Leu Pro Phe Lys Lys Gly Asp Ile Leu Arg Ile Arg Asp Lys Pro
20 25 30
Glu Glu Gln Trp Trp Asn Ala Glu Asp Ser Glu Gly Lys Arg Gly Met
35 40 45
Ile Pro Val Pro Tyr Val Glu Lys Tyr
50 55
<210> 19
<211> 188
<212> DNA
<213> Saccharomyces cerevisiae
<400> 19
gcgaatttct tatgatttat gatttttatt attaaataag ttataaaaaa aataagtgta 60
tacaaatttt aaagtgactc ttaggtttta aaacgaaaat tcttattctt gagtaactct 120
ttcctgtagg tcaggttgct ttctcaggta tagcatgagg tcgctcttat tgaccacacc 180
tctaccgg 188
<210> 20
<211> 417
<212> DNA
<213> Saccharomyces cerevisiae
<400> 20
ataccaggca tggagcttat ctggtccgtt cgagttttcg acgagtttgg agacattctt 60
tatagatgtc cttttttttt aatgatattc gttaaagaac aaaaagtcaa agcagtttaa 120
cctaacacct gttgttgatg ctacttgaaa caaggcttct aggcgaatac ttaaaaaggt 180
aatttcaata gcggtttata tatctgtttg cttttcaaga tattatgtaa acgcacgatg 240
tttttcgccc aggctttatt ttttttgttg ttgttgtctt ctcgaagaat tttctcgggc 300
agatctttgt cggaatgtaa aaaagcgcgt aattaaactt tctattatgc tgactaaaat 360
ggaagtgatc accaaaggct atttctgatt atataatcta gtcattactc gctcgag 417
<210> 21
<211> 33
<212> DNA
<213> 人工序列
<220>
<223> SH3-結合配體
<220>
<221> CDS
<222> (1)..(33)
<400> 21
cct cca cct gct ctg cca cct aag aga agg aga 33
Pro Pro Pro Ala Leu Pro Pro Lys Arg Arg Arg
1 5 10
<210> 22
<211> 11
<212> PRT
<213> 人工序列
<220>
<223> 合成構建物
<400> 22
Pro Pro Pro Ala Leu Pro Pro Lys Arg Arg Arg
1 5 10
<210> 23
<211> 269
<212> DNA
<213> 釀酒酵母
<400> 23
tctttgaaaa gataatgtat gattatgctt tcactcatat ttatacagaa acttgatgtt 60
ttctttcgag tatatacaag gtgattacat gtacgtttga agtacaactc tagattttgt 120
agtgccctct tgggctagcg gtaaaggtgc gcattttttc acaccctaca atgttctgtt 180
caaaagattt tggtcaaacg ctgtagaagt gaaagttggt gcgcatgttt cggcgttcga 240
aacttctccg cagtgaaaga taaatgatc 269
<210> 24
<211> 14
<212> DNA
<213> 釀酒酵母
<400> 24
tgttttttat gtct 14
<210> 25
<211> 20
<212> DNA
<213> 釀酒酵母
<400> 25
gatacgttct ctatggagga 20
<210> 26
<211> 20
<212> DNA
<213> 釀酒酵母
<400> 26
ttggagaaac ccaggtgcct 20
<210> 27
<211> 20
<212> DNA
<213> 釀酒酵母
<400> 27
aacccaggtg cctggggtcc 20
<210> 28
<211> 20
<212> DNA
<213> 釀酒酵母
<400> 28
ttggccaagt cattcaattt 20
<210> 29
<211> 20
<212> DNA
<213> 釀酒酵母
<400> 29
ataacggaat ccaactgggc 20
<210> 30
<211> 20
<212> DNA
<213> 釀酒酵母
<400> 30
gtcaattacg aagactgaac 20
<210> 31
<211> 19
<212> DNA
<213> 釀酒酵母
<400> 31
gtcaatagga tcccctttt 19
<210> 32
<211> 10126
<212> DNA
<213> 人工序列
<220>
<223> 質粒搭載dCas9-PmCDA1融合蛋白和嵌合RNA
靶向E.coli的galK基因
<220>
<221> misc_feature
<222> (5561)..(5580)
<223> n is a, c, g, or t
<400> 32
atcgccattc gccattcagg ctgcgcaact gttgggaagg gcgatcggtg cgggcctctt 60
cgctattacg ccagctggcg aaagggggat gtgctgcaag gcgattaagt tgggtaacgc 120
cagggttttc ccagtcacga cgttgtaaaa cgacggccag tgaattcgag ctcggtaccc 180
ggccgcaaac aacagataaa acgaaaggcc cagtctttcg actgagcctt tcgttttatt 240
tgatgcctgt caagtaacag caggactctt agtggtgtgg agtattttta cctgaatcat 300
aatggacaac tcgctccgtc gtttttcagc tcgcttcaaa gtcttctcaa gccatctatt 360
ctcattcaat tgattgtgcg acgattggat gaatattttc ctgcaacatt ggtagtgttc 420
acttaccatt acattcaacc caaccccgtt atctctgagg ttccacagcc caatttgatt 480
cctcgcattt ttctcgtaat agagtttgca agcccagatt ttcaaagtgt ggccgttccc 540
ccgcagctcc tggttatacc attctaagat cttttcagcg caatctgcac aaggactcca 600
ggatgagtac caatttatcg tgaattgtcc ggggttgtcg cgcaggtatt cttcgacttt 660
tctaatgcta aagatttcgg cgtgaatgcc acgttctgtc ccgctctgtg gtttattcac 720
agcatagccc caaaaacacg ctctacgttc accccgtcgt tttaattcaa agagaacgta 780
gcatctatgc gacacggatt ttttgttgtt gaaaaactgt ttcttaaacg tgtagatgtc 840
caacttctca tggattctca cgtactcagc gtcggtcatc ctagacttat cgtcatcgtc 900
tttgtaatca atatcatgat ccttgtagtc tccgtcgtgg tccttatagt ctccggactc 960
gagcctagac ttatcgtcat cgtctttgta atcaatatca tgatccttgt agtctccgtc 1020
gtggtcctta tagtctccgg aatacttctc cacgtaaggg acaggaatca tccccctctt 1080
tccttcgctg tcctctgcat tccaccactg ctcctcaggc ttatcccgga ttctcaggat 1140
gtctcctttc ttaaagggaa gatcctcttc atcattccca ttaaagtcaa agagggctcg 1200
cacatactca gcagaacctc cacctccaga acctcctcca ccgtcacctc ctagctgact 1260
caaatcaatg cgtgtttcat aaagaccagt gatggattga tggataagag tggcatctaa 1320
aacttctttt gtagacgtat atcgtttacg atcaattgtt gtatcaaaat atttaaaagc 1380
agcgggagct ccaagattcg tcaacgtaaa taaatgaata atattttctg cttgttcacg 1440
tattggtttg tctctatgtt tgttatatgc actaagaact ttatctaaat tggcatctgc 1500
taaaataaca cgcttagaaa attcactgat ttgctcaata atctcatcta aataatgctt 1560
atgctgctcc acaaacaatt gtttttgttc gttatcttct ggactaccct tcaacttttc 1620
ataatgacta gctaaatata aaaaattcac atatttgctt ggcagagcca gctcatttcc 1680
tttttgtaat tctccggcac tagccagcat ccgtttacga ccgttttcta actcaaaaag 1740
actatattta ggtagtttaa tgattaagtc ttttttaact tccttatatc ctttagcttc 1800
taaaaagtca atcggatttt tttcaaagga acttctttcc ataattgtga tccctagtaa 1860
ctctttaacg gattttaact tcttcgattt ccctttttcc accttagcaa ccactaggac 1920
tgaataagct accgttggac tatcaaaacc accatatttt tttggatccc agtctttttt 1980
acgagcaata agcttgtccg aatttctttt tggtaaaatt gactccttgg agaatccgcc 2040
tgtctgtact tctgttttct tgacaatatt gacttggggc atggacaata ctttgcgcac 2100
tgtggcaaaa tctcgccctt tatcccagac aatttctcca gtttccccat tagtttcgat 2160
tagagggcgt ttgcgaatct ctccatttgc aagtgtaatt tctgttttga agaagttcat 2220
gatattagag taaaagaaat attttgcggt tgctttgcct atttcttgct cagacttagc 2280
aatcatttta cgaacatcat aaactttata atcaccatag acaaactccg attcaagttt 2340
tggatatttc ttaatcaaag cagttccaac gacggcattt agatacgcat catgggcatg 2400
atggtaattg ttaatctcac gtactttata gaattggaaa tcttttcgga agtcagaaac 2460
taatttagat tttaaggtaa tcactttaac ctctcgaata agtttatcat tttcatcgta 2520
tttagtattc atgcgactat ccaaaatttg tgccacatgc ttagtgattt ggcgagtttc 2580
aaccaattgg cgtttgataa aaccagcttt atcaagttca ctcaaacctc cacgttcagc 2640
tttcgttaaa ttatcaaact tacgttgagt gattaacttg gcgtttagaa gttgtctcca 2700
atagtttttc atctttttga ctacttcttc acttggaacg ttatccgatt taccacgatt 2760
tttatcagaa cgcgttaaga ccttattgtc tattgaatcg tctttaagga aactttgtgg 2820
aacaatggca tcgacatcat aatcacttaa acgattaata tctaattctt ggtccacata 2880
catgtctctt ccattttgga gataatagag atagagcttt tcattttgca attgagtatt 2940
ttcaacagga tgctctttaa gaatctgact tcctaattct ttgatacctt cttcgattcg 3000
tttcatacgc tctcgcgaat ttttctggcc cttttgagtt gtctgatttt cacgtgccat 3060
ttcaataacg atattttctg gcttatgccg ccccattact ttgaccaatt catcaacaac 3120
ttttacagtc tgtaaaatac cttttttaat agcagggcta ccagctaaat ttgcaatatg 3180
ttcatgtaaa ctatcgcctt gtccagacac ttgtgctttt tgaatgtctt ctttaaatgt 3240
caaactatca tcatggatca gctgcataaa attgcgattg gcaaaaccat ctgatttcaa 3300
aaaatctaat attgttttgc cagattgctt atccctaata ccattaatca attttcgaga 3360
caaacgtccc caaccagtat aacggcgacg tttaagctgt ttcatcacct tatcatcaaa 3420
gaggtgagca tatgttttaa gtctttcctc aatcatctcc ctatcttcaa ataaggtcaa 3480
tgttaaaaca atatcctcta agatatcttc attttcttca ttatccaaaa aatctttatc 3540
tttaataatt tttagcaaat catggtaggt acctaatgaa gcattaaatc tatcttcaac 3600
tcctgaaatt tcaacactat caaaacattc tatttttttg aaataatctt cttttaattg 3660
cttaacggtt acttttcgat ttgttttgaa gagtaaatca acaatggctt tcttctgttc 3720
acctgaaaga aatgctggtt ttcgcattcc ttcagtaaca tatttgacct ttgtcaattc 3780
gttataaacc gtaaaatact cataaagcaa actatgtttt ggtagtactt tttcatttgg 3840
aagattttta tcaaagtttg tcatgcgttc aataaatgat tgagctgaag cacctttatc 3900
gacaacttct tcaaaattcc atggggtaat tgtttcttca gacttccgag tcatccatgc 3960
aaaacgacta ttgccacgcg ccaatggacc aacataataa ggaattcgaa aagtcaagat 4020
tttttcaatc ttctcacgat tgtcttttaa aaatggataa aagtcttctt gtcttctcaa 4080
aatagcatgc agctcaccca agtgaatttg atggggaata gagccgttgt caaaggtccg 4140
ttgcttgcgc agcaaatctt cacgatttag tttcaccaat aattcctcag taccatccat 4200
tttttctaaa attggtttga taaatttata aaattcttct tggctagctc ccccatcaat 4260
ataacctgca tatccgtttt ttgattgatc aaaaaagatt tctttatact tttctggaag 4320
ttgttgtcga actaaagctt ttaaaagagt caagtcttga tgatgttcat cgtagcgttt 4380
aatcattgaa gctgataggg gagccttagt tatttcagta tttactctta ggatatctga 4440
aagtaaaata gcatctgata aattcttagc tgccaaaaac aaatcagcat attgatctcc 4500
aatttgcgcc aataaattat ctaaatcatc atcgtaagta tcttttgaaa gctgtaattt 4560
agcatcttct gccaaatcaa aatttgattt aaaattaggg gtcaaaccca atgacaaagc 4620
aatgagattc ccaaataagc catttttctt ctcaccgggg agctgagcaa tgagattttc 4680
taatcgtctt gatttactca atcgtgcaga aagaatcgct ttagcatcta ctccacttgc 4740
gttaataggg ttttcttcaa ataattgatt gtaggtttgt accaactgga taaatagttt 4800
gtccacatca ctattatcag gatttaaatc tccctcaatc aaaaaatgac cacgaaactt 4860
aatcatatgc gctaaggcca aatagattaa gcgcaaatcc gctttatcag tagaatctac 4920
caattttttt cgcagatgat agatagttgg atatttctca tgataagcaa cttcatctac 4980
tatatttcca aaaataggat gacgttcatg cttcttgtct tcttccacca aaaaagactc 5040
ttcaagtcga tgaaagaaac tatcatctac tttcgccatc tcatttgaaa aaatctcctg 5100
tagataacaa atacgattct tccgacgtgt ataccttcta cgagctgtcc gtttgagacg 5160
agtcgcttcc gctgtctctc cactgtcaaa taaaagagcc cctataagat tttttttgat 5220
actgtggcgg tctgtatttc ccagaacctt gaacttttta gacggaacct tatattcatc 5280
agtgatcacc gcccatccga cgctatttgt gccgatagct aagcctattg agtatttctt 5340
atccattttt gcctcctaaa atgggccctt taaattaaat ccataatgag tttgatgatt 5400
tcaataatag ttttaatgac ctccgaaatt agtttaatat gctttaattt ttctttttca 5460
aaatatctct tcaaaaaata ttacccaata cttaataata aatagattat aacacaaaat 5520
tcttttgaca agtagtttat tttgttataa ttctatagta nnnnnnnnnn nnnnnnnnnn 5580
gttttagagc tagaaatagc aagttaaaat aaggctagtc cgttatcaac ttgaaaaagt 5640
ggcaccgagt cggtgctttt tttgatactt ctattctact ctgactgcaa accaaaaaaa 5700
caagcgcttt caaaacgctt gttttatcat ttttagggaa attaatctct taatcctttt 5760
atcattctac atttaggcgc tgccatcttg ctaaacctac taagctccac aggatgattt 5820
cgtaatcccg caagaggccc ggcagtaccg gcataaccaa gcctatgcct acagcatcca 5880
gggtgacggt gccgaggatg acgatgagcg cattgttaga tttcatacac ggtgcctgac 5940
tgcgttagca atttaactgt gataaactac cgcattaaag cttatcgatg ataagctgtc 6000
aaacatgaga attacaactt atatcgtatg gggctgactt caggtgctac atttgaagag 6060
ataaattgca ctgaaatcta gtcggatcct cgctcactga ctcgctgcgc tcggtcgttc 6120
ggctgcggcg agcggtatca gctcactcaa aggcggtaat acggttatcc acagaatcag 6180
gggataacgc aggaaagaac atgtgagcaa aaggccagca aaaggccagg aaccgtaaaa 6240
aggccgcgtt gctggcgttt ttccataggc tccgcccccc tgacgagcat cacaaaaatc 6300
gacgctcaag tcagaggtgg cgaaacccga caggactata aagataccag gcgtttcccc 6360
ctggaagctc cctcgtgcgc tctcctgttc cgaccctgcc gcttaccgga tacctgtccg 6420
cctttctccc ttcgggaagc gtggcgcttt ctcatagctc acgctgtagg tatctcagtt 6480
cggtgtaggt cgttcgctcc aagctgggct gtgtgcacga accccccgtt cagcccgacc 6540
gctgcgcctt atccggtaac tatcgtcttg agtccaaccc ggtaagacac gacttatcgc 6600
cactggcagc agccactggt aacaggatta gcagagcgag gtatgtaggc ggtgctacag 6660
agttcttgaa gtggtggcct aactacggct acactagaag gacagtattt ggtatctgcg 6720
ctctgctgaa gccagttacc ttcggaaaaa gagttggtag ctcttgatcc ggcaaacaaa 6780
ccaccgctgg tagcggtggt ttttttgttt gcaagcagca gattacgcgc agaaaaaaag 6840
gatctcaaga agatcctttg atcttttcta cggggtctga cgctcagtgg aacgaaaact 6900
cacgttaagg gattttggtc atgagattat caaaaaggat cttcacctag atccttttaa 6960
attaaaaatg aagttttaaa tcaatctaaa gtatatatga gtaaacttgg tctgacagtt 7020
accaatgctt aatcagtgag gcacctatct cagcgatctg tctatttcgt tcatccatag 7080
ttgcctgact ccccgtcgtg tagataacta cgatacggga gggcttacca tctggcccca 7140
gtgctgcaat gataccgcga gaaccacgct caccggctcc agatttatca gcaataaacc 7200
agccagccgg aagggccgag cgcagaagtg gtcctgcaac tttatccgcc tccatccagt 7260
ctattaattg ttgccgggaa gctagagtaa gtagttcgcc agttaatagt ttgcgcaacg 7320
ttgttgccat tgctgcaggc atcgtggtgt cacgctcgtc gtttggtatg gcttcattca 7380
gctccggttc ccaacgatca aggcgagtta catgatcccc catgttgtgc aaaaaagcgg 7440
ttagctcctt cggtcctccg atcgttgtca gaagtaagtt ggccgcagtg ttatcactca 7500
tggttatggc agcactgcat aattctctta ctgtcatgcc atccgtaaga tgcttttctg 7560
tgactggtga gtactcaacc aagtcattct gagaatagtg tatgcggcga ccgagttgct 7620
cttgcccggc gtcaacacgg gataataccg cgccacatag cagaacttta aaagtgctca 7680
tcattggaaa acgttcttcg gggcgaaaac tctcaaggat cttaccgctg ttgagatcca 7740
gttcgatgta acccactcgt gcacccaact gatcttcagc atcttttact ttcaccagcg 7800
tttctgggtg agcaaaaaca ggaaggcaaa atgccgcaaa aaagggaata agggcgacac 7860
ggaaatgttg aatactcata ctcttccttt ttcaatatta ttgaagcatt tatcagggtt 7920
attgtctcat gagcggatac atatttgaat gtatttagaa aaataaacaa ataggggttc 7980
cgcgcacatt tccccgaaaa gtgccacctg acgtcaatgc cgagcgaaag cgagccgaag 8040
ggtagcattt acgttagata accccctgat atgctccgac gctttatata gaaaagaaga 8100
ttcaactagg taaaatctta atataggttg agatgataag gtttataagg aatttgtttg 8160
ttctaatttt tcactcattt tgttctaatt tcttttaaca aatgttcttt tttttttaga 8220
acagttatga tatagttaga atagtttaaa ataaggagtg agaaaaagat gaaagaaaga 8280
tatggaacag tctataaagg ctctcagagg ctcatagacg aagaaagtgg agaagtcata 8340
gaggtagaca agttataccg taaacaaacg tctggtaact tcgtaaaggc atatatagtg 8400
caattaataa gtatgttaga tatgattggc ggaaaaaaac ttaaaatcgt taactatatc 8460
ctagataatg tccacttaag taacaataca atgatagcta caacaagaga aatagcaaaa 8520
gctacaggaa caagtctaca aacagtaata acaacactta aaatcttaga agaaggaaat 8580
attataaaaa gaaaaactgg agtattaatg ttaaaccctg aactactaat gagaggcgac 8640
gaccaaaaac aaaaatacct cttactcgaa tttgggaact ttgagcaaga ggcaaatgaa 8700
atagattgac ctcccaataa caccacgtag ttattgggag gtcaatctat gaaatgcgat 8760
taagcttttt ctaattcaca taagcgtgca ggtttaaagt acataaaaaa tataatgaaa 8820
aaaagcatca ttatactaac gttataccaa cattatactc tcattatact aattgcttat 8880
tccaatttcc tattggttgg aaccaacagg cgttagtgtg ttgttgagtt ggtactttca 8940
tgggattaat cccatgaaac ccccaaccaa ctcgccaaag ctttggctaa cacacacgcc 9000
attccaacca atagttttct cggcataaag ccatgctctg acgcttaaat gcactaatgc 9060
cttaaaaaaa cattaaagtc taacacacta gacttattta cttcgtaatt aagtcgttaa 9120
accgtgtgct ctacgaccaa aagtataaaa cctttaagaa ctttcttttt tcttgtaaaa 9180
aaagaaacta gataaatctc tcatatcttt tattcaataa tcgcatcaga ttgcagtata 9240
aatttaacga tcactcatca tgttcatatt tatcagagct cgtgctataa ttatactaat 9300
tttataagga ggaaaaaata aagagggtta taatgaacga gaaaaatata aaacacagtc 9360
aaaactttat tacttcaaaa cataatatag ataaaataat gacaaatata agattaaatg 9420
aacatgataa tatctttgaa atcggctcag gaaaagggca ttttaccctt gaattagtac 9480
agaggtgtaa tttcgtaact gccattgaaa tagaccataa attatgcaaa actacagaaa 9540
ataaacttgt tgatcacgat aatttccaag ttttaaacaa ggatatattg cagtttaaat 9600
ttcctaaaaa ccaatcctat aaaatatttg gtaatatacc ttataacata agtacggata 9660
taatacgcaa aattgttttt gatagtatag ctgatgagat ttatttaatc gtggaatacg 9720
ggtttgctaa aagattatta aatacaaaac gctcattggc attattttta atggcagaag 9780
ttgatatttc tatattaagt atggttccaa gagaatattt tcatcctaaa cctaaagtga 9840
atagctcact tatcagatta aatagaaaaa aatcaagaat atcacacaaa gataaacaga 9900
agtataatta tttcgttatg aaatgggtta acaaagaata caagaaaata tttacaaaaa 9960
atcaatttaa caattcctta aaacatgcag gaattgacga tttaaacaat attagctttg 10020
aacaattctt atctcttttc aatagctata aattatttaa taagtaagtt aagggatgca 10080
taaactgcat cccttaactt gtttttcgtg tacctatttt ttgtga 10126
<210> 33
<211> 20
<212> DNA
<213> 大腸桿菌(Escherichia coli)
<400> 33
tcaatgggct aactacgttc 20
<210> 34
<211> 20
<212> DNA
<213> 大腸桿菌
<400> 34
ggtccataaa ctgagacagc 20
<210> 35
<211> 223
<212> DNA
<213> 大腸桿菌
<400> 35
gagttataca cagggctggg atctattctt tttatctttt tttattcttt ctttattcta 60
taaattataa ccacttgaat ataaacaaaa aaaacacaca aaggtctagc ggaatttaca 120
gagggtctag cagaatttac aagttttcca gcaaaggtct agcagaattt acagataccc 180
acaactcaaa ggaaaaggac tagtaattat cattgactag ccc 223
<210> 36
<211> 951
<212> DNA
<213> 大腸桿菌
<400> 36
atgtctgaat tagttgtttt caaagcaaat gaactagcga ttagtcgcta tgacttaacg 60
gagcatgaaa ccaagctaat tttatgctgt gtggcactac tcaaccccac gattgaaaac 120
cctacaatga aagaacggac ggtatcgttc acttataacc aatacgttca gatgatgaac 180
atcagtaggg aaaatgctta tggtgtatta gctaaagcaa ccagagagct gatgacgaga 240
actgtggaaa tcaggaatcc tttggttaaa ggctttgaga ttttccagtg gacaaactat 300
gccaagttct caagcgaaaa attagaatta gtttttagtg aagagatatt gccttatctt 360
ttccagttaa aaaaattcat aaaatataat ctggaacatg ttaagtcttt tgaaaacaaa 420
tactctatga ggatttatga gtggttatta aaagaactaa cacaaaagaa aactcacaag 480
gcaaatatag agattagcct tgatgaattt aagttcatgt taatgcttga aaataactac 540
catgagttta aaaggcttaa ccaatgggtt ttgaaaccaa taagtaaaga tttaaacact 600
tacagcaata tgaaattggt ggttgataag cgaggccgcc cgactgatac gttgattttc 660
caagttgaac tagatagaca aatggatctc gtaaccgaac ttgagaacaa ccagataaaa 720
atgaatggtg acaaaatacc aacaaccatt acatcagatt cctacctaca taacggacta 780
agaaaaacac tacacgatgc tttaactgca aaaattcagc tcaccagttt tgaggcaaaa 840
tttttgagtg acatgcaaag taagcatgat ctcaatggtt cgttctcatg gctcacgcaa 900
aaacaacgaa ccacactaga gaacatactg gctaaatacg gaaggatctg a 951
<210> 37
<211> 714
<212> DNA
<213> λ噬菌體(Bacteriophage lambda)
<400> 37
tcagccaaac gtctcttcag gccactgact agcgataact ttccccacaa cggaacaact 60
ctcattgcat gggatcattg ggtactgtgg gtttagtggt tgtaaaaaca cctgaccgct 120
atccctgatc agtttcttga aggtaaactc atcaccccca agtctggcta tgcagaaatc 180
acctggctca acagcctgct cagggtcaac gagaattaac attccgtcag gaaagcttgg 240
cttggagcct gttggtgcgg tcatggaatt accttcaacc tcaagccaga atgcagaatc 300
actggctttt ttggttgtgc ttacccatct ctccgcatca cctttggtaa aggttctaag 360
cttaggtgag aacatccctg cctgaacatg agaaaaaaca gggtactcat actcacttct 420
aagtgacggc tgcatactaa ccgcttcata catctcgtag atttctctgg cgattgaagg 480
gctaaattct tcaacgctaa ctttgagaat ttttgtaagc aatgcggcgt tataagcatt 540
taatgcattg atgccattaa ataaagcacc aacgcctgac tgccccatcc ccatcttgtc 600
tgcgacagat tcctgggata agccaagttc atttttcttt ttttcataaa ttgctttaag 660
gcgacgtgcg tcctcaagct gctcttgtgt taatggtttc ttttttgtgc tcat 714
<210> 38
<211> 5097
<212> DNA
<213> 人工序列
<220>
<223> dCas9-PmCDA1
<400> 38
atggataaga aatactcaat aggcttagct atcggcacaa atagcgtcgg atgggcggtg 60
atcactgatg aatataaggt tccgtctaaa aagttcaagg ttctgggaaa tacagaccgc 120
cacagtatca aaaaaaatct tataggggct cttttatttg acagtggaga gacagcggaa 180
gcgactcgtc tcaaacggac agctcgtaga aggtatacac gtcggaagaa tcgtatttgt 240
tatctacagg agattttttc aaatgagatg gcgaaagtag atgatagttt ctttcatcga 300
cttgaagagt cttttttggt ggaagaagac aagaagcatg aacgtcatcc tatttttgga 360
aatatagtag atgaagttgc ttatcatgag aaatatccaa ctatctatca tctgcgaaaa 420
aaattggtag attctactga taaagcggat ttgcgcttaa tctatttggc cttagcgcat 480
atgattaagt ttcgtggtca ttttttgatt gagggagatt taaatcctga taatagtgat 540
gtggacaaac tatttatcca gttggtacaa acctacaatc aattatttga agaaaaccct 600
attaacgcaa gtggagtaga tgctaaagcg attctttctg cacgattgag taaatcaaga 660
cgattagaaa atctcattgc tcagctcccc ggtgagaaga aaaatggctt atttgggaat 720
ctcattgctt tgtcattggg tttgacccct aattttaaat caaattttga tttggcagaa 780
gatgctaaat tacagctttc aaaagatact tacgatgatg atttagataa tttattggcg 840
caaattggag atcaatatgc tgatttgttt ttggcagcta agaatttatc agatgctatt 900
ttactttcag atatcctaag agtaaatact gaaataacta aggctcccct atcagcttca 960
atgattaaac gctacgatga acatcatcaa gacttgactc ttttaaaagc tttagttcga 1020
caacaacttc cagaaaagta taaagaaatc ttttttgatc aatcaaaaaa cggatatgca 1080
ggttatattg atgggggagc tagccaagaa gaattttata aatttatcaa accaatttta 1140
gaaaaaatgg atggtactga ggaattattg gtgaaactaa atcgtgaaga tttgctgcgc 1200
aagcaacgga cctttgacaa cggctctatt ccccatcaaa ttcacttggg tgagctgcat 1260
gctattttga gaagacaaga agacttttat ccatttttaa aagacaatcg tgagaagatt 1320
gaaaaaatct tgacttttcg aattccttat tatgttggtc cattggcgcg tggcaatagt 1380
cgttttgcat ggatgactcg gaagtctgaa gaaacaatta ccccatggaa ttttgaagaa 1440
gttgtcgata aaggtgcttc agctcaatca tttattgaac gcatgacaaa ctttgataaa 1500
aatcttccaa atgaaaaagt actaccaaaa catagtttgc tttatgagta ttttacggtt 1560
tataacgaat tgacaaaggt caaatatgtt actgaaggaa tgcgaaaacc agcatttctt 1620
tcaggtgaac agaagaaagc cattgttgat ttactcttca aaacaaatcg aaaagtaacc 1680
gttaagcaat taaaagaaga ttatttcaaa aaaatagaat gttttgatag tgttgaaatt 1740
tcaggagttg aagatagatt taatgcttca ttaggtacct accatgattt gctaaaaatt 1800
attaaagata aagatttttt ggataatgaa gaaaatgaag atatcttaga ggatattgtt 1860
ttaacattga ccttatttga agatagggag atgattgagg aaagacttaa aacatatgct 1920
cacctctttg atgataaggt gatgaaacag cttaaacgtc gccgttatac tggttgggga 1980
cgtttgtctc gaaaattgat taatggtatt agggataagc aatctggcaa aacaatatta 2040
gattttttga aatcagatgg ttttgccaat cgcaatttta tgcagctgat ccatgatgat 2100
agtttgacat ttaaagaaga cattcaaaaa gcacaagtgt ctggacaagg cgatagttta 2160
catgaacata ttgcaaattt agctggtagc cctgctatta aaaaaggtat tttacagact 2220
gtaaaagttg ttgatgaatt ggtcaaagta atggggcggc ataagccaga aaatatcgtt 2280
attgaaatgg cacgtgaaaa tcagacaact caaaagggcc agaaaaattc gcgagagcgt 2340
atgaaacgaa tcgaagaagg tatcaaagaa ttaggaagtc agattcttaa agagcatcct 2400
gttgaaaata ctcaattgca aaatgaaaag ctctatctct attatctcca aaatggaaga 2460
gacatgtatg tggaccaaga attagatatt aatcgtttaa gtgattatga tgtcgatgcc 2520
attgttccac aaagtttcct taaagacgat tcaatagaca ataaggtctt aacgcgttct 2580
gataaaaatc gtggtaaatc ggataacgtt ccaagtgaag aagtagtcaa aaagatgaaa 2640
aactattgga gacaacttct aaacgccaag ttaatcactc aacgtaagtt tgataattta 2700
acgaaagctg aacgtggagg tttgagtgaa cttgataaag ctggttttat caaacgccaa 2760
ttggttgaaa ctcgccaaat cactaagcat gtggcacaaa ttttggatag tcgcatgaat 2820
actaaatacg atgaaaatga taaacttatt cgagaggtta aagtgattac cttaaaatct 2880
aaattagttt ctgacttccg aaaagatttc caattctata aagtacgtga gattaacaat 2940
taccatcatg cccatgatgc gtatctaaat gccgtcgttg gaactgcttt gattaagaaa 3000
tatccaaaac ttgaatcgga gtttgtctat ggtgattata aagtttatga tgttcgtaaa 3060
atgattgcta agtctgagca agaaataggc aaagcaaccg caaaatattt cttttactct 3120
aatatcatga acttcttcaa aacagaaatt acacttgcaa atggagagat tcgcaaacgc 3180
cctctaatcg aaactaatgg ggaaactgga gaaattgtct gggataaagg gcgagatttt 3240
gccacagtgc gcaaagtatt gtccatgccc caagtcaata ttgtcaagaa aacagaagta 3300
cagacaggcg gattctccaa ggagtcaatt ttaccaaaaa gaaattcgga caagcttatt 3360
gctcgtaaaa aagactggga tccaaaaaaa tatggtggtt ttgatagtcc aacggtagct 3420
tattcagtcc tagtggttgc taaggtggaa aaagggaaat cgaagaagtt aaaatccgtt 3480
aaagagttac tagggatcac aattatggaa agaagttcct ttgaaaaaaa tccgattgac 3540
tttttagaag ctaaaggata taaggaagtt aaaaaagact taatcattaa actacctaaa 3600
tatagtcttt ttgagttaga aaacggtcgt aaacggatgc tggctagtgc cggagaatta 3660
caaaaaggaa atgagctggc tctgccaagc aaatatgtga attttttata tttagctagt 3720
cattatgaaa agttgaaggg tagtccagaa gataacgaac aaaaacaatt gtttgtggag 3780
cagcataagc attatttaga tgagattatt gagcaaatca gtgaattttc taagcgtgtt 3840
attttagcag atgccaattt agataaagtt cttagtgcat ataacaaaca tagagacaaa 3900
ccaatacgtg aacaagcaga aaatattatt catttattta cgttgacgaa tcttggagct 3960
cccgctgctt ttaaatattt tgatacaaca attgatcgta aacgatatac gtctacaaaa 4020
gaagttttag atgccactct tatccatcaa tccatcactg gtctttatga aacacgcatt 4080
gatttgagtc agctaggagg tgacggtgga ggaggttctg gaggtggagg ttctgctgag 4140
tatgtgcgag ccctctttga ctttaatggg aatgatgaag aggatcttcc ctttaagaaa 4200
ggagacatcc tgagaatccg ggataagcct gaggagcagt ggtggaatgc agaggacagc 4260
gaaggaaaga gggggatgat tcctgtccct tacgtggaga agtattccgg agactataag 4320
gaccacgacg gagactacaa ggatcatgat attgattaca aagacgatga cgataagtct 4380
aggctcgagt ccggagacta taaggaccac gacggagact acaaggatca tgatattgat 4440
tacaaagacg atgacgataa gtctaggatg accgacgctg agtacgtgag aatccatgag 4500
aagttggaca tctacacgtt taagaaacag tttttcaaca acaaaaaatc cgtgtcgcat 4560
agatgctacg ttctctttga attaaaacga cggggtgaac gtagagcgtg tttttggggc 4620
tatgctgtga ataaaccaca gagcgggaca gaacgtggca ttcacgccga aatctttagc 4680
attagaaaag tcgaagaata cctgcgcgac aaccccggac aattcacgat aaattggtac 4740
tcatcctgga gtccttgtgc agattgcgct gaaaagatct tagaatggta taaccaggag 4800
ctgcggggga acggccacac tttgaaaatc tgggcttgca aactctatta cgagaaaaat 4860
gcgaggaatc aaattgggct gtggaacctc agagataacg gggttgggtt gaatgtaatg 4920
gtaagtgaac actaccaatg ttgcaggaaa atattcatcc aatcgtcgca caatcaattg 4980
aatgagaata gatggcttga gaagactttg aagcgagctg aaaaacgacg gagcgagttg 5040
tccattatga ttcaggtaaa aatactccac accactaaga gtcctgctgt tacttga 5097
<210> 39
<211> 105
<212> DNA
<213> 大腸桿菌
<400> 39
acgttaaatc tatcaccgca agggataaat atctaacacc gtgcgtgttg actattttac 60
ctctggcggt gataatggtt gcagggccca ttttaggagg caaaa 105
<210> 40
<211> 247
<212> DNA
<213> 人工序列
<220>
<223> gRNA
<400> 40
ggtttagcaa gatggcagcg cctaaatgta gaatgataaa aggattaaga gattaatttc 60
cctaaaaatg ataaaacaag cgttttgaaa gcgcttgttt ttttggtttg cagtcagagt 120
agaatagaag tatcaaaaaa agcaccgact cggtgccact ttttcaagtt gataacggac 180
tagccttatt ttaacttgct atttctagct ctaaaactga gaccatcccg ggtctctact 240
gcagaat 247
<210> 41
<211> 64
<212> DNA
<213> 大腸桿菌
<400> 41
tatcaccgcc agtggtattt atgtcaacac cgccagagat aatttatcac cgcagatggt 60
tatc 64
<210> 42
<211> 10867
<212> DNA
<213> 人工序列
<220>
<223> 質粒
<400> 42
gtcggaactg actaaagtag tgagttatac acagggctgg gatctattct ttttatcttt 60
ttttattctt tctttattct ataaattata accacttgaa tataaacaaa aaaaacacac 120
aaaggtctag cggaatttac agagggtcta gcagaattta caagttttcc agcaaaggtc 180
tagcagaatt tacagatacc cacaactcaa aggaaaagga ctagtaatta tcattgacta 240
gcccatctca attggtatag tgattaaaat cacctagacc aattgagatg tatgtctgaa 300
ttagttgttt tcaaagcaaa tgaactagcg attagtcgct atgacttaac ggagcatgaa 360
accaagctaa ttttatgctg tgtggcacta ctcaacccca cgattgaaaa ccctacaatg 420
aaagaacgga cggtatcgtt cacttataac caatacgttc agatgatgaa catcagtagg 480
gaaaatgctt atggtgtatt agctaaagca accagagagc tgatgacgag aactgtggaa 540
atcaggaatc ctttggttaa aggctttgag attttccagt ggacaaacta tgccaagttc 600
tcaagcgaaa aattagaatt agtttttagt gaagagatat tgccttatct tttccagtta 660
aaaaaattca taaaatataa tctggaacat gttaagtctt ttgaaaacaa atactctatg 720
aggatttatg agtggttatt aaaagaacta acacaaaaga aaactcacaa ggcaaatata 780
gagattagcc ttgatgaatt taagttcatg ttaatgcttg aaaataacta ccatgagttt 840
aaaaggctta accaatgggt tttgaaacca ataagtaaag atttaaacac ttacagcaat 900
atgaaattgg tggttgataa gcgaggccgc ccgactgata cgttgatttt ccaagttgaa 960
ctagatagac aaatggatct cgtaaccgaa cttgagaaca accagataaa aatgaatggt 1020
gacaaaatac caacaaccat tacatcagat tcctacctac ataacggact aagaaaaaca 1080
ctacacgatg ctttaactgc aaaaattcag ctcaccagtt ttgaggcaaa atttttgagt 1140
gacatgcaaa gtaagcatga tctcaatggt tcgttctcat ggctcacgca aaaacaacga 1200
accacactag agaacatact ggctaaatac ggaaggatct gaggttctta tggctcttgt 1260
atctatcagt gaagcatcaa gactaacaaa caaaagtaga acaactgttc accgttacat 1320
atcaaaggga aaactgtcca tatgcacaga gataatctca tgaccaaaac cggtagctag 1380
aggggccgca ttaggcaccc caggctttac actttatgct tccggctcgt ataatgtgtg 1440
gattttgagt taggatccgg cgagattttc aggagctaag gaagctaaaa tggagaaaaa 1500
aatcactgga tataccaccg ttgatatatc ccaatggcat cgtaaagaac attttgaggc 1560
atttcagtca gttgctcaat gtacctataa ccagaccgtt cagctggata ttacggcctt 1620
tttaaagacc gtaaagaaaa ataagcacaa gttttatccg gcctttattc acattcttgc 1680
ccgcctgatg aatgctcatc cggaattccg tatggcaatg aaagacggtg agctggtgat 1740
atgggatagt gttcaccctt gttacaccgt tttccatgag caaactgaaa cgttttcatc 1800
gctctggagt gaataccacg acgatttccg gcagtttcta cacatatatt cgcaagatgt 1860
ggcgtgttac ggtgaaaacc tggcctattt ccctaaaggg tttattgaga atatgttttt 1920
cgtctcagcc aatccctggg tgagtttcac cagttttgat ttaaacgtgg ccaatatgga 1980
caacttcttc gcccccgttt tcaccatggg caaatattat acgcaaggcg acaaggtgct 2040
gatgccgctg gcgattcagg ttcatcatgc cgtctgtgat ggcttccatg tcggcagaat 2100
gcttaatgaa ttacaacagt actgcgatga gtggcagggc ggggcgtaaa cgcgtggatc 2160
cggcttacta aaagccagat aacagtatgc gtatttgcgc gctgattttt gcggtctaga 2220
ggtttagcaa gatggcagcg cctaaatgta gaatgataaa aggattaaga gattaatttc 2280
cctaaaaatg ataaaacaag cgttttgaaa gcgcttgttt ttttggtttg cagtcagagt 2340
agaatagaag tatcaaaaaa agcaccgact cggtgccact ttttcaagtt gataacggac 2400
tagccttatt ttaacttgct atttctagct ctaaaactga gaccatcccg ggtctctact 2460
gcagaattat caccgccagt ggtatttatg tcaacaccgc cagagataat ttatcaccgc 2520
agatggttat cgatgaagat tcttgctcaa ttgttatcag ctatgcgccg accagaacac 2580
cttgccgatc agccaaacgt ctcttcaggc cactgactag cgataacttt ccccacaacg 2640
gaacaactct cattgcatgg gatcattggg tactgtgggt ttagtggttg taaaaacacc 2700
tgaccgctat ccctgatcag tttcttgaag gtaaactcat cacccccaag tctggctatg 2760
cagaaatcac ctggctcaac agcctgctca gggtcaacga gaattaacat tccgtcagga 2820
aagcttggct tggagcctgt tggtgcggtc atggaattac cttcaacctc aagccagaat 2880
gcagaatcac tggctttttt ggttgtgctt acccatctct ccgcatcacc tttggtaaag 2940
gttctaagct taggtgagaa catccctgcc tgaacatgag aaaaaacagg gtactcatac 3000
tcacttctaa gtgacggctg catactaacc gcttcataca tctcgtagat ttctctggcg 3060
attgaagggc taaattcttc aacgctaact ttgagaattt ttgtaagcaa tgcggcgtta 3120
taagcattta atgcattgat gccattaaat aaagcaccaa cgcctgactg ccccatcccc 3180
atcttgtctg cgacagattc ctgggataag ccaagttcat ttttcttttt ttcataaatt 3240
gctttaaggc gacgtgcgtc ctcaagctgc tcttgtgtta atggtttctt ttttgtgctc 3300
atacgttaaa tctatcaccg caagggataa atatctaaca ccgtgcgtgt tgactatttt 3360
acctctggcg gtgataatgg ttgcagggcc cattttagga ggcaaaaatg gataagaaat 3420
actcaatagg cttagctatc ggcacaaata gcgtcggatg ggcggtgatc actgatgaat 3480
ataaggttcc gtctaaaaag ttcaaggttc tgggaaatac agaccgccac agtatcaaaa 3540
aaaatcttat aggggctctt ttatttgaca gtggagagac agcggaagcg actcgtctca 3600
aacggacagc tcgtagaagg tatacacgtc ggaagaatcg tatttgttat ctacaggaga 3660
ttttttcaaa tgagatggcg aaagtagatg atagtttctt tcatcgactt gaagagtctt 3720
ttttggtgga agaagacaag aagcatgaac gtcatcctat ttttggaaat atagtagatg 3780
aagttgctta tcatgagaaa tatccaacta tctatcatct gcgaaaaaaa ttggtagatt 3840
ctactgataa agcggatttg cgcttaatct atttggcctt agcgcatatg attaagtttc 3900
gtggtcattt tttgattgag ggagatttaa atcctgataa tagtgatgtg gacaaactat 3960
ttatccagtt ggtacaaacc tacaatcaat tatttgaaga aaaccctatt aacgcaagtg 4020
gagtagatgc taaagcgatt ctttctgcac gattgagtaa atcaagacga ttagaaaatc 4080
tcattgctca gctccccggt gagaagaaaa atggcttatt tgggaatctc attgctttgt 4140
cattgggttt gacccctaat tttaaatcaa attttgattt ggcagaagat gctaaattac 4200
agctttcaaa agatacttac gatgatgatt tagataattt attggcgcaa attggagatc 4260
aatatgctga tttgtttttg gcagctaaga atttatcaga tgctatttta ctttcagata 4320
tcctaagagt aaatactgaa ataactaagg ctcccctatc agcttcaatg attaaacgct 4380
acgatgaaca tcatcaagac ttgactcttt taaaagcttt agttcgacaa caacttccag 4440
aaaagtataa agaaatcttt tttgatcaat caaaaaacgg atatgcaggt tatattgatg 4500
ggggagctag ccaagaagaa ttttataaat ttatcaaacc aattttagaa aaaatggatg 4560
gtactgagga attattggtg aaactaaatc gtgaagattt gctgcgcaag caacggacct 4620
ttgacaacgg ctctattccc catcaaattc acttgggtga gctgcatgct attttgagaa 4680
gacaagaaga cttttatcca tttttaaaag acaatcgtga gaagattgaa aaaatcttga 4740
cttttcgaat tccttattat gttggtccat tggcgcgtgg caatagtcgt tttgcatgga 4800
tgactcggaa gtctgaagaa acaattaccc catggaattt tgaagaagtt gtcgataaag 4860
gtgcttcagc tcaatcattt attgaacgca tgacaaactt tgataaaaat cttccaaatg 4920
aaaaagtact accaaaacat agtttgcttt atgagtattt tacggtttat aacgaattga 4980
caaaggtcaa atatgttact gaaggaatgc gaaaaccagc atttctttca ggtgaacaga 5040
agaaagccat tgttgattta ctcttcaaaa caaatcgaaa agtaaccgtt aagcaattaa 5100
aagaagatta tttcaaaaaa atagaatgtt ttgatagtgt tgaaatttca ggagttgaag 5160
atagatttaa tgcttcatta ggtacctacc atgatttgct aaaaattatt aaagataaag 5220
attttttgga taatgaagaa aatgaagata tcttagagga tattgtttta acattgacct 5280
tatttgaaga tagggagatg attgaggaaa gacttaaaac atatgctcac ctctttgatg 5340
ataaggtgat gaaacagctt aaacgtcgcc gttatactgg ttggggacgt ttgtctcgaa 5400
aattgattaa tggtattagg gataagcaat ctggcaaaac aatattagat tttttgaaat 5460
cagatggttt tgccaatcgc aattttatgc agctgatcca tgatgatagt ttgacattta 5520
aagaagacat tcaaaaagca caagtgtctg gacaaggcga tagtttacat gaacatattg 5580
caaatttagc tggtagccct gctattaaaa aaggtatttt acagactgta aaagttgttg 5640
atgaattggt caaagtaatg gggcggcata agccagaaaa tatcgttatt gaaatggcac 5700
gtgaaaatca gacaactcaa aagggccaga aaaattcgcg agagcgtatg aaacgaatcg 5760
aagaaggtat caaagaatta ggaagtcaga ttcttaaaga gcatcctgtt gaaaatactc 5820
aattgcaaaa tgaaaagctc tatctctatt atctccaaaa tggaagagac atgtatgtgg 5880
accaagaatt agatattaat cgtttaagtg attatgatgt cgatgccatt gttccacaaa 5940
gtttccttaa agacgattca atagacaata aggtcttaac gcgttctgat aaaaatcgtg 6000
gtaaatcgga taacgttcca agtgaagaag tagtcaaaaa gatgaaaaac tattggagac 6060
aacttctaaa cgccaagtta atcactcaac gtaagtttga taatttaacg aaagctgaac 6120
gtggaggttt gagtgaactt gataaagctg gttttatcaa acgccaattg gttgaaactc 6180
gccaaatcac taagcatgtg gcacaaattt tggatagtcg catgaatact aaatacgatg 6240
aaaatgataa acttattcga gaggttaaag tgattacctt aaaatctaaa ttagtttctg 6300
acttccgaaa agatttccaa ttctataaag tacgtgagat taacaattac catcatgccc 6360
atgatgcgta tctaaatgcc gtcgttggaa ctgctttgat taagaaatat ccaaaacttg 6420
aatcggagtt tgtctatggt gattataaag tttatgatgt tcgtaaaatg attgctaagt 6480
ctgagcaaga aataggcaaa gcaaccgcaa aatatttctt ttactctaat atcatgaact 6540
tcttcaaaac agaaattaca cttgcaaatg gagagattcg caaacgccct ctaatcgaaa 6600
ctaatgggga aactggagaa attgtctggg ataaagggcg agattttgcc acagtgcgca 6660
aagtattgtc catgccccaa gtcaatattg tcaagaaaac agaagtacag acaggcggat 6720
tctccaagga gtcaatttta ccaaaaagaa attcggacaa gcttattgct cgtaaaaaag 6780
actgggatcc aaaaaaatat ggtggttttg atagtccaac ggtagcttat tcagtcctag 6840
tggttgctaa ggtggaaaaa gggaaatcga agaagttaaa atccgttaaa gagttactag 6900
ggatcacaat tatggaaaga agttcctttg aaaaaaatcc gattgacttt ttagaagcta 6960
aaggatataa ggaagttaaa aaagacttaa tcattaaact acctaaatat agtctttttg 7020
agttagaaaa cggtcgtaaa cggatgctgg ctagtgccgg agaattacaa aaaggaaatg 7080
agctggctct gccaagcaaa tatgtgaatt ttttatattt agctagtcat tatgaaaagt 7140
tgaagggtag tccagaagat aacgaacaaa aacaattgtt tgtggagcag cataagcatt 7200
atttagatga gattattgag caaatcagtg aattttctaa gcgtgttatt ttagcagatg 7260
ccaatttaga taaagttctt agtgcatata acaaacatag agacaaacca atacgtgaac 7320
aagcagaaaa tattattcat ttatttacgt tgacgaatct tggagctccc gctgctttta 7380
aatattttga tacaacaatt gatcgtaaac gatatacgtc tacaaaagaa gttttagatg 7440
ccactcttat ccatcaatcc atcactggtc tttatgaaac acgcattgat ttgagtcagc 7500
taggaggtga cggtggagga ggttctggag gtggaggttc tgctgagtat gtgcgagccc 7560
tctttgactt taatgggaat gatgaagagg atcttccctt taagaaagga gacatcctga 7620
gaatccggga taagcctgag gagcagtggt ggaatgcaga ggacagcgaa ggaaagaggg 7680
ggatgattcc tgtcccttac gtggagaagt attccggaga ctataaggac cacgacggag 7740
actacaagga tcatgatatt gattacaaag acgatgacga taagtctagg ctcgagtccg 7800
gagactataa ggaccacgac ggagactaca aggatcatga tattgattac aaagacgatg 7860
acgataagtc taggatgacc gacgctgagt acgtgagaat ccatgagaag ttggacatct 7920
acacgtttaa gaaacagttt ttcaacaaca aaaaatccgt gtcgcataga tgctacgttc 7980
tctttgaatt aaaacgacgg ggtgaacgta gagcgtgttt ttggggctat gctgtgaata 8040
aaccacagag cgggacagaa cgtggcattc acgccgaaat ctttagcatt agaaaagtcg 8100
aagaatacct gcgcgacaac cccggacaat tcacgataaa ttggtactca tcctggagtc 8160
cttgtgcaga ttgcgctgaa aagatcttag aatggtataa ccaggagctg cgggggaacg 8220
gccacacttt gaaaatctgg gcttgcaaac tctattacga gaaaaatgcg aggaatcaaa 8280
ttgggctgtg gaacctcaga gataacgggg ttgggttgaa tgtaatggta agtgaacact 8340
accaatgttg caggaaaata ttcatccaat cgtcgcacaa tcaattgaat gagaatagat 8400
ggcttgagaa gactttgaag cgagctgaaa aacgacggag cgagttgtcc attatgattc 8460
aggtaaaaat actccacacc actaagagtc ctgctgttac ttgacaggca tcaaataaaa 8520
cgaaaggctc agtcgaaaga ctgggccttt cgttttatct gttgtttgcg gccgggtacc 8580
gagctcgaat tcactggccg tcgttttaca acgtcgtgac tgggaaaacc ctggcgttac 8640
ccaacttaat cgccttgcag cacatccccc tttcgccagc tggcgtaata gcgaagaggc 8700
ccgcaccgat cgcccttccc aacagttgcg cagcctgaat ggcgaatggc gattcacaaa 8760
aaataggtac acgaaaaaca agttaaggga tgcagtttat gcatccctta acttacttat 8820
taaataattt atagctattg aaaagagata agaattgttc aaagctaata ttgtttaaat 8880
cgtcaattcc tgcatgtttt aaggaattgt taaattgatt ttttgtaaat attttcttgt 8940
attctttgtt aacccatttc ataacgaaat aattatactt ctgtttatct ttgtgtgata 9000
ttcttgattt ttttctattt aatctgataa gtgagctatt cactttaggt ttaggatgaa 9060
aatattctct tggaaccata cttaatatag aaatatcaac ttctgccatt aaaaataatg 9120
ccaatgagcg ttttgtattt aataatcttt tagcaaaccc gtattccacg attaaataaa 9180
tctcatcagc tatactatca aaaacaattt tgcgtattat atccgtactt atgttataag 9240
gtatattacc aaatatttta taggattggt ttttaggaaa tttaaactgc aatatatcct 9300
tgtttaaaac ttggaaatta tcgtgatcaa caagtttatt ttctgtagtt ttgcataatt 9360
tatggtctat ttcaatggca gttacgaaat tacacctctg tactaattca agggtaaaat 9420
gcccttttcc tgagccgatt tcaaagatat tatcatgttc atttaatctt atatttgtca 9480
ttattttatc tatattatgt tttgaagtaa taaagttttg actgtgtttt atatttttct 9540
cgttcattat aaccctcttt attttttcct ccttataaaa ttagtataat tatagcacga 9600
gctctgataa atatgaacat gatgagtgat cgttaaattt atactgcaat ctgatgcgat 9660
tattgaataa aagatatgag agatttatct agtttctttt tttacaagaa aaaagaaagt 9720
tcttaaaggt tttatacttt tggtcgtaga gcacacggtt taacgactta attacgaagt 9780
aaataagtct agtgtgttag actttaatgt ttttttaagg cattagtgca tttaagcgtc 9840
agagcatggc tttatgccga gaaaactatt ggttggaatg gcgtgtgtgt tagccaaagc 9900
tttggcgagt tggttggggg tttcatggga ttaatcccat gaaagtacca actcaacaac 9960
acactaacgc ctgttggttc caaccaatag gaaattggaa taagcaatta gtataatgag 10020
agtataatgt tggtataacg ttagtataat gatgcttttt ttcattatat tttttatgta 10080
ctttaaacct gcacgcttat gtgaattaga aaaagcttaa tcgcatttca tagattgacc 10140
tcccaataac tacgtggtgt tattgggagg tcaatctatt tcatttgcct cttgctcaaa 10200
gttcccaaat tcgagtaaga ggtatttttg tttttggtcg tcgcctctca ttagtagttc 10260
agggtttaac attaatactc cagtttttct ttttataata tttccttctt ctaagatttt 10320
aagtgttgtt attactgttt gtagacttgt tcctgtagct tttgctattt ctcttgttgt 10380
agctatcatt gtattgttac ttaagtggac attatctagg atatagttaa cgattttaag 10440
tttttttccg ccaatcatat ctaacatact tattaattgc actatatatg cctttacgaa 10500
gttaccagac gtttgtttac ggtataactt gtctacctct atgacttctc cactttcttc 10560
gtctatgagc ctctgagagc ctttatagac tgttccatat ctttctttca tctttttctc 10620
actccttatt ttaaactatt ctaactatat cataactgtt ctaaaaaaaa aagaacattt 10680
gttaaaagaa attagaacaa aatgagtgaa aaattagaac aaacaaattc cttataaacc 10740
ttatcatctc aacctatatt aagattttac ctagttgaat cttcttttct atataaagcg 10800
tcggagcata tcagggggtt atctaacgta aatgctaccc ttcggctcgc tttcgctcgg 10860
cattgac 10867
- 還沒有人留言評論。精彩留言會獲得點贊!